8+结合10种机器学习算法构建模型,可复现。

作为最具侵袭性的肿瘤,胰腺癌(PACA)的预后在过去十年中没有明显改善。基于解剖学的TNM分期不能准确识别对治疗敏感的患者,精准医学迫切需要一种理想的生物标志物。
1. 胰腺癌症共识基因特征的综合开发
作者的工作流程如图1所示。基于单变量Cox回归,作者从训练和9个测试队列中的15288个交集基因中筛选了32个CPG(图2B)。接下来,这32个CPG被进一步纳入作者的集成计划,以开发AIDPS。在PACA AU阵列训练队列中,作者通过十倍交叉验证应用了76种算法组合来构建预测模型,并计算了其余9个测试队列中每种算法的平均C指数。如图2A所示,选择平均C指数最高(0.675)的CoxBoost和生存SVM组合作为最终模型。根据AIDPS中包含的9个基因的表达文件,作者进一步计算了所有13个队列中每个样本的AIDPS得分(图2)。

图1 研究的工作流程

图2 人工智能衍生的预后标志(AIDPS)的构建和测试
2. AIDPS的一致预后价值
为了评估AIDPS的预后表现,作者根据中位数将PACA患者分为AIDPS高组和AIDPS低组。OS和RFS的Kaplan–Meier曲线表明,在PACA AU阵列训练队列中,高AIDPS组具有显著更长的生存期(OS中p<0.0001,RFS中p=0.012,图3A和B)。在去除批量效应后,结合10个队列(训练和9个测试队列)的Meta队列也表现出相同的趋势(均p<0.05,图3C和D)。此外,作者进一步纳入了几个重要的临床特征进行多变量Cox分析,结果表明,在PACA AU阵列队列中,AIDPS是OS和RFS的独立保护因素(OS的HR:0.593[0.504–0.697],RFS的HR=0.762[0.611–0.949],均p<0.05,图3E和F)。Meta队列中也发现了类似的结果(OS的HR:0.603[0.531–0.685]和RFS的HR:0.667[0.552–0.805],均p<0.05,图3G和H)。

图3 人工智能衍生预后标志(AIDPS)的生存分析和预测性能评估

补充图3-1 九个测试队列中人工智能衍生预后标志(AIDPS)的生存分析
补充图3-2 九个测试队列中人工智能衍生预后标志(AIDPS)的生存分析

补充图3-3 人工智能衍生预后特征(AIDPS)在九个测试队列中的预测性能
补充图3-4 人工智能衍生预后标志(AIDPS)在三个外部验证队列中的生存分析和预测性能
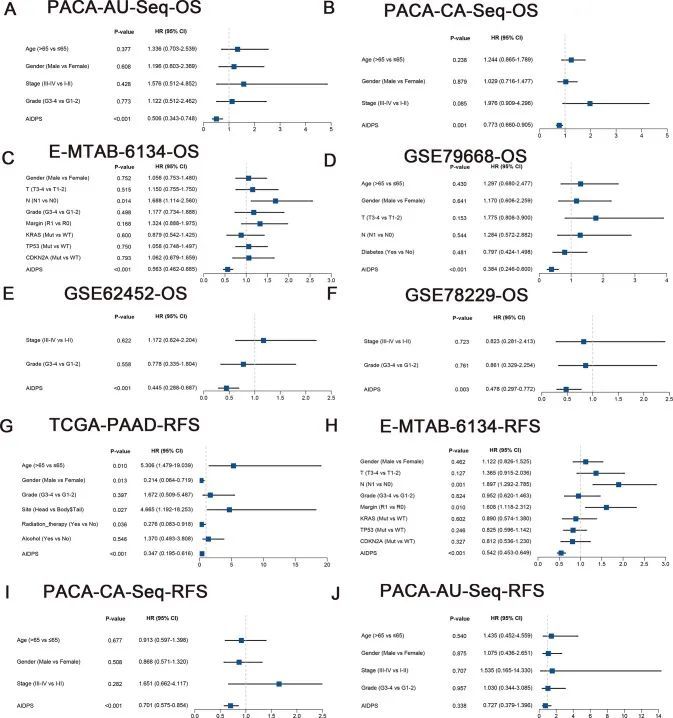
在九个测试队列中,Kaplan–Meier曲线一致显示,与低AIDPS组相比,高AIDPS组的OS显著延长(均p<0.05,图3补充1A–I)。同样,RFS的比较也表明,在TCGA-PAAD(n=69,p=0.029)、PACA-CA-Seq(n=113,p=0.0023)和E-MTAB-6134(n=288,p<0.0001)队列中,高AIDPS组的患者的复发率显著低于低AIDPS组(图3,补充1J,L,M)。值得一提的是,PACA AU-Seq中只有28个样本拥有完整的RFS信息。尽管Kaplan–Meier分析显示出相应的趋势,但对数秩检验没有达到统计学显著性(p=0.063,图3,补充1K)。在调整了可用的临床病理特征后,如年龄、性别、TNM分期、分级、手术切缘、放疗史或饮酒史以及KRAS、TP53或CDKN2A突变,多变量Cox分析结果仍然表明AIDPS是OS的独立预后因素(均p<0.05,图3补充1N和2A-F)。一致的是,RFS的多变量结果还显示,AIDPS在TCGA-PAAD、PACA CA Seq和E-MTAB-6134中仍然具有统计学意义(均p<0.05,图3补充2G–I)。然而,鉴于PACA AU-Seq的样本量较小,p值在统计学上并不显著(p=0.338,图3,补充2J)。
3. AIDPS的Robust预测性能
为了测量AIDPS的辨别力,作者绘制了校准曲线和受试者操作特性(ROC)曲线。PACA AU阵列训练队列和Meta队列的校准曲线均显示AIDPS具有良好的预测性能(图3I和J)。在PACA AU阵列训练队列中,1年、2年和3年OS的ROC曲线下面积(AUCs)分别为0.715、0.748和0.671,在Meta队列中分别为0.717、0.719和0.719(图3K和L)。在九个测试队列中也发现了类似的优异结果,TCGA-PAAD中分别为0.705、0.711和0.797;PACA AU序列中的0.749、0.808和0.827;PACA CA序列中的0.662、0.683和0.691;E-MTAB-6134中的0.773、0.698和0.675;GSE62452中的0.676、0.787和0.834;GSE28735中的0.734、0.865和0.871;GSE78229中的0.669、0.809和0.844;GSE79668中的0.791、0.761和0.786;GSE85916中分别为0.748、0.766和0.811(图3-图补充3A–I)。多个独立队列中AUCs大于0.65的结果表明,作者的AIDPS可以稳定而稳健地预测PACA患者的预后。
在临床环境中,某些临床病理特征,如手术切缘、分期和分级,用于预后评估、临床分层管理和治疗决策(Ferrone等人,2005)。因此,作者在包含临床信息的八个队列中对比了AIDPS的预测疗效和这些常见的临床特征。C指数的结果表明,AIDPS比这些特征显著提高了准确性,包括年龄、性别、种族、糖尿病史、TNM分期、分级、原发部位、放疗或饮酒史、手术切缘以及KRAS、TP53或CDKN2A突变(图2C-J)。
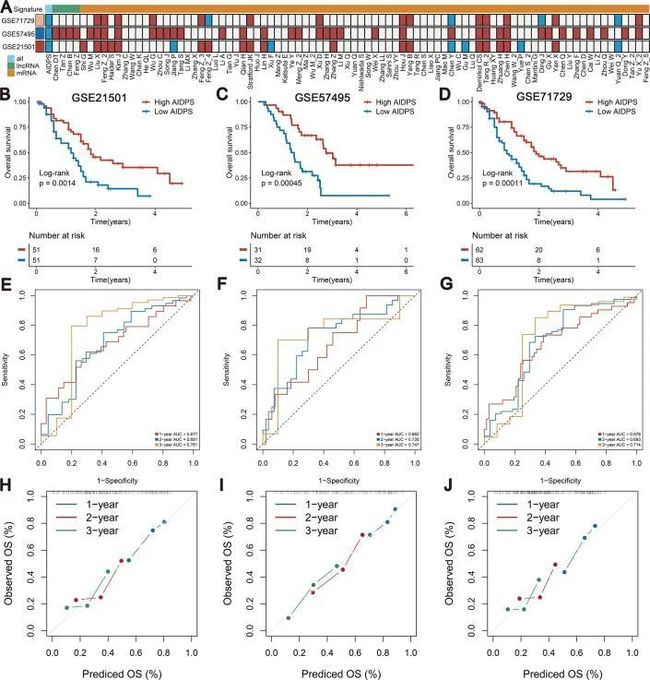
此外,为了更严格地验证作者的模型,作者在验证队列中评估了AIDPS的预测性能。Kaplan–Meier生存率分析表明,在三个外部验证队列中,高AIDPS组的OS显著延长(GSE21501中的log rank p=0.0014,GSE57495中的log rank p=0.00045,GSE71729中的log-rank p=0.00011,图3补充4B–D)。GSE21501的1年、2年和3年OS的AUC分别为0.677、0.681和0.761,GSE57495的AUC为0.682、0.728和0.747;GSE71729中的0.676、0.693和0.714(图3-图补充4E-G)。校准曲线也证实了AIDPS具有良好的预测性能(图3-图补充4H–J)。
总体而言,训练队列、9个测试队列、Meta队列和3个外部验证队列的Kaplan–Meier生存率分析、Cox回归分析、时间ROC曲线、C指数和校准曲线结果一致表明,AIDPS可以准确而稳健地预测PACA患者的预后,这表明AIDPS可能成为一种有吸引力的服务于临床实践的工具。
4. 对先前在PACA中公布的86个基因特征的重新评估
高通量测序的快速发展为肿瘤的分层管理和精确治疗提供了思路。近年来,基于大量高质量数据,通过LASSO和Stepwise Cox等机器学习算法构建了大量PACA的预后特征。因此,作者还收集了86个已发表的信使核糖核酸/lncRNA预后特征,以比较AIDPS和这些模型的预测准确性(图4)。由于缺乏必要的miRNA表达信息,包含miRNA的特征被排除在外。单变量Cox回归结果显示,在所有13个独立队列和Meta队列中,只有作者的AIDPS和Demirkol CS的20个基因特征具有一致的统计学意义(图4A,图3补充4A)。

图4 人工智能衍生的预后特征(AIDPS)和86个基于表达的特征之间的比较
补充图4-1 人工智能衍生的预后标志(AIDPS)与三个验证队列中86个已发表的标志以及9个AIDPS基因的其他方法构建的模型的比较
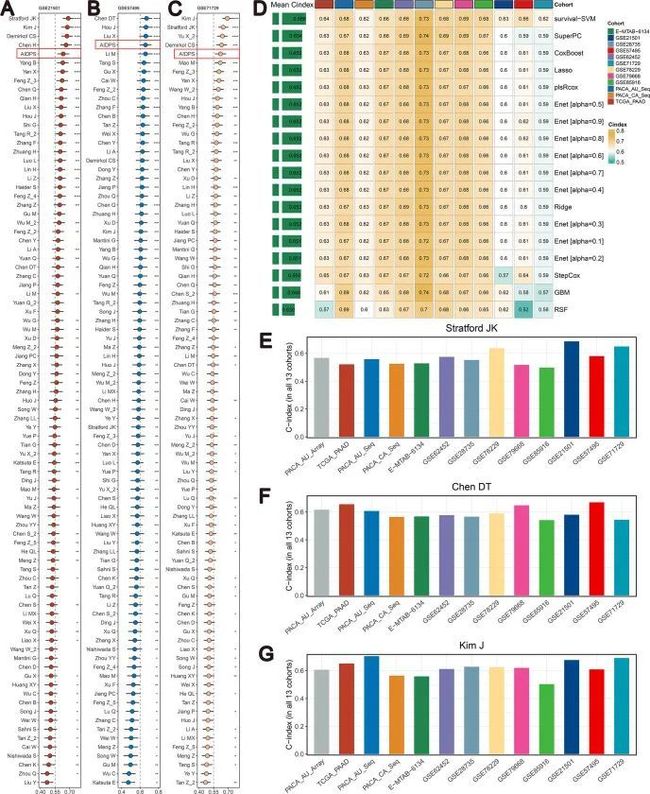
然后,作者通过C指数在训练队列、9个测试队列和Meta队列中比较了AIDPS和这86个特征的预测能力(图4B)。作者的AIDPS在几乎所有队列中都表现出明显优于其他模型的准确性(在四个队列中排名第一,在三个队列中位列第二,在两个队列中名列第三),揭示了AIDPS的稳健性。值得注意的是,在他们的TCGA-PAAD训练队列中,各种预后特征具有较高的C指数,但在其他队列中表现不佳,这可能是由于过度拟合导致的泛化能力受损(图4B)。
此外,作者的AIDPS在三个外部验证队列中也具有稳健的预测性能,在GSE57495队列中排名第四,在GSE21501和GSE71729队列中排名五,其表现优于几乎所有已发表的签名(图4图补充1A–C)。值得注意的是,尽管Stratford JK的六个基因特征在GSE21501和GSE71729队列中明显优于AIDPS,但它是在GSE2150 1队列中构建的,在其他队列中表现非常差,在GSE57495、TCGA-PAAD、PACA AU-Seq等中C指数甚至小于0.6(图4-图补充1E)。Chen DT的15个基因特征在他自己的训练队列GSE57495中具有显著优越的表现,但在GSE21501、GSE71729、PACA CA Seq和其他队列中表现不令人满意(图4-图补充1F)。类似地,Kim J的五个基因特征在训练队列GSE71729、GSE21501和PACA-AU-Seq队列中表现良好,但在大多数其他队列中表现非常差,如GSE85916、GSE57495、PACA-CA-Seq和E-MTAB-6134(图4图补充1G)。此外,尽管Demirkol CS的20基因签名、Chen H的3基因签名、Hou J的6基因签名、Liu X的7基因签名和Yu X_2的6基因标记在其训练队列或其他少数队列中优于AIDPS,但只有作者的AIDPS在所有PACA队列中具有可接受的性能,绝大多数队列表现良好,C指数大于0.65(图4B,图4-图1A–C)。总之,上述结果表明,9个基因的AIDPS可以有力地预测PACA患者的预后,并且更少的基因可能使其更有临床推广价值。
5. AIDPS的临床特征
作者进一步比较了高AIDPS组和低AIDPS组之间的几个常见临床特征,结果表明在年龄、性别和TNM分期方面没有统计学差异(图5A-C,图5-图1A–L)。然而,AIDPS较低的患者具有更高的分级,这可能会导致其预后更差(图5D,图5-图补充1M-P)。
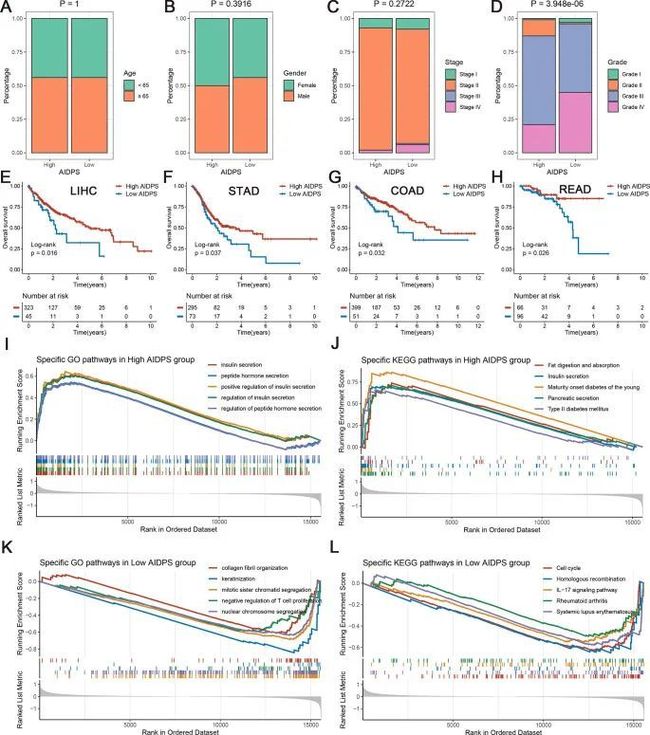
图5 高和低人工智能衍生预后标志(AIDPS)组的临床标志和功能特征

补充图5-1 高和低人工智能衍生预后标志(AIDPS)组的临床特征
此外,鉴于AIDPS在PACA中具有出色的预测能力,作者还测试了其在其他几种常见消化系统肿瘤中的表现。如图5E–H所示,低AIDPS组患者的Kaplan–Meier生存曲线在四种肿瘤中表现出显著的惨淡OS,包括LIHC(p=0.016)、STAD(p=0.037)、COAD(p=0.032)和READ(p=0.026)。这些结果支持了作者的假设,表明在PACA中构建的AIDPS作为一种生物标志物,具有广泛的推广到其他肿瘤的前景。
6. AIDPS的潜在生物学机制
应用基因集富集分析(GSEA)来阐明AIDPS的潜在功能途径。如图5I和J所示,高AIDPS组在消化和代谢相关途径上显著富集,如胰岛素分泌和调节、肽类激素分泌和调控、脂肪消化和吸收、胰腺分泌、年轻人成熟期糖尿病和Ⅱ型糖尿病。而低AIDPS组主要与T细胞增殖、IL-17信号通路和其他免疫相关通路的调节,以及细胞周期、核染色体分离、同源重组和其他增殖相关的生物学过程有关,这在一定程度上解释了其分级更高和预后更差(图5K和L)。
7. AIDPS的基因组改变景观
为了研究高AIDPS组和低AIDPS组之间的基因组异质性,作者对突变和拷贝数变化进行了全面分析(CNA,图6A)。如图6C所示,低AIDPS组具有显著更高的肿瘤突变负荷(TMB)。结合TCGA中的10种致癌信号通路(Sanchez-Vega et al.,2018),作者发现经典的抑癌基因TP53、CDKN2A和癌基因KRAS在低AIDPS组比高AIDPS组更频繁地突变,而SMAD4、TTN和RNF43则相反(图6A和B)。接下来,基于PACA中流行的突变特征,作者发现突变特征3(BRCA1/2突变相关)在高AIDPS组中富集,而突变特征1(年龄相关)在低AIDPS组更占主导地位。

图6 基于突变、拷贝数改变(CNA)和甲基化的多组学分析
此外,作者还进一步探讨了这两个群体的CNA景观。与高AIDPS组相比,低AIDPS组在局灶和染色体臂水平上具有明显更高的扩增或缺失,如8q24.21、19q13.2和8p111.22的扩增以及9p21.3、18q21.2、6p22.2和22q13.31的缺失(图6A和D)。在基因水平上,8q24.21内癌基因MYC的明显扩增以及9p21.3和18q21.2内抑癌基因CDKN2A、CDKN2B和SMAD4的明显缺失再次证实了这一结果(图6A)。总体而言,低AIDPS组的癌基因扩增和抑癌基因缺失可能导致其预后不良。
8. AIDPS的甲基化驱动事件
参考作者之前的过程(Liu et al.,2021b;Liu等人,2021c),作者筛选了四个甲基化驱动基因(MDGs),其甲基化水平与PACA中匹配的基因表达水平显著负相关。与低AIDPS组相比,高AIDPS组具有更高的MAP3K8和PCDH7甲基化水平以及显著更低的mRNA表达水平,而PCDHB1和SPAG6则相反(图6E和F)。此外,Kaplan–Meier分析显示,MAP3K8和PCDH7的较高甲基化水平以及SPAG6的较低甲基化水平显著延长了高AIDPS组的OS(均p<0.05,图6G、H和J)。PCDHB1也表现出与SPAG6相似的趋势,尽管没有达到统计学显著性(p=0.07,图6I)。
9. AIDPS的免疫景观与分子表达
上述GSEA显示,几种免疫相关途径在低AIDPS组中高度富集,因此作者研究了两组之间的免疫景观和免疫检查点分子(ICM)表达。根据单样本基因集富集分析(ssGSEA),作者发现低AIDPS组表现出相对较高的免疫细胞类型浸润丰度,包括活化的CD4+T细胞、CD56dim自然杀伤细胞、中央记忆CD8+T细胞,γ-ΔT细胞和2T型辅助细胞(均p<0.05,图7A和B)。此外,在作者纳入的27个ICM中,低AIDPS组的相对表达水平显著增加,如CD274、CD276、PDCD1LG2、CD40、CD70、TNFRSF18、TNFRSF 4、TNFRSF9和NT5E(图7C)。总之,这些结果一致表明,AIDPS低的PACA患者更有可能对免疫疗法产生反应。

图7 高和低人工智能衍生预后标志(AIDPS)组的免疫景观

补充图7-1

补充图7-2

补充图7-3
10. 9个AIDPS基因的预后价值和生物学相关性
基于来自训练队列、九个测试队列和三个外部验证队列的大型多中心生存数据,作者使用AIDPS及其九个基因作为连续变量,对生存变量进行了综合单变量Cox回归分析。如图7图补充1A所示,AIDPS在所有13个队列中都是一个独立的保护因子(与图3图补充1K一致,PACA AU-Seq中只有28个样本具有RFS信息,尽管表现出相应的趋势,但没有达到统计学意义)。相应地,由于32个CPG用于构建至少8/10个队列中具有一致预后价值的AIDPS,因此9个AIDPS基因在训练和9个测试队列中具有相对稳定的表现(图7图补充1B–J)。然而,在三个外部验证队列中,这九个AIDPS基因的较差表现很难令人满意。此外,基于PACA AU Array训练队列中这9个AIDPS基因的表达文件,作者通过10种常见的机器学习算法构建了18个模型(弹性网络的α值从0.1到0.9),在剩下的12个多中心队列中实现了0.666的最大平均C指数(图4-图补充1D)。也就是说,对于通过CoxBoost降维后从32个CPG中获得的9个基因,这些结果再次证实了作者之前的管道结果(图2A),通过生存支持向量机构建的AIDPS是最佳选择。总的来说,与仅9个AIDPS基因相比,作者的AIDPS带来了显著的性能改进。
为了描述AIDPS与其9个基因之间的生物学相关性,作者参考先前的研究进行了Pearson相关性分析(Zhang et al.,2020)。结果显示,与AIDPS显著正相关的SELENBP1和PLCB4与总体上与AIDPS呈显著负相关的7个基因呈中度负相关,且AIDPS TCGA-PAAD较低(图7、图补充2A和C)。有趣的是,高AIDPS组的总体相关性较低,许多基因表现出不同甚至相反的趋势(例如,SELENBP1与AIDPS和PLCB4从正相关变为负相关,图7、图补充2B)。此外,鉴于AIDPS在肿瘤微环境(TME)中的明显影响,作者比较了AIDPS及其9个基因与27个ICM和28种免疫细胞类型的关系,总体上,高AIDPS和低AIDPS的TCGA-PAAD。Pearson相关分析显示,AIDPS及其正相关的SELENBP1和PLCB4与ICMs和免疫细胞类型呈强负相关,而DCBLD2、PRR11、UNC13D、EREG和TGM2与AIDPS呈负相关,在整个和高AIDPS TCGA-PAAD中与ICMs和免疫细胞类型显著正相关(图7、图补充2D、E、G、H)。然而,低AIDPS组的趋势相反,AIDPS和PLCB4与ICMs和免疫细胞类型显著正相关,而DCBLD2、PRR11、UNC13D、EREG、ADM、CDCA4和TGM2与ICMs或免疫细胞类型呈负相关(图7-图补充2F和I)。
在基因组改变方面,作者还观察到,与AIDPS呈正相关的SELENBP1和PLCB4在具有较高突变和CNA频率的低AIDPS组中显著较低,这表明它们与TMB和CNA负荷显著负相关(图7-图补充3)。相应地,与AIDPS呈负相关的DCBLD2、PRR11、UNC13D、EREG、ADM、CDCA4和TGM2在低AIDPS组中显著增加,暗示它们与TMB和CNA负荷显著正相关(例如,低AIDPS小组具有更高的TP53突变和8q24.21扩增,以及更高的DCBLD2表达,图7、图补充3)。
11. AIDPS对免疫治疗的预测价值
鉴于低AIDPS组的患者具有更高的基因组改变频率和TMB,再加上他们相对活化的TME和增加的ICMs表达,作者推测低AIDPS的PACA患者对免疫疗法更敏感。基于肿瘤免疫功能障碍和排除(TIDE)网络工具,低AIDPS组的TIDE评分显著较低,免疫治疗应答率较高(图8A和B)。亚类映射(Submap)的结果还表明,低AIDPS患者的表达模式与对ICIs有反应的黑色素瘤患者的表达方式更相似(图8C)。总体而言,这些结果表明,低AIDPS组更有可能从免疫疗法中获益。

图8 评估治疗药物的益处
12. 为高AIDPS人群寻找潜在的治疗剂
如图8E所示,作者使用来自癌症治疗反应门户(CTRP)的敏感性数据为患有高AIDPS的PACA患者开发了潜在药物,包括481种化合物和835种癌症细胞系[CCLs],并在混合物(PRISM)(包括1448种化合物和482种CCLs)数据集中同时分析相对抑制(Yang等人,2021)。为了确保作者方案的可靠性,吉西他滨作为PACA的一线治疗方法,用于研究估计的敏感性和临床实践是否一致。一项实验室研究发现,在PACA中,提高PAK1活性是吉西他滨耐药性所必需的,并且PAK1抑制增强了吉西他宾的疗效。与这项研究一致,作者的结果显示,PAK1表达低的患者具有明显较低的AUC估计值,这表明对吉西他滨的敏感性更高(图8F)。之后,作者应用该公式来确定高AIDPS组的潜在敏感药物,并最终产生了四种CTRP衍生药物(布雷费尔德素A、寡霉素A、哇巴因和帕诺比司他)和九种PRISM衍生药物(阿司匹林、BAY-87-2243、EVP4593、GSK2656157、I-BET151、LY303511、OTX015、奥沙利铂和XL388)。这些药物的估计AUC值不仅与AIDPS评分呈统计学负相关,而且在高AIDPS组中显著较低(图8G-J)。
此外,基于PACA患者和正常对照之间的差异表达谱,作者进一步应用了连接图(CMap,https://clue.io/)用于鉴定PACA候选化合物的工具。在与CTRP和PRISM获得的结果进行交集后,作者最终得到了两个候选化合物:ATP酶抑制剂ouabain和组蛋白脱乙酰酶(HDAC)抑制剂panobinostat。其中,CMap评分为–98.11的帕诺比诺司他对PACA患者高度敏感,这表明它可能成为高AIDPS组PACA患者的潜在治疗剂(图8D)。
总结
总之,基于来自训练队列、9个测试队列和3个外部验证队列的32个CPG,作者通过76个机器学习算法组合构建并验证了一致预后特征(称为AIDPS)。在结合了几个重要的临床病理特征和86个已发表的特征后,AIDPS还表现出强大且显著优越的预测能力。值得注意的是,作者的AIDPS对PACA的临床管理和个体化治疗具有重要的临床意义,低AIDPS患者对免疫疗法更敏感,而帕诺司他可能是高AIDPS患者的潜在药物。此外,在其他流行的消化系统肿瘤中,9个基因的AIDPS仍然可以准确地对预后进行分层,这表明推断的可能性很大。总之,作者的研究为临床实践中PACA患者的预后评估、风险分层和个体化治疗提供了一个有吸引力的工具。