根据脑图谱获取感兴趣区域的mask
根据脑图谱获取感兴趣区域的mask
- 1,引入
-
- 1.1 ASPECT-Atlas
- 2,获取脑图谱感兴趣区域mask
- 参考:
1,引入
脑影像分析中,我们常常会针对性的对某些感兴趣区域进行分析,而对它们进行分析的前提是获取该区域的mask。感兴趣区域可以用以某些坐标为球心的球形区域定义,也可以用脑图谱上对应的某些脑区定义,其中,后者是较为常见的,也是我们今天要讨论的。脑图谱是一类特殊脑影像,它的每一个位置上不是信号值,而是脑区编号(标签),这也就意味着我们可以通过感兴趣区域的脑区编号,得到对应的感兴趣区域位置的集合,它也就是感兴趣区域的mask。
本文参考自:【MRI脑影像分析——根据脑图谱获取感兴趣区域mask,以海马体与丘脑为例(matlab+nilearn+nibabel+REST1.8)】。https://blog.csdn.net/sinat_35907936/article/details/118481241
1.1 ASPECT-Atlas
脑图谱是一类特殊脑影像,它的每一个位置上不是信号值,而是脑区编号(标签)。任何一个图谱都会有其对应的脑区编号(标签)表。非常常见的是一些包含海马体,丘脑等部位的脑图谱。
本文是做ASPECT评分任务的,为了更方便讲述,下面主要以ASPECT-Altas来进行讲述。
如下图所示为ASPECT的图谱。如果需要此图谱的可以私聊我。

这个图谱的对应标签值为:

我们可以看到,当点击图谱的M1左区域时,itk-ansp可以显示其标签强度值为10. 与列表相同。

以下将使用ASPECT-Atlas,提取岛叶,尾状核等所有部位,并保存成nii.gz格式数据。
这里主要使用python的方式,使用nibabel函数库。
2,获取脑图谱感兴趣区域mask
nibabel是python平台上用于解析神经影像的工具,不仅可以解析nifti也可以解析gifti。这里用它来解析并封装nifti,以生成mask。mask类型与保持上面一致。
pip install nibabel
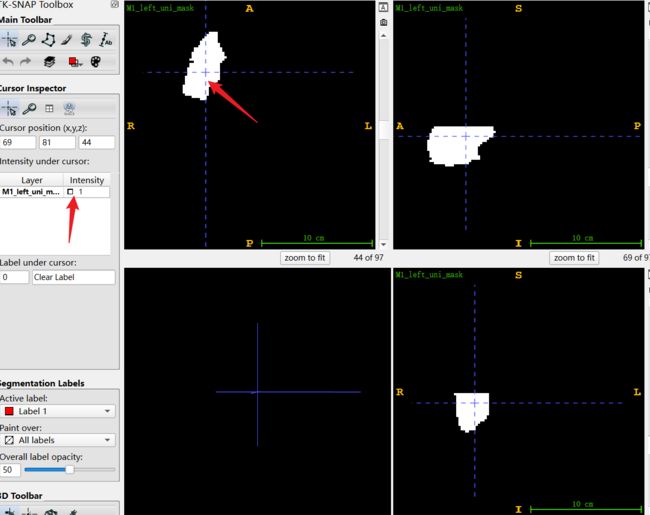
提取M1左,对应标签:10.
一般建议选ROI内保存成1,区域外为0.
import numpy as np
import nibabel as nib
area_name = 'm1_left'
ROI_label = 10
img = nib.load('./templete/AspectsAtlas_padding_2mm.nii.gz')
data = img.get_fdata()
mask = np.zeros(data.shape)
# 创建mask
mask[data==ROI_label] = 1
# 用原先的仿射矩阵与头包装mask成nifti
#区域内为1,区域外为0,一个编号
new_img = nib.Nifti1Image(mask,img.affine, header=img.header)
new_img.to_filename(area_name + '_uni_mask.nii')
# 区域内为原来编号,区域外为0
data = np.array(mask*data, dtype=np.uint8)
new_img = nib.Nifti1Image(data, img.affine, header=img.header)
new_img.to_filename(area_name + '_sep_mask.nii')
下面展示一次性读取excel表所有脑区,并保存:
import numpy as np
import nibabel as nib
# 读取excel
import pandas as pd
# 指定 Excel 文件路径
excel_file_path = './templete/ASPECTAtlas_label_lookup.xlsx' # 替换为你的Excel文件路径
# 读取 Excel 文件
df = pd.read_excel(excel_file_path)
# 提取两列数据到列表
column1_data = df['parts'].tolist() # 替换 'Column1' 为第一列的实际列名
column2_data = df['label'].tolist() # 替换 'Column2' 为第二列的实际列名
print(column1_data,column2_data)
# 读取ASPECT-Atlas
img = nib.load('./templete/AspectsAtlas_padding_2mm.nii.gz')
data = img.get_fdata()
for i, name in enumerate(column1_data):
mask = np.zeros(data.shape)
ROI_label = column2_data[i]
area_name = './ASPECT_Region_3D/' + name
# 创建mask
mask[data == ROI_label] = 1
# 用原先的仿射矩阵与头包装mask成nifti
# 区域内为1,区域外为0,一个编号
new_img = nib.Nifti1Image(mask,img.affine, header=img.header)
new_img.to_filename(area_name + '_uni_mask.nii.gz')
参考:
[MRI脑影像分析——根据脑图谱获取感兴趣区域mask,以海马体与丘脑为例(matlab+nilearn+nibabel+REST1.8)]
https://blog.csdn.net/sinat_35907936/article/details/118481241