【练一下1】糖尿病遗传风险检测挑战赛 【讯飞开放平台】
文章目录
- 完成coggle任务
-
- 任务1:报名比赛
-
- 步骤1:报名比赛http://challenge.xfyun.cn/topic/info?type=diabetes&ch=ds22-dw-zmt05
-
- 赛事
- 提交说明
- 步骤2:下载比赛数据(点击比赛页面的赛题数据)
-
- 字段说明
- 步骤3:解压比赛数据,并使用pandas进行读取;
- 步骤4:查看训练集和测试集字段类型,并将数据读取代码写到博客;
- 任务2:比赛数据分析
-
- 步骤1:统计字段的缺失值,计算缺失比例;
- 步骤2:分析字段的类型;
- 步骤3:计算字段相关性;
-
- 通过.corr()计算字段之间的相关性;
- 可视化
- 补充学习 b站7.1录播
-
- groupby
-
- apply
- agg
- map、transform
- 可视化
- 特征工程
-
- onehot
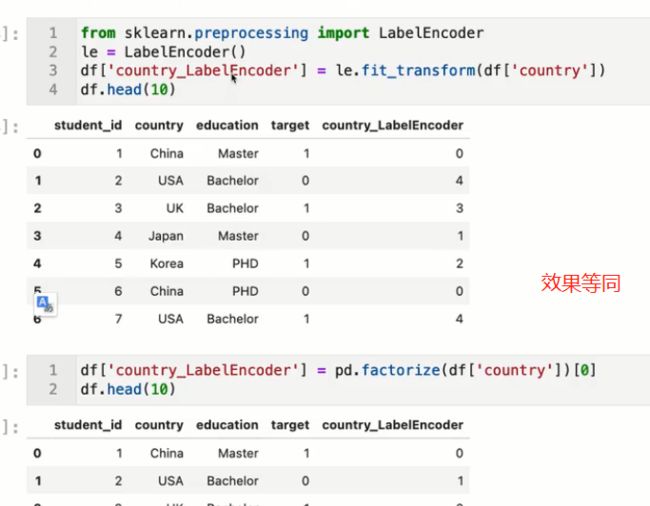
- labelEncoder
- 有序变量
- 任务3:逻辑回归尝试
-
- 步骤1:导入sklearn中的逻辑回归;
- 步骤1.5 数据预处理
- 步骤2:使用训练集和逻辑回归进行训练,并在测试集上进行预测;
- 步骤3:将步骤2预测的结果文件提交到比赛,截图分数;
- 步骤4:将训练集20%划分为验证集,在训练部分进行训练,在测试部分进行预测,调节逻辑回归的超参数;
- 任务4:特征工程(使用pandas完成)
-
- 步骤1:统计每个性别对应的【体重指数】、【舒张压】平均值
- 步骤2:计算每个患者与每个性别平均值的差异;
- 步骤3:在上述基础上将训练集20%划分为验证集,使用逻辑回归完成训练,精度是否有提高?
- 步骤4:思考字段含义,尝试新的特征,将你的尝试写入博客;
- 任务5:特征筛选
-
- 步骤1:使用树模型完成模型的训练,通过特征重要性筛选出Top5的特征;
- 步骤2:使用筛选出的特征和逻辑回归进行训练,在验证集精度是否有提高?
- 步骤3:如果有提高,为什么?如果没有提高,为什么?
- 步骤4:将你的尝试写入博客;
- 任务6:高阶树模型
-
- 步骤1:安装LightGBM,并学习基础的使用方法;
- 步骤2:将训练集20%划分为验证集,使用LightGBM完成训练,精度是否有提高?
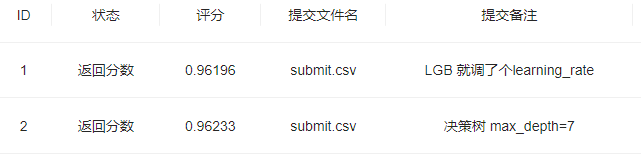
- 步骤3:将步骤2预测的结果文件提交到比赛,截图分数;
- 步骤4:尝试调节搜索LightGBM的参数;
- 步骤5:将步骤4调参之后的模型从新训练,将最新预测的结果文件提交到比赛,截图分数;
- 任务7:多折训练与集成
-
- 步骤1:使用KFold完成数据划分;
- 步骤2:使用StratifiedKFold完成数据划分;
- 步骤3:使用StratifiedKFold配合LightGBM完成模型的训练和预测
- 步骤4:在步骤3训练得到了多少个模型,对测试集多次预测,将最新预测的结果文件提交到比赛,截图分数;
- 步骤4:使用交叉验证训练5个机器学习模型(svm、lr等),使用stacking完成集成,将最新预测的结果文件提交到比赛,截图分数;
-
- 集成
- 总结
完成coggle任务
任务1:报名比赛
步骤1:报名比赛http://challenge.xfyun.cn/topic/info?type=diabetes&ch=ds22-dw-zmt05
赛事
在这次比赛中,您需要通过训练数据集构建糖尿病遗传风险预测模型,然后预测出测试数据集中个体是否患有糖尿病。对于测试数据集当中的个体,您必须预测其是否患有糖尿病(患有糖尿病:1,未患有糖尿病:0),预测值只能是整数1或者0。
训练集(比赛训练集.csv)一共有5070条数据,用于构建您的预测模型(您可能需要先进行数据分析)。数据的字段有编号、性别、出生年份、体重指数、糖尿病家族史、舒张压、口服耐糖量测试、胰岛素释放实验、肱三头肌皮褶厚度、患有糖尿病标识(最后一列),您也可以通过特征工程技术构建新的特征。
测试集(比赛测试集.csv)一共有1000条数据,用于验证预测模型的性能。数据的字段有编号、性别、出生年份、体重指数、糖尿病家族史、舒张压、口服耐糖量测试、胰岛素释放实验、肱三头肌皮褶厚度。
提交说明
对于测试数据集当中的个体,您必须预测其是否患有糖尿病(患有糖尿病:1,未患有糖尿病:0),预测值只能是整数1或者0。提交的数据应该具有如下格式:
uuid,label
1,0
2,1
3,1
…
本次比赛中,预测模型的结果文件需要命名成:预测结果.csv,然后提交。请确保您提交的文件格式规范。
步骤2:下载比赛数据(点击比赛页面的赛题数据)
字段说明
编号:标识个体身份的数字;
性别:1表示男性,0表示女性;
出生年份:出生的年份;
体重指数:体重除以身高的平方,单位kg/m2;
糖尿病家族史:标识糖尿病的遗传特性,记录家族里面患有糖尿病的家属,分成三种标识,分别是父母有一方患有糖尿病、叔叔或者姑姑有一方患有糖尿病、无记录;
舒张压:心脏舒张时,动脉血管弹性回缩时,产生的压力称为舒张压,单位mmHg;
口服耐糖量测试:诊断糖尿病的一种实验室检查方法。比赛数据采用120分钟耐糖测试后的血糖值,单位mmol/L;
胰岛素释放实验:空腹时定量口服葡萄糖刺激胰岛β细胞释放胰岛素。比赛数据采用服糖后120分钟的血浆胰岛素水平,单位pmol/L;
肱三头肌皮褶厚度:在右上臂后面肩峰与鹰嘴连线的重点处,夹取与上肢长轴平行的皮褶,纵向测量,单位cm;
患有糖尿病标识:数据标签,1表示患有糖尿病,0表示未患有糖尿病。
步骤3:解压比赛数据,并使用pandas进行读取;
import pandas as pd
train_df = pd.read_csv("./比赛训练集.csv", encoding='gbk')
test_df = pd.read_csv("./比赛测试集.csv", encoding='gbk')
print(train_df.shape, test_df.shape)
print(train_df.dtypes, test_df.dtypes)
步骤4:查看训练集和测试集字段类型,并将数据读取代码写到博客;
任务2:比赛数据分析
步骤1:统计字段的缺失值,计算缺失比例;
通过缺失值统计,训练集和测试集的缺失值分布是否一致?
通过缺失值统计,有没有缺失比例很高的列?
#缺失值计算
train_df.isnull().mean(0)
test_df.isnull().mean(0)
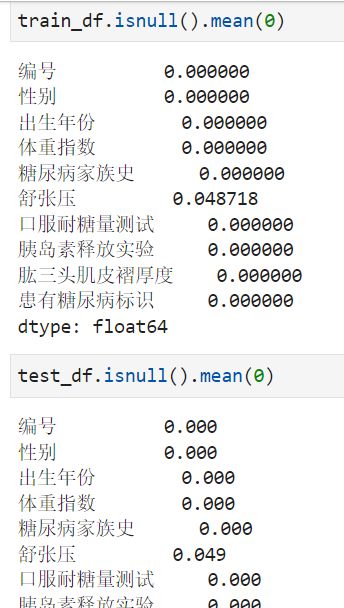
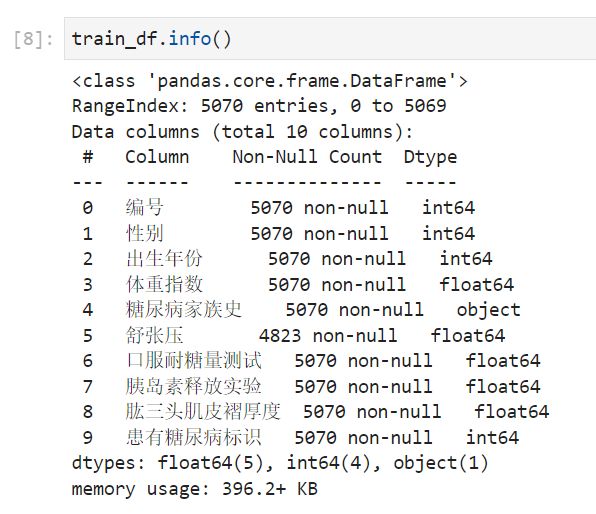
可以看出舒张压这个字段缺失了 4.9%。测试集和训练集比例差不多的。
步骤2:分析字段的类型;
从info和head(查看前5行的数据)可以看出,体重,舒张压,口服耐糖量测试 ,胰岛素释放实验,肱三头肌皮褶厚度是连续型,其余都是整型(糖尿病标识是0 1)。家族史那一列是备注(后期这个特征应该要由字符型可以处理为整型加入模型的使用)。
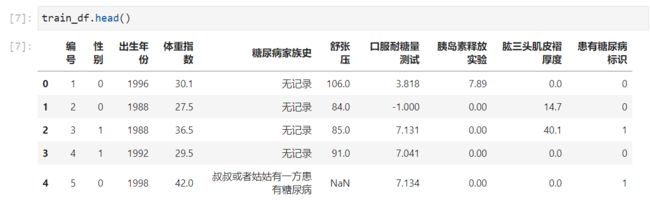
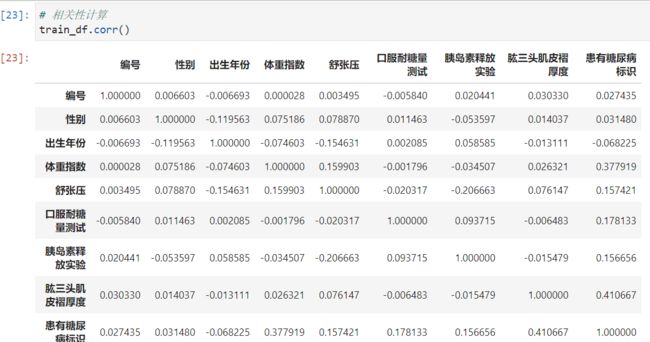
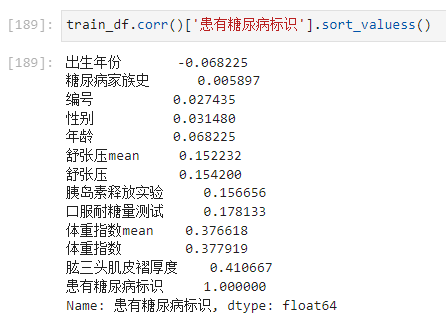
步骤3:计算字段相关性;
通过.corr()计算字段之间的相关性;
# 相关性计算
train_df.corr()
- 可以看出 肱三头肌皮褶厚度和体重系数和糖尿病关系比较大。
可视化
尝试使用其他可视化方法将字段 与 标签的分布差异进行可视化;
以下也是用图表看一下糖尿病之间的分布。
之前没用过seaborn,boxplot应该是箱式图,countplot,和violinplot就不知道了
查了一下,countplot就是直方图,violinplot是小提琴样式的箱线图。
出现上图的原因是1.seaborn的几个图都叠在一起了,2.我语言设置没有切换为中文也没用(之后加入设置代码,这样中文和负坐标轴的符号才能正常显示),从老师那里偷的图片设置svg语句,更清楚一些
import seaborn as sns
import matplotlib.pyplot as plt
plt.rcParams['font.family'] = ['SimHei'] #用来正常显示中文标签
plt.rcParams['axes.unicode_minus'] = False #用来正常显示负号
from IPython.display import set_matplotlib_formats
set_matplotlib_formats('svg')#展示图片格式
# train_df['性别'].value_counts().plot(kind='barh')# barh水平的,kind类型
# sns.countplot(x='患有糖尿病标识', hue='性别', data=train_df)#hue分列 value_counts和 countplot其实是一样的
# sns.boxplot(y='出生年份', x='患有糖尿病标识', hue='性别', data=train_df)
sns.violinplot(y="体重指数", x="患有糖尿病标识", hue="性别", data=train_df)
依次注释图像语句,得到下列图片

1是男性,0是女性。女性数据多一些
barh 水平的

横坐标轴0是没得糖尿病的,蓝色竖条0是女性。反正都是女性多一些,这也看不出糖尿病和性别的相关关系。

男性黄条的都算正态分布吧,女性蓝条的有点上移,不怎么正态。没得糖尿病的男的,有几个很长寿的点(离群点)。
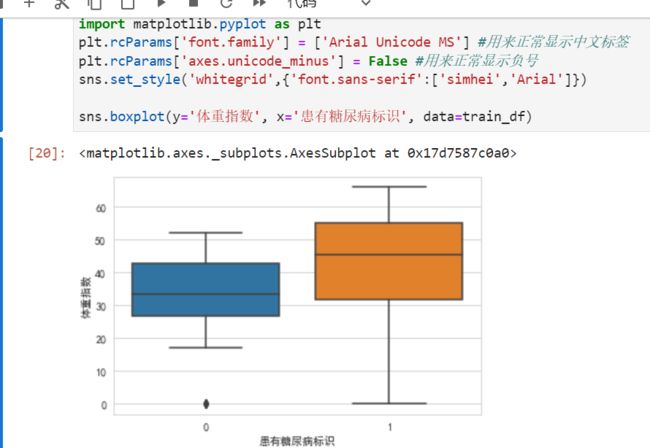
糖尿病人的体重分布比正常的要偏上一些。
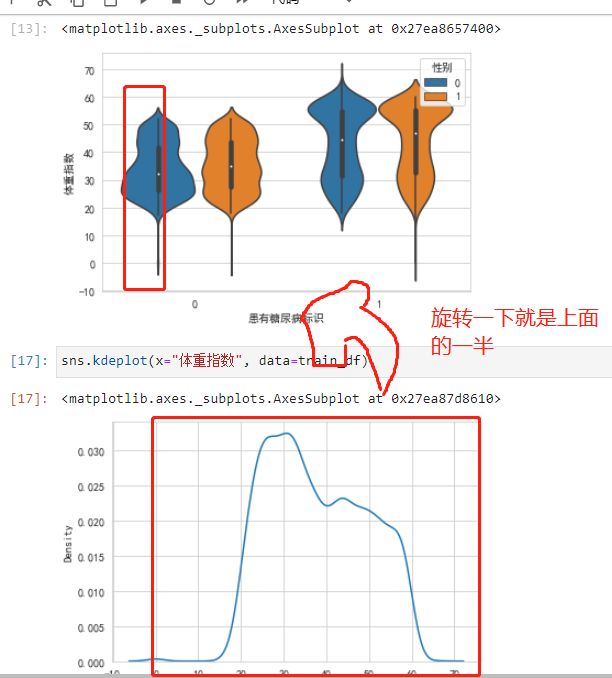
小提琴图其实就是测过来的密度直方图,对称的,看一半就好了.
scatterplot 散点图,xy都是数值型
然后 每个图都有一些 属性,像什么填充fill 还有 alpha透明度,自己可以去官网学。
tips:根据xy轴的数据类型来定用什么图
根据这些来判断能不能 做交叉特征(两列的加减乘除),能不能分离
补充学习 b站7.1录播
groupby
apply
agg
- groupby 和 agg 可以实现优雅的聚合效果
map、transform
 map可以映射改变 键值
map可以映射改变 键值
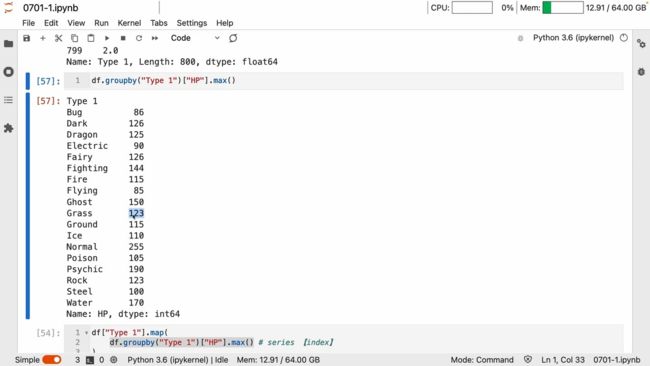
groupby 一系列操作又是得到键值关系
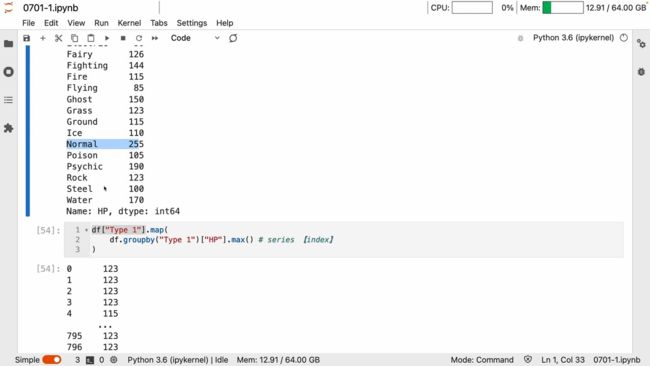
所以 map里面写个groupby 自然而然就可以映射替换了
可视化
见上面步骤三的
特征工程
onehot
labelEncoder
有序变量
任务3:逻辑回归尝试
步骤1:导入sklearn中的逻辑回归;
import pandas as pd
from sklearn.linear_model import LogisticRegression
from sklearn.preprocessing import MinMaxScaler
from sklearn.pipeline import make_pipeline
导入对应的包
步骤1.5 数据预处理
我觉得因为家族史根据常识可以当成有序变量,而且‘叔叔或者姑姑有一方患有糖尿病’和‘叔叔或姑姑有一方患有糖尿病’其实(含义一样)是一类,如果直接用labelEncoder就会变成两类,是不对的,所以用map处理。
train_df = pd.read_csv('./糖尿病遗传风险预测挑战赛公开数据/比赛训练集.csv', encoding='gbk')
test_df = pd.read_csv('./糖尿病遗传风险预测挑战赛公开数据/比赛测试集.csv', encoding='gbk')
dict_糖尿病家族史 = {
'无记录': 0,
'叔叔或姑姑有一方患有糖尿病': 1,
'叔叔或者姑姑有一方患有糖尿病': 1,
'父母有一方患有糖尿病': 2
}
train_df['糖尿病家族史'] = train_df['糖尿病家族史'].map(dict_糖尿病家族史)
test_df['糖尿病家族史'] = test_df['糖尿病家族史'].map(dict_糖尿病家族史)
train_df['舒张压'].fillna(89, inplace=True)
test_df['舒张压'].fillna(89, inplace=True)

舒张压用平均值或者中位数都是89这个值这里,而且观察原来数据都是整数,所以就用89了。

补全之后,没有缺失值了。

家族史和糖尿病标识之间的相关性,这么低吗。(意外,明明地球OL里面,遗传是导致糖尿病的重要因素)
步骤2:使用训练集和逻辑回归进行训练,并在测试集上进行预测;
读数据-》将有序变量转换为数值变量(利用map)
根据前面数据分析,(4.9%的缺失不至于删除),所以补全缺失值
from sklearn.linear_model import LogisticRegression as LR
from sklearn.model_selection import cross_val_score
from sklearn.feature_selection import SelectFromModel
import numpy as np
import pandas as pd
from sklearn.metrics import accuracy_score
from sklearn.preprocessing import MinMaxScaler
from sklearn.pipeline import make_pipeline
from sklearn.tree import DecisionTreeClassifier
from sklearn.model_selection import train_test_split
from sklearn.model_selection import GridSearchCV
from sklearn.model_selection import cross_val_score
features=[f for f in train_df.columns if f not in ['编号','患有糖尿病标识','出生年份']]
X=train_df[features]
y=train_df['患有糖尿病标识']
Xtrain, Xtest, Ytrain, Ytest = train_test_split(X,y,test_size=0.2,random_state=2022)
#修正索引
for i in [Xtrain, Xtest, Ytrain, Ytest]:
i.index=range(i.shape[0])
#用图像来确认
l1 = []
l2 = []
l1test = []
l2test = []
for i in np.linspace(0.05,1,19):
lrl1 = LR(penalty="l1",solver="liblinear",C=i,max_iter=1000)
lrl2 = LR(penalty="l2",solver="liblinear",C=i,max_iter=1000)
lrl1 = lrl1.fit(Xtrain,Ytrain)
l1.append(accuracy_score(lrl1.predict(Xtrain),Ytrain))
l1test.append(accuracy_score(lrl1.predict(Xtest),Ytest))
lrl2 = lrl2.fit(Xtrain,Ytrain)
l2.append(accuracy_score(lrl2.predict(Xtrain),Ytrain))
l2test.append(accuracy_score(lrl2.predict(Xtest),Ytest))
graph = [l1,l2,l1test,l2test]
color = ["green","black","lightgreen","gray"]
label = ["L1","L2","L1test","L2test"]
plt.figure(figsize=(6,6))
for i in range(len(graph)):
plt.plot(np.linspace(0.05,1,19),graph[i],color[i],label=label[i])
plt.legend(loc=4)
plt.show()
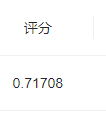
步骤3:将步骤2预测的结果文件提交到比赛,截图分数;
我是看 l2正则化的逻辑回归,然后再测试集上的表现好一点,然后又通过缩小空间,找到了表现最好的C值,结果交上去,才71.(可能是我把 出生年份 给剔除掉的原因)也有可能识别的原因 谁知道呢
lrl2 = LR(penalty="l2",solver="liblinear",C=0.395,max_iter=1000)
lrl2 = lrl2.fit(Xtrain,Ytrain)
test_df['label'] = lrl2.predict(test_df.drop(['编号','出生年份'], axis=1))
test_df.rename({'编号': 'uuid'}, axis=1)[['uuid', 'label']].to_csv('submit.csv', index=None)
步骤4:将训练集20%划分为验证集,在训练部分进行训练,在测试部分进行预测,调节逻辑回归的超参数;
(看了一下别人博客,也没规定l1,l2,就默认l2,然后C=1e2,然后评分就73)
步骤4:如果精度有提高,则重复步骤2和步骤3;如果没有提高,可以尝试树模型,重复步骤2、3;
#网格搜索
parameters = {
'splitter':('best','random')
,'criterion':("gini","entropy")
,"max_depth":[*range(1,10)]
# 'min_samples_leaf':[*range(1,50,5)]
# ,'min_impurity_decrease':[*np.linspace(0,0.5,20)]
}
clf = DecisionTreeClassifier(random_state=25)
GS = GridSearchCV(clf, parameters, cv=10)
GS.fit(Xtrain,Ytrain)
GS.best_params_
# GS.best_score_
#画曲线去找
tr=[]
te=[]
for i in range(40):
clf=DecisionTreeClassifier(random_state=25
,max_depth=7
,criterion="entropy"
,min_samples_leaf=i+1
)
clf=clf.fit(Xtrain,Ytrain)
score_tr=clf.score(Xtrain,Ytrain)
score_te=cross_val_score(clf,X,y,cv=10).mean()
tr.append(score_tr)
te.append(score_te)
print(max(te),te.index(max(te)))
plt.plot(range(1,41),tr,color='red',label='train')
plt.plot(range(1,41),te,color='green',label='test')
plt.xticks(range(1,41))
plt.legend()
plt.show()
提交答案
clf = DecisionTreeClassifier(random_state=25
,min_samples_leaf=25
,max_depth=7
,criterion="entropy"
)
clf.fit(Xtrain,Ytrain)
score=cross_val_score(clf,X,y,cv=10).mean()
score
任务4:特征工程(使用pandas完成)
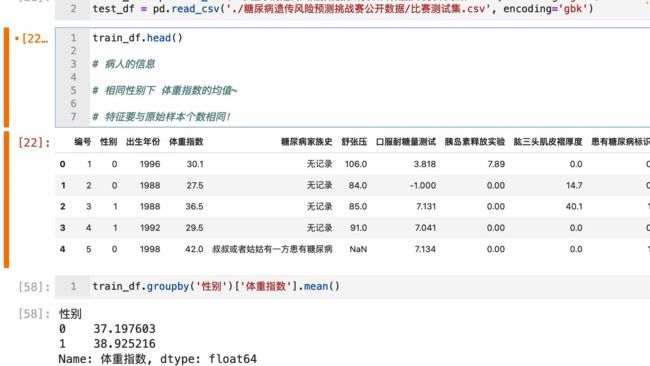
步骤1:统计每个性别对应的【体重指数】、【舒张压】平均值
train_df.groupby("性别").agg({'体重指数':['mean'],'舒张压':['mean']})
步骤2:计算每个患者与每个性别平均值的差异;
train_df['体重指数mean']=train_df['体重指数']-train_df['性别'].map(
train_df.groupby("性别")['体重指数'].mean()
)
train_df['舒张压mean']=train_df['舒张压']-train_df['性别'].map(
train_df.groupby("性别")['舒张压'].mean()
)
步骤3:在上述基础上将训练集20%划分为验证集,使用逻辑回归完成训练,精度是否有提高?
步骤4:思考字段含义,尝试新的特征,将你的尝试写入博客;
import pandas as pd
data={'featureName':features,'importances':clf.feature_importances_.tolist()}
df = pd.DataFrame(data)
df=df.sort_values(by=['importances'],ascending=False)
df
任务5:特征筛选
步骤1:使用树模型完成模型的训练,通过特征重要性筛选出Top5的特征;
根据上面的图片可以看出
体重指数 0.316459
肱三头肌皮褶厚度 0.252025
舒张压 0.149648
口服耐糖量测试 0.149392
胰岛素释放实验 0.116728
步骤2:使用筛选出的特征和逻辑回归进行训练,在验证集精度是否有提高?
提高了
步骤3:如果有提高,为什么?如果没有提高,为什么?

划重点了,考的好了一点点。
但这肯定不是明显提高,模型到极限了,逻辑回归也就这了。
步骤4:将你的尝试写入博客;
任务6:高阶树模型
import pandas as pd
import numpy as np
import seaborn as sns
from sklearn.model_selection import KFold
import lightgbm as lgb
# 读取数据
train_df = pd.read_csv('./糖尿病遗传风险预测挑战赛公开数据/比赛训练集.csv', encoding='gbk')
test_df = pd.read_csv('./糖尿病遗传风险预测挑战赛公开数据/比赛测试集.csv', encoding='gbk')
# 基础特征工程
train_df['体重指数_round'] = train_df['体重指数'] // 10
test_df['体重指数_round'] = train_df['体重指数'] // 10
# //地板除 Python中两个斜杠即双斜杠(//)表示地板除,即先做除法(/),然后向下取整(floor)。
train_df['口服耐糖量测试'] = train_df['口服耐糖量测试'].replace(-1, np.nan)
test_df['口服耐糖量测试'] = test_df['口服耐糖量测试'].replace(-1, np.nan)
# 这个特征里面有-1 懵逼了
dict_糖尿病家族史 = {
'无记录': 0,
'叔叔或姑姑有一方患有糖尿病': 1,
'叔叔或者姑姑有一方患有糖尿病': 1,
'父母有一方患有糖尿病': 2
}
train_df['糖尿病家族史'] = train_df['糖尿病家族史'].map(dict_糖尿病家族史)
test_df['糖尿病家族史'] = test_df['糖尿病家族史'].map(dict_糖尿病家族史)
train_df['糖尿病家族史'] = train_df['糖尿病家族史'].astype('category')
test_df['糖尿病家族史'] = train_df['糖尿病家族史'].astype('category')
train_df['性别'] = train_df['性别'].astype('category')
test_df['性别'] = train_df['性别'].astype('category')
# 这个转换为类别是有什么特别的含义吗
train_df['年龄'] = 2022 - train_df['出生年份']
test_df['年龄'] = 2022 - test_df['出生年份']
#前面操作过了,可是没什么用
train_df['口服耐糖量测试_diff'] = train_df['口服耐糖量测试'] - train_df.groupby('糖尿病家族史').transform('mean')['口服耐糖量测试']
test_df['口服耐糖量测试_diff'] = test_df['口服耐糖量测试'] - test_df.groupby('糖尿病家族史').transform('mean')['口服耐糖量测试']
#原来这个特征要操作,而不是体重和舒张压
步骤1:安装LightGBM,并学习基础的使用方法;
步骤2:将训练集20%划分为验证集,使用LightGBM完成训练,精度是否有提高?
用coogle老师给的模板代码跑一下
# 模型交叉验证
def run_model_cv(model, kf, X_tr, y, X_te, cate_col=None):
train_pred = np.zeros( (len(X_tr), len(np.unique(y))) )
test_pred = np.zeros( (len(X_te), len(np.unique(y))) )
cv_clf = []
for tr_idx, val_idx in kf.split(X_tr, y):
x_tr = X_tr.iloc[tr_idx]; y_tr = y.iloc[tr_idx]
x_val = X_tr.iloc[val_idx]; y_val = y.iloc[val_idx]
call_back = [
lgb.early_stopping(50),
]
eval_set = [(x_val, y_val)]
model.fit(x_tr, y_tr, eval_set=eval_set, callbacks=call_back, verbose=-1)
cv_clf.append(model)
train_pred[val_idx] = model.predict_proba(x_val)
test_pred += model.predict_proba(X_te)
test_pred /= kf.n_splits
return train_pred, test_pred, cv_clf
```python
clf = lgb.LGBMClassifier(
max_depth=3,
n_estimators=4000,
n_jobs=-1,
verbose=-1,
verbosity=-1,
learning_rate=0.1,
)
train_pred, test_pred, cv_clf = run_model_cv(
clf, KFold(n_splits=5),
train_df.drop(['编号', '患有糖尿病标识'], axis=1),
train_df['患有糖尿病标识'],
test_df.drop(['编号'], axis=1),
)
print((train_pred.argmax(1) == train_df['患有糖尿病标识']).mean())
test_df['label'] = test_pred.argmax(1)
test_df.rename({'编号': 'uuid'}, axis=1)[['uuid', 'label']].to_csv('submit.csv', index=None)
步骤3:将步骤2预测的结果文件提交到比赛,截图分数;
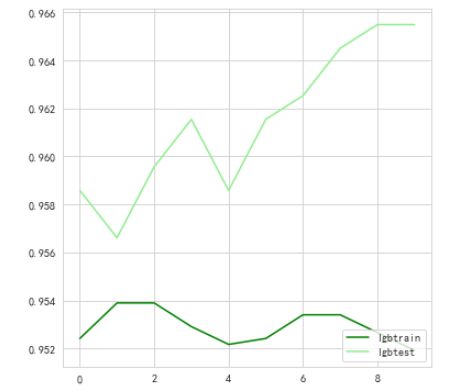
步骤4:尝试调节搜索LightGBM的参数;
看了不同深度的参数的影响
lgbtrain = []
lgbtest = []
for i in range(10):
clf = lgb.LGBMClassifier(
max_depth=i+1,
n_estimators=4000,
n_jobs=-1,
verbose=-1,
verbosity=-1,
learning_rate=0.1,
)
train_pred, test_pred, cv_clf = run_model_cv(
clf, KFold(n_splits=5),
Xtrain,
Ytrain,
Xtest,
)
lgbtrain.append((train_pred.argmax(1) == Ytrain).mean())
lgbtest.append((test_pred.argmax(1) == Ytest).mean())
看了不同学习率的参数的影响
lgbtrain = []
lgbtest = []
# for i in np.linspace(0.39,0.40,19):
for i in np.linspace(0.001,0.1,19):
clf = lgb.LGBMClassifier(
max_depth=2,
n_estimators=4000,
n_jobs=-1,
verbose=-1,
verbosity=-1,
learning_rate=i,
)
train_pred, test_pred, cv_clf = run_model_cv(
clf, KFold(n_splits=5),
Xtrain,
Ytrain,
Xtest,
)
lgbtrain.append((train_pred.argmax(1) == Ytrain).mean())
lgbtest.append((test_pred.argmax(1) == Ytest).mean())
graph = [lgbtrain,lgbtest]
color = ["green","lightgreen"]
label = ["lgbtrain","lgbtest"]
plt.figure(figsize=(6,6))
for i in range(len(graph)):
plt.plot(np.linspace(0.001,0.1,19),graph[i],color[i],label=label[i])
plt.legend(loc=4)
plt.show()
步骤5:将步骤4调参之后的模型从新训练,将最新预测的结果文件提交到比赛,截图分数;
任务7:多折训练与集成
可以参考 大佬的https://blog.csdn.net/qq_39473431/article/details/125478123
还有大佬提及的 https://www.kaggle.com/code/arthurtok/introduction-to-ensembling-stacking-in-python/notebook
步骤1:使用KFold完成数据划分;
KFold划分数据集的原理:根据n_split直接进行划分。已经划分过了
步骤2:使用StratifiedKFold完成数据划分;
StratifiedKFold划分数据集的原理:划分后的训练集和验证集中类别分布尽量和原数据集一样
使用
数据竞赛中的实践
理论
调参
步骤3:使用StratifiedKFold配合LightGBM完成模型的训练和预测
coggle给的范例是KFold的,修改相应代码即可
步骤4:在步骤3训练得到了多少个模型,对测试集多次预测,将最新预测的结果文件提交到比赛,截图分数;
步骤4:使用交叉验证训练5个机器学习模型(svm、lr等),使用stacking完成集成,将最新预测的结果文件提交到比赛,截图分数;
集成
做一个好的模型,首先要有好的数据,数据的质量决定了你模型效果的上限;其次,要做好特征工程,在无法改变数据质量的条件下,特征工程是重中之重;最后,建立模型。
有三种主流的模型思想,一种是Bagging,代表模型是随机森林;一种是Boosting,代表模型是GBDT、Xgboost、lightGBM;还一种是stacking或blending。前2种模型可以调参。
总结
可以像这样
把难点和过程都叙述出来