R语言使用surveyCV包对NHANES数据(复杂调查加权数据)进行10折交叉验证
美国国家健康与营养调查( NHANES, National Health and Nutrition Examination Survey)是一项基于人群的横断面调查,旨在收集有关美国家庭人口健康和营养的信息。
地址为:https://wwwn.cdc.gov/nchs/nhanes/Default.aspx

既往咱们通过多篇文章对复杂加权数据的线性模型、逻辑回归模型、生存分析模型进行了分析。我们在建立数据模型后通常希望在外部数据验证模型的检验能力。然而当没有外部数据可以验证的时候,交叉验证也不失为一种方法。交叉验验证(交叉验证,CV)则是一种评估模型泛化能力的方法,广泛应用中于数证据采挖掘和机器学习领域,在交叉验证通常将数据集分为两部分,一部分为训练集,用于建立预测模型;另一部分为测试集,用于测试该模型的泛化能力。
咱们既往文章《基于R语言进行K折交叉验证》介绍了普通数据交叉验证,今天咱们来介绍一下使用surveyCV包进行复杂加权数据交叉验证,
该包通过在创建 CV 折叠以及计算测试集损失估计时考虑分层、聚类、FPC 的调查权重MSE(均方误差),对复杂的调查数据实现交叉验证 (CV)。模型,或逻辑模型的二元交叉熵)。
咱们先导入R包和数据
library(surveyCV)
library(survey)
library(ISLR)
data("api")

这次使用survey自带的加州学生的数据,包含有学生的成绩和其他数据。这个数据集带有6个数据,咱们使用的是apistrat数据
假设咱们想了解api00和ell线性关系,nfolds代表你想用多少折,其他都是一些调查函数的参数。
咱们先写出它的函数,这是一个默认线性函数
a<-"api00~ell"
cv.svy(apistrat, a,
nfolds = 10, strataID = "stype", weightsID = "pw", fpcID = "fpc")
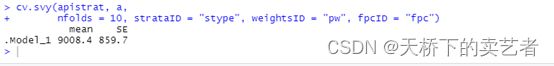
这样结果就出来了,这里的mean相当于MSE的平均值,表示误差的平均值,它可以有助于改善我们的模型,它和单用svymean函数这种算法是完全不一样的
如果咱们想了解多个模型
cv.svy(apistrat, c("api00~ell",
"api00~ell+meals",
"api00~ell+meals+mobility"),
nfolds = 10, strataID = "stype", weightsID = "pw", fpcID = "fpc")
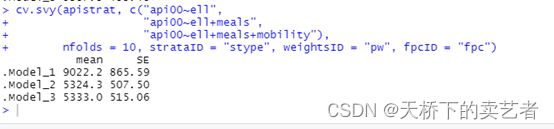
这样就轻松出结果了,非常方便好用。我们可以看到添加协变量以后,MSE出现明显变化,变小了,表明添加协变量有助于改善MSE。
如果我们想指定集群而不是分层,更改一下clusterID这个变量,也非常方便
cv.svy(apiclus1, c("api00~ell",
"api00~ell+meals",
"api00~ell+meals+mobility"),
nfolds = 10, clusterID = "dnum", weightsID = "pw", fpcID = "fpc")

如果咱们是有调查函数的,咱们需要用到cv.svydesign这个函数,指定一下就可以了
dstrat <- svydesign(id = ~1, strata = ~stype, weights = ~pw, data = apistrat, fpc = ~fpc)
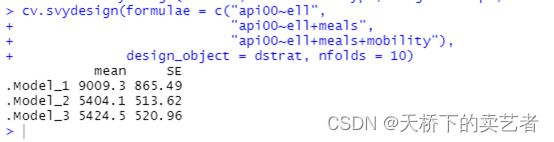
cv.svydesign(formulae = c("api00~ell",
"api00~ell+meals",
"api00~ell+meals+mobility"),
design_object = dstrat, nfolds = 10)
如果是已经生成了svyglm模型的,咱们需要使用cv.svyglm这个函数指定
glmstrat <- svyglm(api00 ~ ell+meals+mobility, design = dstrat)
cv.svyglm(glmstrat, nfolds = 10)
library(splines)
NSFG.svydes <- svydesign(id = ~SECU, strata = ~strata, nest = TRUE,
weights = ~wgt, data = NSFG_data)
生成结果
NSFG.svyglm.logistic <- svyglm(LBW ~ ns(age, df = 3), design = NSFG.svydes,
family = quasibinomial())
cv.svyglm(glm_object = NSFG.svyglm.logistic, nfolds = 4)

