如何找出最优的【SVC】核函数和参数值—以乳腺癌数据集为例
在实际的工作中,有的时候我们不知道数据特征,也不知道我们的数据是线性还是非线性。因此我们需要对数据一步步进行摸索,来找到最优的核函数和参数值。接下来我们以sklearn乳腺癌数据集为例。
先来导入相应的模块:
from sklearn.datasets import load_breast_cancer
from sklearn.svm import SVC
from sklearn.model_selection import train_test_split
import matplotlib.pyplot as plt
import numpy as np
from time import time
import datetime导入数据集,并将特征矩阵和标签赋值给X和Y:
data = load_breast_cancer()
X = data.data
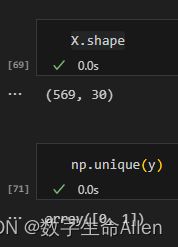
y = data.target可以看到数据集有569个样本,30个特征,2种标签。我们先来选取前两列特征,画出散点图看看效果:
plt.scatter(X[:,0],X[:,1],c=y)
plt.show()我们用PCA降维,保留数据的两个特征:
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
data_pca = pca.fit_transform(x)
data_pca.shape画图查看效果:
plt.scatter(data_pca[:,0],data_pca[:,1],c=y)
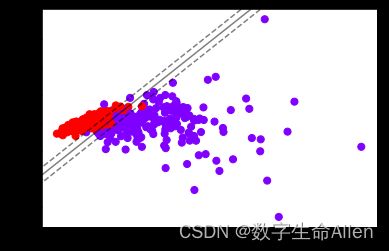
plt.show()此时我们使用SVC看看图像:
def plot_svc_decision_function(model,ax=None):
if ax is None:
ax = plt.gca()
xlim = ax.get_xlim()
ylim = ax.get_ylim()
x = np.linspace(xlim[0],xlim[1],30)
y = np.linspace(ylim[0],ylim[1],30)
Y,X = np.meshgrid(y,x)
xy = np.vstack([X.ravel(), Y.ravel()]).T
P = model.decision_function(xy).reshape(X.shape)
ax.contour(X, Y, P,colors="k",levels=[-1,0,1],alpha=0.5,linestyles=["--","-","--"])
ax.set_xlim(xlim)
ax.set_ylim(ylim)
plt.scatter(data_pca[:,0],data_pca[:,1],c=y,s=50,cmap="rainbow") # 画散点图
clf = SVC(kernel = "linear").fit(data_pca,y)
plot_svc_decision_function(clf)接下来我们将数据集分隔为训练集和测试集,并看看另外三个核函数的准确率,并使用时间戳函数计算每个函数的运行时长:
x_train,x_test,y_train,y_test = train_test_split(data_pca,y,test_size=0.3,random_state=420)
kernel = ['linear','poly','rbf','sigmoid']
for i in kernel:
time0 = time()
clf = SVC(kernel=i
,gamma="auto"
,degree=1
,cache_size=5000 #缓存大小,以MB为单位,默认为200
).fit(x_train,y_train)
print("The accuracy under kernel %s is %f" % (i,clf.score(x_test,y_test)))
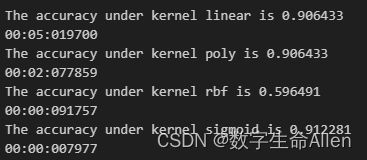
print(datetime.datetime.fromtimestamp(time()-time0).strftime("%M:%S:%f"))输出结果如下:
从输出结果来看,rbf核函数显然不能用。运行时间中,线性核函数运行时间最长。在这里我们要重新强调一个概念,在机器学习和数据分析中,量纲的概念非常重要。因为不同的特征可能有不同的量纲,如果直接使用这些特征进行计算,可能会导致一些问题。例如,一个特征的范围是1到10,另一个特征的范围是1到10000,那么在计算距离或者相似度时,范围大的特征可能会主导结果,而忽略了范围小的特征。为了解决这个问题,我们通常会进行特征缩放,使得所有的特征都在同一量纲上,或者说有相同的尺度。常见的特征缩放方法有标准化等。
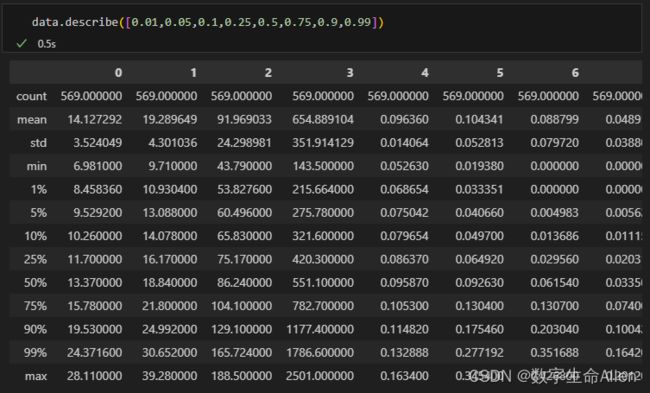
现在我们把X放在我们的dataframe里面,用describe()函数看看描述性统计的结果:
import pandas as pd
data = pd.DataFrame(X)
data.describe([0.01,0.05,0.1,0.25,0.5,0.75,0.9,0.99]).T#描述性统计通过观察数据我们可以发现,平均值有的仅有0.04,有的高达654,说明存在严重的量纲不统一问题。我们再来看看数据的分布,我们通过从1%的数据和最小值相对比,90%的数据和最大值相对比,查看是否是正态分布或偏态分布,如果差的太多就是偏态分布,谁大方向就偏向谁。可以发现数据大的特征存在偏态问题,这个时候就需要对数据进行标准化。
from sklearn.preprocessing import StandardScaler
X = StandardScaler().fit_transform(X)#将数据转化为0,1正态分布
data = pd.DataFrame(X)
data.describe([0.01,0.05,0.1,0.25,0.5,0.75,0.9,0.99]).T#均值很接近,方差为1了我们将标准化后的数据去训练模型,再去计算模型的准确率和运行时间:
Xtrain, Xtest, Ytrain, Ytest = train_test_split(X,y,test_size=0.3,random_state=420)
Kernel = ["linear","poly","rbf","sigmoid"]
for kernel in Kernel:
time0 = time()
clf= SVC(kernel = kernel
, gamma="auto"
, degree = 1
, cache_size=5000
).fit(Xtrain,Ytrain)
print("The accuracy under kernel %s is %f" % (kernel,clf.score(Xtest,Ytest)))
print(time()-time0)可以发现四个模型的分数都有大幅度的提高!而且运行时间也可以大幅缩短!这说明标准化可以有效的提升分类器的效果。因此,SVM执行之前,非常推荐先进行数据的无量纲化!到 了这一步,我们是否已经完成建模了呢?虽然线性核函数的效果是最好的,但它是没有核函数相关参数可以调整的,rbf和多项式却还有着可以调整的相关参数,接下来我们就来看看这些参数。
从核函数的公式来看,我们其实很难去界定具体每个参数如何影响了SVM的表现。当gamma的符号变化,或者 degree的大小变化时,核函数本身甚至都不是永远单调的。所以如果我们想要彻底地理解这三个参数,我们要先推 导出它们如何影响核函数地变化,再找出核函数的变化如何影响了我们的预测函数(可能改变我们的核变化所在的 维度),再判断出决策边界随着预测函数的改变发生了怎样的变化。无论是从数学的角度来说还是从实践的角度来 说,这个过程太复杂也太低效。所以,我们往往避免去真正探究这些参数如何影响了我们的核函数,而直接使用学 习曲线或者网格搜索来帮助我们查找最佳的参数组合。
接下来我们先来画gamma的学习曲线:
score = []
gamma_range = np.logspace(-10, 1, 50) #返回在对数刻度上均匀间隔的数字
for i in gamma_range:
clf = SVC(kernel="rbf",gamma = i,cache_size=5000).fit(Xtrain,Ytrain)
score.append(clf.score(Xtest,Ytest))
print(max(score), gamma_range[score.index(max(score))])
plt.plot(gamma_range,score)
plt.show()输出结果为:0.9766081871345029 0.012067926406393264
说明把gamma设置成0.012时,准确率最高可达0.9766。
接下来我们来调整poly核函数的参数:gamma和coef。在这里我们用交叉验证和网格搜索,先导入相应的模块:
from sklearn.model_selection import StratifiedShuffleSplit#用来实例化交叉验证
from sklearn.model_selection import GridSearchCV#带交叉验证的网格搜索再来确定我们的参数范围:
gamma_range = np.logspace(-10,1,20)
coef0_range = np.linspace(0,5,10)把参数放在字典里面:
param_grid = dict(gamma = gamma_range
,coef0 = coef0_range)实例化一个交叉验证对象:
cv = StratifiedShuffleSplit(n_splits=5, test_size=0.3, random_state=420)#将数据分为5份,5份数据中测试集占30%实例化一个网格搜索对象:
grid = GridSearchCV(SVC(kernel = "poly",degree=1,cache_size=5000
,param_grid=param_grid
,cv=cv))训练数据:
grid.fit(X, y)输出最佳的参数组合,准确率和时间戳:
print("The best parameters are %s with a score of %0.5f" % (grid.best_params_,
grid.best_score_))
print(time()-time0)输出结果为: