炸裂!Sklearn 的 10 个宝藏级使用方法!
大家好,本次给大家介绍10个Sklearn方法,比较小众但非常好用。
1️.FunctionTransformer
虽然Sklearn中有很多内置的预处理操作可以放进pipeline管道,但很多时候并不能满足我们的需求。
如果是比较简单并且通过一个函数可以实现需求的情况,我们可以将函数通过FunctionTransformer进行包装生成可与Sklearn兼容的转换器,然后装进pipeline。
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import FunctionTransformer
def reduce_memory(X: pd.DataFrame, y=None):
"""将数值型列的类型转换为float32类型,以减少内存占用
"""
num_cols = X.select_dtypes(incluce=np.number).columns
for col in num_cols:
X[col] = X.astype("float32")
return X, y
ReduceMemoryTransformer = FunctionTransformer(reduce_memory)
# 装进一个pipeline
>>> make_pipeline(SimpleImputer(), ReduceMemoryTransformer)
Pipeline(steps=[('simpleimputer', SimpleImputer()),
('functiontransformer', ReduceMemoryTransformer()])
技术交流
技术要学会分享、交流,不建议闭门造车。一个人可以走的很快、一堆人可以走的更远。
本文由粉丝群小伙伴总结与分享,如果你也想学习交流,资料获取,均可加交流群获取,群友已超过2000人,添加时最好的备注方式为:来源+兴趣方向,方便找到志同道合的朋友。
方式①、添加微信号:dkl88194,备注:来自CSDN + 加群
方式②、微信搜索公众号:Python学习与数据挖掘,后台回复:加群
2.自定义transformers
但有更复杂需求的时候,可能一个简单函数也是无法完成功能实现的,这时就需要自己实实在在地创建一个转换器了。
比如,数据清洗中比较常见的操作缩放特征变量并使其呈正态分布。通常我们会使用对数变换器如PowerTransformer或np.log,但默认的方法会有一点问题,即如果某个特征包含零值,那么底层的对数函数无法处理会提出报错。
因此,一般的应对方法是将特征向量加上1,然后再执行转换,以避免报错。如果想要还原原始向量,直接调用指数函数然后再减去1,就可以了。非常的丝滑。
当然,这个操作并未内置于Sklearn中,并且也不是一个简单函数能搞定的。下面看看如何自定义一个转换器解决这个问题。
from sklearn.base import BaseEstimator, TransformerMixin
from sklearn.preprocessing import PowerTransformer
class CustomLogTransformer(BaseEstimator, TransformerMixin):
def __init__(self):
self._estimator = PowerTransformer() # 初始化一个转换器
def fit(self, X, y=None):
X_copy = np.copy(X) + 1 # 防止零值出现报错,进行加一操作
self._estimator.fit(X_copy)
return self
def transform(self, X):
X_copy = np.copy(X) + 1
return self._estimator.transform(X_copy) # 执行转换
def inverse_transform(self, X):
X_reversed = self._estimator.inverse_transform(np.copy(X))
return X_reversed - 1 # 指数函数后减一
上面创建了一个类,继承了BaseEstimator并使其TransformerMixin能够插入pipeline管道的类。
3.TransformedTargetRegressor
有些时候,不仅仅是特征X需要处理,目标变量y也需要预处理操作。一个典型的场景就是我们上面提到的缩放数据使其呈现正态分布。通常我们会在pipeline以外做额外的处理,但 Sklearn 有一个方法可以同时在管道中处理。
TransformedTargetRegressor是一个专门针对regressor回归器进行转换的类,通过它可以同时将特征X和目标变量y在管道pipeline中做处理。比如下面的lgb回归的例子,它使用CustomLogTransformer对目标y进行对数缩放,然后拟合回归模型。
from sklearn.compose import TransformedTargetRegressor
reg_lgbm = lgbm.LGBMRegressor()
final_estimator = TransformedTargetRegressor(
regressor=reg_lgbm, transformer=CustomLogTransformer()
)
final_estimator.fit(X_train, y_train)
TransformedTargetRegressor(regressor=LGBMRegressor(),
transformer=CustomLogTransformer())
如果转换器是一个函数如np.log,可以将其传递给func参数。
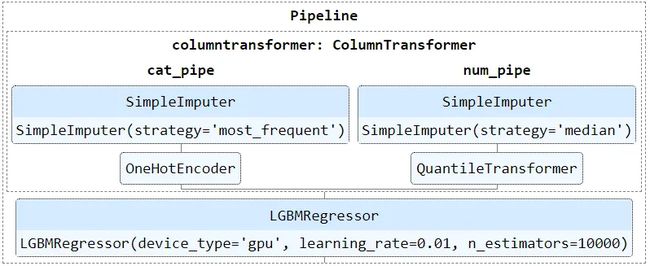
4.管道流程图
如果管道由多个步骤或子管道组成,代码上可能会比较混乱。Sklearn提供了估计器的HTML表示形式,让整理处理流程更直观清晰:
>>> giant_pipeline
Pipeline(steps=[('columntransformer',
ColumnTransformer(transformers=[('cat_pipe',
Pipeline(steps=[('impute',
SimpleImputer(strategy='most_frequent')),
('oh',
OneHotEncoder())]),
<sklearn.compose._column_transformer.make_column_selector object at 0x000001B6D8BD9310>),
('num_pipe',
Pipeline(steps=[('impute',
SimpleImputer(strategy='median')),
('transform',
QuantileTransformer())]),
<sklearn.compose._column_transformer.make_column_selector object at 0x000001B6D8BD9160>)])),
('lgbmregressor',
LGBMRegressor(device_type='gpu', learning_rate=0.01,
n_estimators=10000))])
from sklearn import set_config
set_config(display="diagram")
>>> giant_pipeline
将dispaly参数设置为diagram,就可以获得管道的HTML的交互式形式。
5.QuadraticDiscriminantAnalysis
QDA为QuadraticDiscriminantAnalysis的简称,是二次判别分析的意思。在Kaggle竞赛中,即使没有超参数调整,二次判别分析分类器也可以获得AUC0.965这样高的分数,超过了大多数树模型,包括XGBoost和LightGBM。
那为什么之前很少听说过该算法呢?因为它的使用有严格的限制条件,它要求训练特征严格的正态分布,这样QDA就可以轻松计算并拟合分布周围的椭球形状了。
QDA 的另一个优点是它的速度非常快,在百万行数据集上训练它只需要几秒钟。以下是QDA在Sklearn中的执行速度。
%%time
from sklearn.discriminant_analysis import QuadraticDiscriminantAnalysis
# Generate 1M samples and 100 features
X, y = make_classification(n_samples=1000000, n_features=100)
qda = QuadraticDiscriminantAnalysis().fit(X, y)
Wall time: 13.4 s
6.Voting Classifier/Regressor
在模型训练中,我们经常会遇到几个模型效果相似的情况,想要进一步提升效果,这时可以使用投票方法,是一种简单的模型集成方法。
投票方法效果提升原因在于概率论,简单来说就是少数服从多数。具体的就是,投票分类器会将多个分类器的多数票作为最终预测,而如果类别是概率或预测是连续的,则对预测进行平均。
Sklearn提供了两个方法VotingClassifier和VotingRegressor,我们只需要传递一个分类器或回归器的列表,将它们组合起来就可以了。下面是VotingClassifier的示例。
from sklearn.ensemble import VotingClassifier
X, y = make_classification(n_samples=1000)
ensemble = VotingClassifier(
estimators=[
("xgb", xgb.XGBClassifier(eval_metric="auc")),
("lgbm", lgbm.LGBMClassifier()),
("cb", cb.CatBoostClassifier(verbose=False)),
],
voting="soft",
# n_jobs=-1,
)
_ = ensemble.fit(X, y)
以上设置voting参数为soft,代表预测是概率。此外,还可以为不同的模型分配weights权重系数进行更精准的预测。
7.Stacking Classifier/Regressor
另一种比投票更强大的集成方法是stacking。
stacking背后的思想是,子模型应该尽可能多样化,因为不同的模型从不同的角度学习训练集的信息,可以覆盖整个信息空间。
换句话说,各种模型(如树、线性模型、表面拟合器、近邻模型、贝叶斯模型和高斯模型)最大化了训练潜力,它们的组合输出减少了偏差并防止了过拟合。
Kaggle竞赛中,stacking是一个提分的神器,很多获胜方案中都有提及。示例代码如下。
from sklearn.ensemble import StackingClassifier, StackingRegressor
from sklearn.linear_model import LogisticRegression
X, y = make_classification(n_samples=1000)
ensemble = StackingClassifier(
estimators=[
("xgb", xgb.XGBClassifier(eval_metric="auc")),
("lgbm", lgbm.LGBMClassifier()),
("cb", cb.CatBoostClassifier(verbose=False)),
],
final_estimator=LogisticRegression(),
cv=5,
passthrough=False
# n_jobs=-1,
)
_ = ensemble.fit(X, y)
8.LocalOutlierFactor
异常值会使模型的目标函数产生偏差,可能导致过于乐观或过于悲观的结果。
对于小数据集来说,查找异常值不成问题。如果特征超过50-100个时,就需要一种快速准确的算法来检测高维异常值了。
对于具有数百个特征和数百万行的数据集,原始算法可能也需要运行几个小时。这时可以将降维算法与异常值检测方法结合起来,一个比较好的组合是UMAP和LocalOutlierFactor。LocalOutlierFactor是一种基于近邻的算法,旨在快速处理大型数据集。
%%time
import umap # pip install umap
from sklearn.neighbors import LocalOutlierFactor
X, y = make_classification(n_samples=5000, n_classes=2, n_features=10)
X_reduced = umap.UMAP(n_components=2).fit_transform(X, y)
lof = LocalOutlierFactor()
labels = lof.fit_predict(X_reduced, y)
Wall time: 17.8 s
>>> np.where(labels == -1)
(array([ 119, 155, 303, 331, 333, 407, 418, 549, 599, 664, 795,
3092, 3262, 3271, 3280, 3289, 3311, 3477, 3899, 3929, 3975, 4301,
4358, 4442, 4522, 4561, 4621, 4631, 4989], dtype=int64),)
9️.QuantileTransformer
某些时候得到的模型结果分布非常不规则,可能通过对数转换器或缩放器都无法强制转换为正态分布,比如双峰、三峰、或者n峰的分布。
这种情况下可以使用QuantileTransformer,它使用分位数的统计指标实现中心化和缩放分布。
import pandas as pd
from sklearn.preprocessing import QuantileTransformer
qt = QuantileTransformer().fit(crazy_distributions)
crazy_feature_names = ["f18", "f31", "f61"]
crazy_distributions = pd.DataFrame(qt.transform(crazy_distributions), columns=crazy_feature_names)
fig, axes = plt.subplots(1, 3, figsize=(20, 6))
for ax, f_name in zip(axes.flatten(), crazy_feature_names):
sns.kdeplot(crazy_distributions[f_name], ax=ax, color="#E50914")
PCA + tSNE/UMAP
这个一个降维的组合使用方法。因为PCA主成分分析对于高维度处理速度是比较快的,因此通常作为第一阶段的处理方法,比如使用PCA缩减到30-50的维度,然后再用其他算法如tSNE或UMAP作为第二阶段的处理方法。
下面是 PCA 和 tSNE 的组合:
from sklearn.decomposition import PCA
from sklearn.manifold import TSNE
df = dt.fread("data/large.csv").to_pandas()
>>> df.shape
(1000000, 287)
X, y = df.drop("target", axis=1), df[["target"]].values.flatten()
%%time
manifold_pipe = make_pipeline(QuantileTransformer(), PCA(n_components=30), TSNE())
reduced_X = manifold_pipe.fit_transform(X, y)
------------------------------------------
Wall time: 4h 27min 46s
以上在100万行和约300个特征的数据集上,先通过PCA投影到前30个维度,然后再投影到二维,整个过程需要4.5小时,并且结果也不是很好。
>>> plt.scatter(reduced_X[:, 0], reduced_X[:, 1], c=y, s=0.05);
因此建议使用UMAP,它比tSNE快得多,并且可以更好地保留了数据的局部结构。
%%time
manifold_pipe = make_pipeline(QuantileTransformer(), PCA(n_components=30))
X_pca = manifold_pipe.fit_transform(X, y)
embedding = umap.UMAP(n_components=2).fit(X_pca, y)
Wall time: 14min 27s
>>> plt.scatter(embedding.embedding_[:, 0], embedding.embedding_[:, 1], c=y, s=0.05);
UMAP设法找到目标类别之间的明显区别,并且速度比tSNE快了20倍。
以上是全部内容,点个赞交个朋友。