深度学习医学图像语义分割实战(一)
1.什么是图像语义分割segementation
一般是只对图像整体做分类,那么如果是将图像的目标提取出来,这就是语义分割。与分类不同的是,语义分割需要判断每个像素点的类别,进行精确分割,产生目标的掩码,图像的语义分割是像素级别的。
2.如何对每个像素点进行分类
语义分割最经典网络--FCN,常规的图像分类网络是最后展成全连接层,是一维输出,而FCN则可以将全连接层换成卷积,这样就可以得到一张二维的featuremap,后结softmax获得每个像素的分类信息,从而解决分类信息。
FCN(Fully Convolutional NetWorks)网络,全卷积神经网络 卷积,反卷积
图像输入经过卷积池化,卷积池化,得到特征图,在经过上采样还原成原始图像大小,进行对每个像素点预测。使用多层特征融合有利于提高分割准确性。
语义分割都是编码解码框架的。下采样(编码,提取信息,信息浓缩),上采样(解码,返还信息)。
VGG,ResNet,MoblieNet,inception,DenseNet都可以用来做下采样,backbone,提取特征的主干网络。选取不同的主干网络,但是预测结果可能不同。通常用的就是VGG,ResNet。
上采样还原图像大小,方法有:
插值法(FCN就是使用双线性插值变为更大的图像,有点,速度快)
反卷积(UNet使用的是反卷积,先填充周边补零,再进行卷积,速度慢,效果好)
反池化(池化的反操作,用的不是特别多,在池化时记录下对应kernel的坐标,在反池化时根据kernel进行方法,根据之前的坐标将元素填写进去,其他位置补零,这是反最大池化,当然还有反平均池化)
语义分割评价指标,mIoU,均交并比,每个类别的, 交并比 IOU交集/并集。
MIoU = A∩B / A∪B = TP / (FP + FN + TP)
应用背景与目标分别,工业零件缺陷检测,医学图像分割(检测病灶)
经典的网络,U-net,PSPNet,DeepLab等都是经典的算法。
3.Unet实现颅脑MRI分割项目
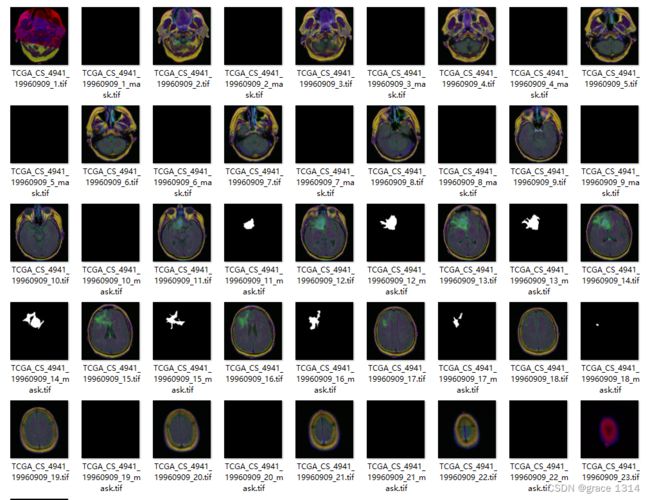
(1)颅脑MRI数据集
Brain MRI segmentation | Kaggle
该数据来自Kaggle, 每个文件夹下有多副图像。
其中对于每副颅脑MRI图像包含对应的一幅mask图像,该mask图像包含颅脑MRI图像的颅脑位置。图像为tif格式,大小为256x256。
(2)医学分割模型Unet
主要分类两部分,分为编码器和解码器,原文论文使用VGG作为backbone,输入图片很大,卷积,池化,卷积,池化,经过5层卷积下采样,代码相同,可以得到特征值,后面进行反卷积操作,对扩大图像,这里再每次反卷积时会对与之前的卷积特征进行特征融合。经过这些操作就会得到最终特征图。一般会进行加深网络,
医学图像语义结构较为固定,都是一个固定的器官成像,而不是一个全身的。
Unet网络参数小,轻型,使用很少的训练图像就得到精确的结果。
应用,病理图片,MRI等。
4.Brain MRI数据处理
首先,还是新建项目,建立data文件目录,将下载好的数据保存至data下。如果觉得麻烦可以点击下面链接直接下载数据集,注意:该数据集已经经过调整。
https://download.csdn.net/download/qq_43616651/88524895
然后再项目文件下创建.py文件,这里我们开始数据处理。
import os
import numpy as np
import glob
import cv2
# 数据目录
kaggle_3m = "./data/kaggle_3m/"
# 循环遍历目录中的文件,将文件名放进一个列表
# 通过glob.glob抓取
# 加*号是将kaggle_3m中的内容全部保存下来,不论是什么文件
dirs = glob.glob(kaggle_3m+'*')
#print(dirs)
#dirs现在是kaggle_3m里的文件路径,还不是我们要拿的数据
#接下来循环遍历每个文件,将每个文件中的图片名拿下来
# 一个存储img,一个存储mask或者叫label。
data_img = []
data_label = []
for subdir in dirs:
dirname = subdir.split('\\')[-1] #将文件路径分开,只拿后面的文件名
#print(dirname)
for filename in os.listdir(subdir):
img_path = subdir+"/"+filename #整个kaggle目录下里面的图像文件绝对路径
#print(img_path)
#存储img,与mask,后缀中有mask的存到label中,没有的存在img中
if 'mask' in img_path:
data_label.append(img_path)
else:
data_img.append(img_path)
#print(data_label[11])
#print(data_img[13]) #测试
#print(len(data_label))
#print(len(data_img)) #测试岔开数据是否对等,若长度不等,则需要调整
#调整文件:
data_imgx = []
for i in range(len(data_label)): #从少的数据进行抓取,data_label
img_mask = data_label[i]
#img文件与mask文件的差距就在文件名后有_mask
img = img_mask[:-9]+'.tif'
#print(img)
data_imgx.append(img)
#展示前5个,看是否一一对应
#print(data_imgx[10:15])
#print(data_label[10:15])
#数据中有些mask什么都没有,是负样本,而我们训练网络是用来做分割的,需要mask有内容
#所以这里将mask有内容的提取出来
data_new_img = []
data_new_label = []
#img = cv2.imread(data_label[0]) #cv2.imread 读进来是np格式
#print(img)
for i in data_label:
value = np.max(cv2.imread(i))
# print(value)
# 对mask数据进行判断,因为mask是二值的,如果有白色的则证明mask有病灶
#使用opencv读取图片,取最大值,如果是255则证明有,0则没有
try: #try 防止出现异常,一般不会
if value>0:
data_new_label.append(i)
i_img = i[:-9]+'.tif'
data_new_img.append(i_img)
except:
pass
#print(data_new_img[:5])
#print(data_new_label[:5]) #产看一一对应情况
这里先看坐标的data目录下,数据都存在与kaggle_3m文件下,且并没有进行分类,数据现在存在TCGA的这些文件下。我们首先还是从kaggle_3m下爬取每个TCGA文件中的文件名,并且将mask与img分开。
再data_img,与data_label中我们拿到的只是文件的路径地址,后面通过调用该地址即可访问图像与mask。
在获得所有文件的索引地址后,现在主要的还是看mask与img的数量是否相等,进行调整,利用mask与img文件名的差异进行调整,将多余的mask或者img去除。这里是先打印了label和img的长度,看到二者label是数量少的一个,所以按照label进行筛选img文件。
将新的图像文件地址保存好后,现在多要考虑的就是我们的项目是图像分割,对于负样本,即没有mask数据的图像也是要剔除的,当然也可以不剔除,这里为了提高后期分割的效果,裁取剔除分样本的做法。
负样本即像上面这种没有病灶位置的mask数据。
这里采用的剔除方法即就是使用opencv读取mask图像,取最大值,如果最大值是255,则保留,如果不是则证明没有病灶,剔除。
最后打印保留新的img与label数据的大小,只剩1319张了。
5.制作数据集
现在已经从文件读取适合当数据集的数据,接下来就是制作数据集,将其分为train,与test,train为1000张,剩下的作为test。
#先定义数据集的transfomer,train,test,先不进行图像增强
train_trasformer = transforms.Compose([
transforms.Resize((256,256)),
transforms.ToTensor()
])
test_trasformer = transforms.Compose([
transforms.Resize((256,256)),
transforms.ToTensor()
])
#自定义dataset类
class BrainMRIdataSet(Dataset):
def __init__(self,img,mask,transformer):
self.img = img
self.mask = mask
self.transformer = transformer
def __getitem__(self, index):
img = self.img[index]
mask = self.mask[index]
img_open = Image.open(img) #读取图片
img_tensor= self.transformer(img_open)#数据增强
mask_open = Image.open(mask)
mask_tensor = self.transformer(mask_open)
#这里mask_tensor经过transformer变化变为tensor型张量,数据为浮点数
#unet本质是分类问题,这里需要将mask的像素值转成整型
#比如mask的像素值有0和255两种,就是二分类
#此外输入mask的大小为256x256x1,需要将mask中最后一个通道数消除
#变成一个二维数据没这使unet的mask输入格式,torch.long就是数据类型
mask_tensor = torch.squeeze(mask_tensor).type(torch.long)
return img_tensor,mask_tensor
def __len__(self):
return len(self.img)
#筛选的数据长度
#print(len(data_new_img))
#print(len(data_new_label))
#这里使1319
#划分数据集和测试集,前1000作为数据集,后319最为训练集
s = 1000
train_img = data_new_img[:s]
train_label = data_new_label[:s]
test_img = data_new_img[s:]
test_label = data_new_label[s:]
train_data = BrainMRIdataSet(train_img,train_label,train_trasformer)
test_data = BrainMRIdataSet(test_img,test_label,test_trasformer)
dl_train = DataLoader(train_data,batch_size=8,shuffle=True)
dl_test = DataLoader(test_data,batch_size=8,shuffle=True)
#检查数据
img,label = next(iter(dl_train))
plt.figure(figsize=(12,8))
for i,(img,label) in enumerate(zip(img[:4],label[:4])):
#zip 打包为元组
img =img.permute(1,2,0).numpy()
label = label.numpy()
plt.subplot(2,4,i+1)
plt.imshow(img)
plt.subplot(2,4,i+5)
plt.imshow(label)
plt.show()对于数据集首先还是图像增强,即自定义transformer,这里不需要对数据做多大变换,因为MRI图像都是固定的角度,也不需要旋转裁剪改变颜色之类的,能做的就是对图像做resize保证图像大小一致,最后变成tensor型变量。这样test与train的transformer就好了。
下面自定义dataset类,继承torch中的Dataset类。
分别对构造函数,索引函数,以及最后长度进行定义。这里需要主义的一点就是数据集输入的是img与mask,一起进行数据增强,但是我们的mask是用来分割的,这里mask的值只能是0和255两种整形,所以这里需要转成整型long,详细可以看代码注释。至此自定义类写好了。
接下来就是对上节得到的数据集进行分割,前1000张为test,剩余为test,然后将其使用自定义dataset类,转为traindata,以及testdata,最后再使用torch中的数据加载器dataloader。这里batch_size选作8,shuffle是洗牌的意思,即对数据是否打乱。
后面就是对数据集的展示。
一一对应。至此数据集准备好了。下面就是建立Unet网络模型。
6.建立Unet网络模型
"""
Unet结构:下采样编码,上采样编码
encoder,decouder,concat
输入 卷积 池化 卷积池化 卷积 上卷积 卷积 上卷积 1X1卷积 输出 分割map
反卷积,将图像的长和宽还原为原来的2倍,再加上卷积得到的feature map
密集链接,相当于resnet的短接操作
unet 特点
1.医学图像结构固定,语义信息没有特别丰富,
所以高级语义信息和低级特征都显得很重要,
2.u型结构可以在很小的数据集中,获得很好的训练效果
google deeplab v3 模型太大,不太适合轻量化学习
3.添加上采样阶段,并添加了很多特征通道,允许更多的原图像纹理
的信息在高分辨率layers中进行传播。
在肝脏,病理图像分割
"""
import os
import math
import numpy as np
import glob
import pandas as pd
import matplotlib.pyplot as plt
from PIL import Image
import random
import time
import cv2
import torch
import torch.nn as nn
from torch.utils.data import Dataset,DataLoader
from torchvision import transforms
# 下采样,encoder 2层卷积+relu+maxpooling
class Encoder(nn.Module):
def __init__(self,in_channel,out_channel):
super(Encoder,self).__init__()
#两层卷积
self.conv_relu = nn.Sequential(
nn.Conv2d(in_channel,out_channel,kernel_size=3,padding=1),
#输入通道数,输出通道数,卷积核大小,填充
nn.ReLU(inplace=True), #inplace 将原来的tensor,替换为计算完后的tensor,减少资源消耗
nn.Conv2d(out_channel, out_channel, kernel_size=3, padding=1),
#在第二层卷积时输入通道数与输出通道数相等,等于第一层输出
nn.ReLU(inplace=True)
)
self.maxpool = nn.MaxPool2d(kernel_size=2,stride=2)
#maxPooling 层,运行完后,h,w减少一半
#在unet的谷底结构不需要做maxpooling,所以需要一个参数控制
def forward(self,x,if_pool = True):
if if_pool:
x = self.maxpool(x)
x = self.conv_relu(x)
return x
#decoder
class Decoder(nn.Module):
def __init__(self,channels):
super(Decoder,self).__init__()
# 两层卷积
self.conv_relu = nn.Sequential(
nn.Conv2d(2*channels, channels, kernel_size=3, padding=1),
#解码层卷积输入由两部分,一部分是网络传递过来的,一部分是与之前卷积的map
#两者结合,通道数变为2倍,输出通道减半
nn.ReLU(inplace=True),
nn.Conv2d(channels, channels, kernel_size=3, padding=1),
#第二层卷积输出与输入通道相等
nn.ReLU(inplace=True)
)
self.upconv_relu = nn.Sequential(
nn.ConvTranspose2d(channels,channels//2,kernel_size=3,stride=2,padding=1,output_padding=1),
#反卷积后得到的map还需添加一圈,即out_padding,才能使图像长宽刚好变为原来的2倍
nn.ReLU(inplace=True),
)
def forward(self,x):
x = self.conv_relu(x) #先两层卷积,再一个反卷积
x = self.upconv_relu(x)
return x
#前向传播
#组装 unet
#encoder 是先池化,后卷积,是否池化可控制
#decoder 卷积 反卷积
class Unet(nn.Module):
def __init__(self):
super(Unet,self).__init__()
self.encode1 = Encoder(3,64)
self.encode2 = Encoder(64, 128)
self.encode3 = Encoder(128, 256)
self.encode4 = Encoder(256, 512)
self.encode5 = Encoder(512, 1024)
#手动加一层反卷积
self.upconv_relu = nn.Sequential(
nn.ConvTranspose2d(1024,512,kernel_size=3,stride=2,padding=1,output_padding=1),
nn.ReLU(inplace=True)
)
self.decode1 = Decoder(512)
self.decode2 = Decoder(256)
self.decode3 = Decoder(128)
#最后两层卷积
self.convDouble = nn.Sequential(
nn.Conv2d(128,64,kernel_size=3,padding=1),
nn.ReLU(inplace=True),
nn.Conv2d(64,64,kernel_size=3,padding=1),
nn.ReLU(inplace=True),
)
#加1x1卷积,由于本项目只有二分类,所以输出通道为2,如果是多分类,可以自己改
self.last = nn.Conv2d(64,2,kernel_size=1)
def forward(self,x):
x1 = self.encode1(x, if_pool=False)
x2 = self.encode2(x1)
x3 = self.encode3(x2)
x4 = self.encode4(x3)
x5 = self.encode5(x4)
#手动反卷积
x5 = self.upconv_relu(x5)
#拼接变成输入
x6 = torch.cat([x4,x5],dim=1) #第一维是batch,第2维是通道,后面是h,w x6输出
#接下来到decoder
x6 = self.decode1(x6)
x7 = torch.cat([x3, x6], dim=1) #x7输入
x7 = self.decode2(x7)
x8 = torch.cat([x2, x7], dim=1) #x8输入
x8 = self.decode3(x8)
x9 = torch.cat([x1, x8], dim=1) #最后输入
#然后是后面两层卷积加1*1卷积
x9 = self.convDouble(x9)
x10 = self.last(x9)
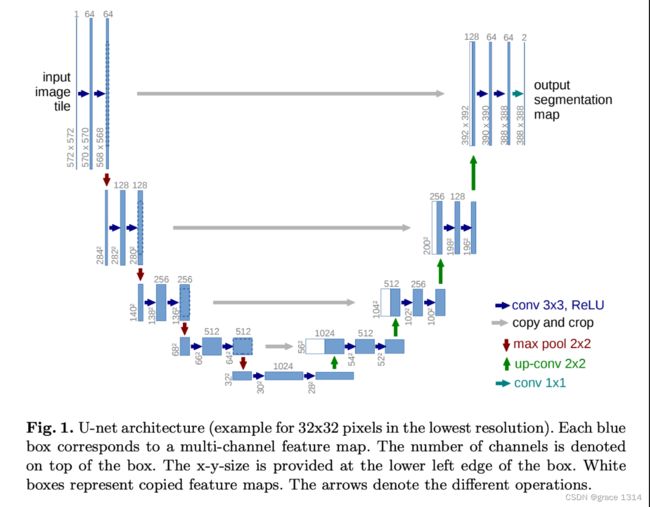
return x10首先,这里书写时,需要对应Unet的网络结构图。
首先Unet结构主要可以分为两个主要块,下采样,编码块与上采样,解码块。
下采样包括卷积,池化,上采样包括卷积,反卷积,这样就可以将这两个主要步骤包装成块,然后最后再组装Unet模型。
除此之外,可以看到,每层两次卷积不改变图像大小,所以卷积采用kernelsize = 3x3,padding=1,stride=1。只有再卷积和池化结构才会改变图像大小,缩小一半或者放大两倍。
再来看看通道数的变换,第一层编码块,第一次卷积是将输入的单通道变为64,我们这里输入的是3通道,也可以输出64通道,第二次卷积不改变通道数,接下来几层也是类似不过是第一次卷积将通道数变为原来2倍。解码块可以依次类推,相反步骤。
除了这些之外,Unet网络还有一个融合或者拼接操作,再对应的U型层,将编码层输出的特征与反卷积的结构进行拼接,这里采用的是cat,cat操作在拼接时,拼接维度的大小可以不同,但其他维度必须相同。这里是对通道数的维度进行拼接,其他维度如H,W,batch_size维度要确保相等才可完成拼接操作。
接下来看实际代码操作:
Encoder类继承torch的nn.module,这是一个子网络,包含initial,和forward实现,initial是构造函数,这个encoder类包含两层 卷积,relu,以及一层maxpool,注意卷积参数以及maxpool参数。
后面的前向传播,是先判断是否进行maxpool,再进行卷积,组成一个编码块,,根据网络结构可以看到第一层不需要maxpool,这里代码的注释有点问题。然后从第二层开始先maxpool,后卷积一直到第5层也是如此。
而decoder类也是类似,不过这里有两点变化,首先是池化变为卷积,前向传播forward也变为先卷积再反卷积扩大图像。这里反卷积采用的是ConvTransposed2d,之后加了一层Relu。解码层的第一次卷积是将通道数减小为输入的一半,后面保持通道数不变。
后面开始组装Unet,同理,Unet继承nn.Module。其构造initial包含:
先是5层encoder,及其对应通道数变化。
之后先加一层手动的反卷积,对第5层的输出进行反卷积,之后加3层解码块,以及最后一层,两个卷积+1X1卷积。最后两个卷积将128通道数降为64,1x1卷积将64通道变为2个通道。由于这里我们的分割任务是mask,只有两类,目标和背景,如果是多目标则需要将2个通道,改为对应目标书+1个背景。
后面就是在forward中进行拼接。这里x1~x10,可能不好看,我大概画一下。
好了以上模型即已经搭好。
现在可以在载入一个批次的数据进行测试。
#检查模型
model = Unet()
#print(model) #查看模型结构
#将数据放入模型
img,label = next(iter(dl_train))
print(img.shape)
pred = model(img)
print(pred.shape)7.训练
这里训练还是和上次目标检测相同,train和test同时开始。多加入了输入的参数,除了模型,traindl,testdl之外还有设备,损失函数,优化器,epoch次数。
在训练时,需要计算的时准确率,Iou。这里的准确率计算并不合理,因为只能看到其预测出来了但是对不对不清楚,所以导致后面的准确率很高。但是IoU时准确的,可以判断该模型的训练好坏。这里采用的是与或运算来求y与ypred的交并比。
这里要提一句,我们的y_pred输出通道是2,这里前面采用argmax挑选了大的值。最后结果还是单通道。
def train_epoch(model,trainloader,testloader,device,loss_fn,optimizer,epoch):
correct = 0 #准确度
total = 0 #总数
running_loss = 0 #损失
epoch_iou = [] #iou
model.train()
for x,y in tqdm(trainloader):
x,y = x.to(device),y.to(device)
y_pred = model(x)
loss = loss_fn(y_pred,y)
optimizer.zero_grad()
loss.backward()
optimizer.step()
with torch.no_grad():
y_pred = torch.argmax(y_pred,dim=1)
correct += (y_pred == y).sum().item()
total += y.size(0)
running_loss +=loss.item()
intersection = torch.logical_and(y,y_pred)
union = torch.logical_or(y,y_pred)
batch_iou = torch.sum(intersection)/torch.sum(union)
epoch_iou.append(batch_iou.item())
epoch_loss = running_loss/len(trainloader.dataset)
epoch_acc = correct/(total*256*256)
test_correct = 0 # 准确度
test_total = 0 # 总数
test_running_loss = 0 # 损失
test_epoch_iou = [] # iou
model.eval()
with torch.no_grad():
for x, y in tqdm(testloader):
x, y = x.to(device), y.to(device)
y_pred = model(x)
loss = loss_fn(y_pred, y)
y_pred = torch.argmax(y_pred, dim=1)
test_correct += (y_pred == y).sum().item()
test_total += y.size(0)
test_running_loss +=loss.item()
intersection = torch.logical_and(y, y_pred)
union = torch.logical_or(y, y_pred)
batch_iou = torch.sum(intersection) / torch.sum(union)
test_epoch_iou.append(batch_iou.item())
test_epoch_loss = test_running_loss / len(testloader.dataset)
test_epoch_acc = test_correct / (test_total * 256 * 256)
#保存模型参数
staic_dict = model.state_dict()
torch.save(staic_dict,'./model_pth/{}_train_Iou_{}_test_Iou_{}.pth'
.format(epoch,round(np.mean(epoch_iou), 3),round(np.mean(test_epoch_iou), 3)))
print(
'epoch: ',epoch,
'loss: ',round(epoch_loss,3),
'accuracy: ',round(epoch_acc,3),
'IOU: ', round(np.mean(epoch_iou), 3),
'test_loss: ',round(test_epoch_loss,3),
'test_accuracy: ',round(test_epoch_acc,3),
'test_IOU: ', round(np.mean(test_epoch_iou), 3)
)
return epoch_loss,epoch_acc,test_epoch_loss,test_epoch_acc后面在进行训练时code:
model = Unet()
#print(model) #查看模型结构
#放到cuda上
device = 'cuda'
model.to(device)
#模型损失函数
loss_fn = nn.CrossEntropyLoss()
optimizer = torch.optim.Adam(model.parameters(),lr = 0.0001)
epochs = 100
for epoch in range(epochs):
train_epoch(model,dl_train,dl_test,device,loss_fn,optimizer,epoch)由于是分类任务,所以采用的损失函数还是交叉熵损失函数,优化器用的Adam自适应优化器。
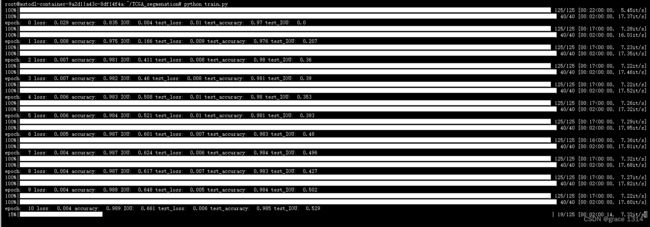
后面就是输入进行训练。训练仍在Autodl上,在上传数据集时注意先将kaggle_3m打包压缩在上传,然后再命令行对其解压再指定文件目录下。因为Autodl不支持上传文件。下面是训练结果,每次epoch的训练权重保存在model_pth文件下。后续训练完后再下载几个比较好的权重文件,后续进行推理。
训练结果:
8.推理测试
dl_train = DataLoader(train_data,batch_size=2,shuffle=True)
dl_test = DataLoader(test_data,batch_size=2,shuffle=True)
#载入权重预测
model = Unet()
start_dict = torch.load('./model_pth/95_train_Iou_0.967_test_Iou_0.695.pth')
model.load_state_dict(start_dict)
model = model.to('cuda')
img,mask = next(iter(dl_test))
img = img.to('cuda')
#模型推理
model.eval()
pred_mask = model(img)
mask = torch.squeeze(mask) #将标签mask的最后一个通道阈值删除。
pred_mask = pred_mask.cpu() #将推理结果放回cpu中
# print(pred_mask.size())
num = 2
plt.figure(figsize=(10, 10))
for i in range(num):
plt.subplot(num, 3, i * 3 + 1)
plt.imshow(img[i].permute(1, 2, 0).cpu().numpy())
plt.subplot(num, 3, i * 3 + 2)
plt.imshow(mask[i].cpu().numpy())
plt.subplot(num, 3, i * 3 + 3)
plt.imshow(torch.argmax(pred_mask[i].permute(1, 2, 0), axis=-1).detach().numpy())
plt.show()以上就是推理的代码。以及下面的推理结果。可以看到效果还不错。