R语言| 缺失值判断
缺失值判断
一、缺失值判断
通常用“NA"表示,判断数据是否存在缺失值的常用方法
- 使用函数is.na( )
使用函数is.na( ),该函数是判断缺失值的最基本的函数。可以用于判断不同的数据对象,如向量、列表和数据框.
其函数的基本书写格式为:is.na( x )
判断数据集中是否存在缺失值,如果存在,返回 TRUE ; 如果不存在,则返回FALSE
例:
>library(DMwR)
>data("algae")
>sum(is.na(algae))
[1] 33
可以看到数据集algae中一共有33个缺失值
- 使用函数complete.cases( )
complete.cases( )判断数据集的每一行是否存在缺失值,如果不存在,则返回TRUE,如果存在,则返回TRUE。
例:
>sum(!complete.cases(algae))
[1] 16
>algae[!complete.cases(algae),] #输出含有缺失值的行
可以看到数据集algae一共有16行记录存在缺失值
- Summary( )判断数据集中分类变量是否含有缺失值
例:

对输出结果进行分析,如可以看到变量mxPH中含有1个缺失值,变量CHla中含有12个缺失值。
二、缺失模型判断
在处理缺失值之前,需要先对缺失模式进行判断,缺失模型主要有以下三种:完全随机缺失(MCAR)、随机缺失(MAR)和完全非随机缺失(MNAR)。
完全随机缺失:数据的缺失不依赖于任何变量
随机缺失:数据的缺失依赖于其他变量,而不由含有缺失值的变量本身决定。
完全非随机缺失:指数据的缺失依赖于变量本身,属于比较严重的问题
缺失值模型的判断方法主要有以下三种:
1、使用md.pattren( )函数
一般使用mice程辑包来判断缺失数据的模式: md.pattren( )

分析输出结果,最左列表明,在200条观测记录中,共有184条记录是完整的,有3条记录缺失变量Chla,有1条记录缺失变量Cl,有7条记录同时缺失变量Chla和变量Cl;最下侧行表明,变量mxPH缺失1个数据,变量Chla缺失12个数据… ,所有变量共缺失33个数据。
- 使用程辑包VIM中的函数 aggr( )进行可视化描述
基本书写格式:aggr(x, delimiter=NULL, plot=TRUE, …. )
x: 一个向量、矩阵或者数据框
delimiter: 一个特征向量,用于区分插补变量,如果赋值则表示变量的值已被插补,如果不赋值,则用于判断缺失模式,默认为NULL
plot:逻辑值,指定是否绘制图形,默认值为TRUE
例:
>library(VIM)
>aggr(algae,numbers=TRUE,ylab=c("Histogram of missing data","Pattern")) #numbers 和 ylab属于函数plot( )中,numbers= TRUE用于指定图形显示相关数据,ylab指定图形的纵坐标名称
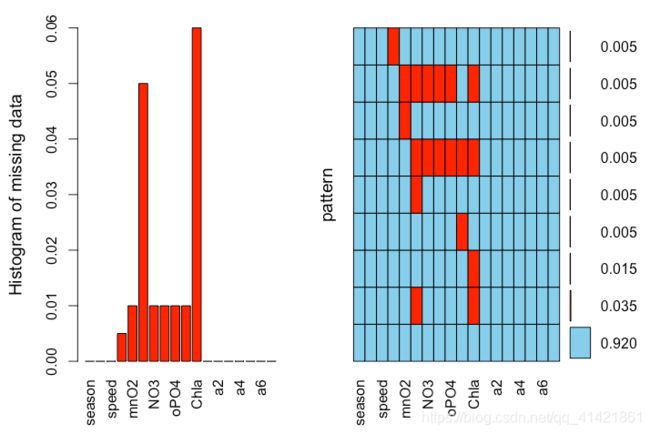 分析输出结果,左边条形图显示了各变量的缺失比例,右边图形显示了综合的缺失情况。可以看出变量Cl与变量Chla的缺失比例是最高的,algae数据集中有92%的数据是完整的。
分析输出结果,左边条形图显示了各变量的缺失比例,右边图形显示了综合的缺失情况。可以看出变量Cl与变量Chla的缺失比例是最高的,algae数据集中有92%的数据是完整的。
3.利用程辑包VIM中的函数marginplot( )绘制箱线图
该函数可以生成一幅散点图,变量的缺失值信息被显示在图形的边界。其基本书写格式为:
marginplot(x, delimiter=NULL, col=c(“skyblue”,“red”,“red4”,“orange”, “orange4”),alpha=NULL,pch=(1,16)….)
其中,x必须是列数为2的矩阵或数据框
例:用algae数据集中的变量mxPH和mnO2来表示:
>marginplot(algae[,4:5])
 图形底部的深色箱线图表示变量mxPH在mnO2缺失下的数据分布,浅色表示mnO2完整下的数据分布,左边含义相同。(由于变量mnO2只含有一个缺失值,所以只有浅色箱线图)
图形底部的深色箱线图表示变量mxPH在mnO2缺失下的数据分布,浅色表示mnO2完整下的数据分布,左边含义相同。(由于变量mnO2只含有一个缺失值,所以只有浅色箱线图)
若同一个变量的两个箱线图比较一致,则可以初步判定缺失数据为完全随机缺失。
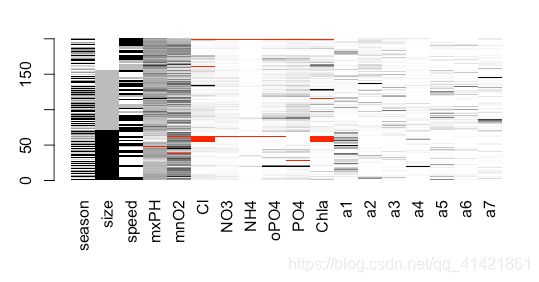
- matrixplot( )函数画散点图
操作对象x可以是任意维的数据,用灰度表示大小,浅色代表值小,深色代表值大,缺失值为红色。基本书写格式为:
matrixplot(x,sortby" ",…)
其中sortby用来指定排序的变量,来观察该变量值的大小是否影响各缺失值。
>matrixplot(algae)