代码+视频,手把手教你R语言ggrcs包绘制限制立方样条图+阈值效应分析
ggrcs包用于绘制限制立方样条图,目前ggrcs包已经升级到3.5版本了,本人出一个规范的教程和视频,包含有代码,供大家参考。
代码+视频,手把手教你R语言ggrcs包绘制限制立方样条图+阈值效应分析
library(ggrcs)
library(rms)
library(ggplot2)
library(scales)
library(cowplot)
dt<-smoke
dd<-datadist(dt)
options(datadist='dd')
fit<- cph(Surv(time,status==1) ~ rcs(age,4)+gender, x=TRUE, y=TRUE,data=dt)
ggrcs(data=dt,fit=fit,x="age")
#改变柱子颜色
ggrcs(data=dt,fit=fit,x="age",histcol="blue")
#改变柱子宽度,默认是0.8
ggrcs(data=dt,fit=fit,x="age",histcol="blue",histbinwidth=1)
#改变可信区间颜色
ggrcs(data=dt,fit=fit,x="age",histcol="blue",histbinwidth=1,ribcol="green")
#改变可信区间透明度,
ggrcs(data=dt,fit=fit,x="age",histcol="blue",histbinwidth=1,ribcol="green",ribalpha=0.5)
#更改X轴和Y轴和标题内容
ggrcs(data=dt,fit=fit,x="age",histcol="blue",
histbinwidth=1,ribcol="green",ribalpha=0.5,xlab ="年龄",ylab="吸烟概率",title ="年龄和吸烟概率关系")
#关掉左轴
ggrcs(data=dt,fit=fit,x="age",histcol="blue",
histbinwidth=1,ribcol="green",ribalpha=0.5,xlab ="年龄",ylab="吸烟概率",
title ="年龄和吸烟概率关系",lift=F)
#双组
ggrcs(data=dt,fit=fit,x="age",group="gender")
#自定义颜色
ggrcs(data=dt,fit=fit,x="age",group="gender",groupcol=c("red","blue"),histbinwidth=1)
#更改左轴名字
ggrcs(data=dt,fit=fit,x="age",group="gender",
groupcol=c("red","blue"),histbinwidth=1,ribalpha=0.5,xlab ="年龄",
ylab="吸烟HR",title ="年龄和吸烟概率关系",liftname="概率密度")
#调整字体位置
ggrcs(data=dt,fit=fit,x="age",group="gender",
groupcol=c("red","blue"),histbinwidth=1,ribalpha=0.5,xlab ="年龄",
ylab="吸烟HR",title ="年龄和吸烟概率关系",px=10,py=18)
#风格定制 深海绿+黄金1
ggrcs(data=dt,fit=fit,x="age",group="gender",colset = "A")
###########逻辑回归
library(foreign)
be <- read.spss("E:/r/test/Breast cancer survival agec.sav",
use.value.labels=F, to.data.frame=T)
be <- na.omit(be)
be$ln_yesno<-as.factor(be$ln_yesno)
dd <- datadist(be)
options(datadist='dd')
fit <-lrm(status ~ rcs(age, 4)+ln_yesno,data=be)
ggrcs(data=be,fit=fit,x="age")
ggrcs(data=be,fit=fit,x="age",histbinwidth=1)
ggrcs(data=be,fit=fit,x="age",group="ln_yesno",histbinwidth=1)
##########线性回归
library(foreign)
be <- read.spss("E:/r/test/ozone.sav",
use.value.labels=F, to.data.frame=T) #????
be$variables2<-sample(0:1,size=330,replace=TRUE)
be$variables2<-as.factor(be$variables2)
dd <- datadist(be)
options(datadist='dd')
fit<-ols(ozon ~rcs(temp, 4)+dpg+variables2,data=be)
ggrcs(data=be,fit=fit,x="temp",histbinwidth=1)
ggrcs(data=be,fit=fit,x="temp",group="variables2",histbinwidth=1)
ggrcs(data=be,fit=fit,x="temp",group="variables2",histbinwidth=1,
groupcol=c("red","blue"),bordercol="green")
##############singlercs
singlercs(data=dt,fit=fit,x="age")
singlercs(data=dt,fit=fit,x="age",group="gender")
#########
source("E:/r/test/cut.tab1.3.R")
fit<- cph(Surv(time,status==1) ~ rcs(age,4)+gender, x=TRUE, y=TRUE,data=dt)
fit1 <-coxph(Surv(time,status==1) ~ age,data=dt)
out<-cut.tab(fit1,"age",dt)
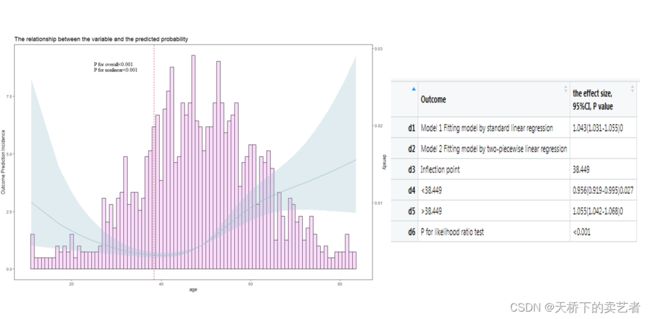
p<-ggrcs(data=dt,fit=fit,x="age")
p+geom_vline(aes(xintercept=38.449),colour="#BB0000", linetype="dashed")