ATAT-mcsqs生成准随机结构(SQS)
经验分享
通常使用第一性原理计算某些多元素占据原胞中同一位置的结构会优先考虑使用准随机结构(special quasirandom structure,SQS)来进行模拟建模。此篇教程意在整理一个较为简便的操作流程,以供参考。
首先,准备原始单一元素(共格的多元素合并为一种)晶胞,并通过vaspkit获得其最原始晶胞。
例如这里准备了一个面心立方的结构,组分单一,面心和顶角元素相同,这里设置为Si。
Si1.000000000000004 0.0000000000000000 0.00000000000000000.0000000000000000 4 0.00000000000000000.0000000000000000 0.0000000000000000 4Si4Direct0.0000000000000000 0.0000000000000000 0.00000000000000000.0000000000000000 0.5000000000000000 0.50000000000000000.5000000000000000 0.0000000000000000 0.50000000000000000.5000000000000000 0.5000000000000000 0.00000000000000000.0000000000000000 0.5000000000000000 0.0000000000000000
通过vaspkit 602功能获得其原胞
vaspkit -task 602+---------------------------------------------------------------+| VASPKIT Standard Edition 1.3.5 (03 Jul. 2022) || Running VASPKIT Under Command-Line Mode |+---------------------------------------------------------------+-->> (01) Reading Structural Parameters from POSCAR File...+-------------------------- Summary ----------------------------+Prototype: ATotal Atoms in Input Cell: 4Lattice Constants in Input Cell: 4.000 4.000 4.000Lattice Angles in Input Cell: 90.000 90.000 90.000Total Atoms in Primitive Cell: 1Lattice Constants in Primitive Cell: 2.828 2.828 2.828Lattice Angles in Primitive Cell: 60.000 60.000 60.000Crystal System: CubicCrystal Class: m-3mBravais Lattice: cFSpace Group: 225Point Group: 32 [ Oh ]International: Fm-3mSymmetry Operations: 192+---------------------------------------------------------------+-->> (02) Written PRIMCELL.vasp file.+---------------------------------------------------------------+
原胞信息如下,只存在一个wyckoff点位。
Primitive Cell1.0000000.00000000000000 2.00000000000000 2.000000000000002.00000000000000 0.00000000000000 2.000000000000002.00000000000000 2.00000000000000 0.00000000000000Si1DIRECT0.0000000000000000 0.0000000000000000 0.0000000000000000 Si1
将vaspkit生成的原胞文件PRIMCELL.vasp复制为POSCAR,然后通过vaspkit 的414功能将POSCAR转化为ATAT的输入文件lat.in
vaspkit -task 414+---------------------------------------------------------------+| VASPKIT Standard Edition 1.3.5 (03 Jul. 2022) || Running VASPKIT Under Command-Line Mode |+---------------------------------------------------------------+-->> (01) Reading Structural Parameters from POSCAR File...-->> (02) Written lat.in File!+---------------------------------------------------------------+
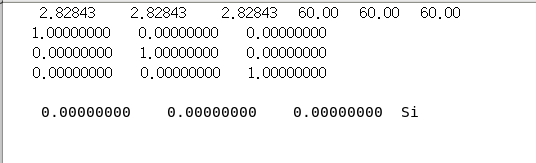
lat.in文件内容如下,后续计算需要重命名为rndstr.in
根据实际需要的某一wyckoff点位的元素比例修改rndst.in文件。这里修改为Si和C各占50%。修改后的rndst.in文件如下
2.82843 2.82843 2.82843 60.00 60.00 60.001.00000000 0.00000000 0.000000000.00000000 1.00000000 0.000000000.00000000 0.00000000 1.000000000.00000000 0.00000000 0.00000000 Si=0.5,C=0.5
注意:各点位各元素之间的比例与扩包比例相关联,最后确保扩包后各元素原子数量总数为整数。
这里选择扩包方式为2×2×2,最后超胞总原子数量为8。
先使用ATAT获得in文件的对称性信息,-2=x需要根据结构实际情况修改,过小会导致ATAT出现段错误。
corrdump -nop -noe -2=4 -ro -l=rndstr.in -clus ; getclus再根据超胞的总原子数获得结构信息,-n=后面的数值与超胞数量相同
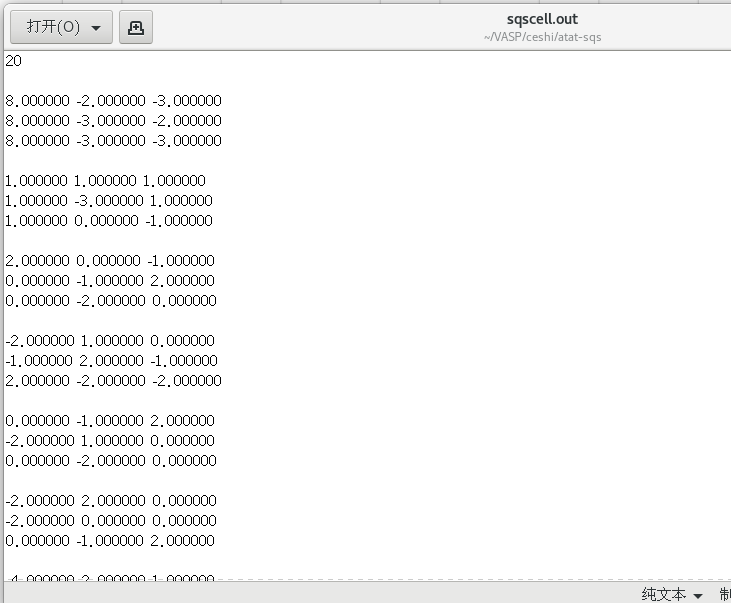
mcsqs -n=8ATAT产生的超胞结构信息文件为sqscell.out文件
根据既定扩包方式修改为:
12 0 00 2 00 0 2
生成超胞准随机结构:
mcsqs -rc产生的准随机结构文件为:bestsqs.out
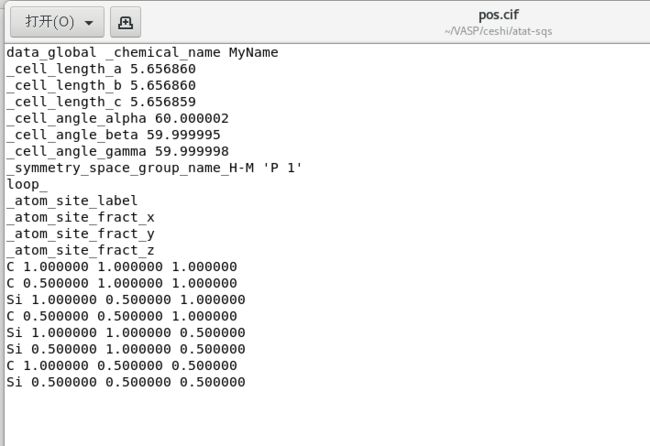
导出为cif文件
str2cif < bestsqs.out > pos.cif通过vaspkit 将cif文件转化为POSCAR
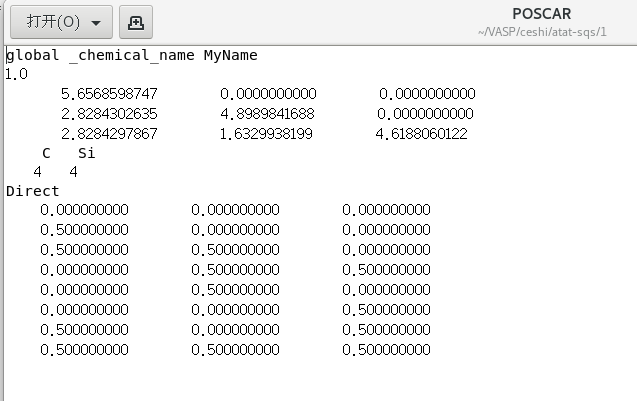
vaspkit -task 105+---------------------------------------------------------------+| VASPKIT Standard Edition 1.3.5 (03 Jul. 2022) || Running VASPKIT Under Command-Line Mode |+---------------------------------------------------------------+Input the filename in cif format (e.g., POSCAR.cif):------------>>pos.cif-->> (01) Written POSCAR file!
更为简便的方法可使用qvasp软件一键转化
qvasp -c2p当然也可以通过VESTA导出。
POSCAR文件如下
愿有所成
IEchoQ
引喻失义 妄自菲薄