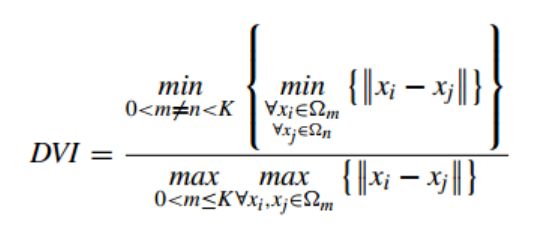聚类模型评估指标之内部方法
欢迎关注”生信修炼手册”!
聚类算法的理想结果是同一类别内的点相似度高,而不同类别之间的点相似度低。聚类属于无监督学习,数据没有标签,为了比较不同聚类模型的好坏,我们也需要一些定量的指标来进行评估。根式是否提供样本的标签信息,相关的指标可以分为以下两大类
1. 外部方法,外部方法指的是从外部提供数据的标签,比如通过专家认为定义类别,或者是本身就是有标签的数据,将标签拿掉之后做聚类
2. 内部方法,内部方法指的是不需要数据的标签,仅仅从聚类效果本身出发,而制定的一些指标
本文主要关注内部方法,常用的指标有以下几种
1. 簇内误差平方和
within-cluster sum of square error, 简称SSE,公式如下
针对每一个聚类簇,计算簇内样本与聚类中心点的距离,然后加和。理论上,该数值越小越好。该指标的局限性在于只考虑了簇内相似度,没有考虑不同簇之间的关系。
2. Compactness
简称CP, 称之为紧密性,公式如下
针对单个聚类簇,计算簇内样本与中心点的平均距离,最后取所有簇的平均值即可计算出该指标。和SSE类似,也是只考虑了簇内相似度, 数值越小,聚类效果越好。
3. Separation
简称SP, 称之为间隔性,公式如下
w表示聚类的中心点,通过计算两两聚类中心点的距离来得到最终的数值。和紧密型相反,该指标仅仅考虑不同簇之间的距离,数值越大,聚类效果越好。
4. Silhouette Coefficient
称之为轮廓系数,对于某个样本而言,将该样本与簇内其他样本点之间的平均距离定义为簇的内聚度a, 将该样本与最近簇中所有样本点之间的平均距离定义为簇之间的分离度b, 则该样本轮廓系数的计算公式如下
对于全体样本的集合而言,轮廓系数是每个样本轮廓系数的平均值。该指标的取值范围-1到1,当簇间分离度b远大于内聚度a时,轮廓系数的值近似于1。所以该指标的值接近1,聚类效果越佳。
5. Calinski-Harabaz Index
简称为CH指数,综合考虑了簇间距离和簇内距离,计算公式如下
其中SSB表示的是簇内距离,SSW表示簇间距离,簇内距离用簇内样本点与簇中心点的距离表示,簇间距离用样本点与其他簇内中心点的距离表示,具体的计算公式表述如下
CH的数值越大,说明簇内距离越小,簇间距离越大,聚类效果越好。
6. Davies-Bouldin Index
简称DBI, 称之为戴维森堡丁指数,公式如下
其中avg(C)表示聚类簇的紧密程度,公式如下
计算该聚类簇内样本点的距离,d表示不同聚类簇中心点之间的距离,公式如下
聚类簇之间的距离越远,聚类内的距离越近,DB指数的值越小,聚类性能越好。
7. Dunn Validity Index
简称DVI, 称之为邓恩指数,公式如下
分子为聚类簇间样本的最小距离,分母为聚类簇内样本的最大距离,类间距离越大,类内距离越小,DVI指数的值越大,聚类性能越好。
·end·
—如果喜欢,快分享给你的朋友们吧—
原创不易,欢迎收藏,点赞,转发!生信知识浩瀚如海,在生信学习的道路上,让我们一起并肩作战!
本公众号深耕耘生信领域多年,具有丰富的数据分析经验,致力于提供真正有价值的数据分析服务,擅长个性化分析,欢迎有需要的老师和同学前来咨询。
更多精彩
KEGG数据库,除了pathway你还知道哪些
全网最完整的circos中文教程
DNA甲基化数据分析专题
突变检测数据分析专题
mRNA数据分析专题
lncRNA数据分析专题
circRNA数据分析专题
miRNA数据分析专题
单细胞转录组数据分析专题
chip_seq数据分析专题
Hi-C数据分析专题
HLA数据分析专题
TCGA肿瘤数据分析专题
基因组组装数据分析专题
CNV数据分析专题
GWAS数据分析专题
2018年推文合集
2019年推文合集
2020推文合集
写在最后
转发本文至朋友圈,后台私信截图即可加入生信交流群,和小伙伴一起学习交流。
扫描下方二维码,关注我们,解锁更多精彩内容!
一个只分享干货的
生信公众号