文献速递:人工智能医学影像分割--- 深度学习分割骨盆骨骼:大规模CT数据集和基线模型
文献速递:人工智能医学影像分割— 深度学习分割骨盆骨骼:大规模CT数据集和基线模型
我们为大家带来人工智能技术在医学影像分割上的应用文献。
人工智能在医学影像分析中发挥着至关重要的作用,尤其体现在图像分割技术上。这项技术的目的是准确划分人体重要器官和异常物体,例如肺部、结节、肿瘤等。高质量的图像分割结果对于医疗手术的规划至关重要,同时也在疾病的诊断和预后中发挥着重要作用。它能够帮助医生清晰地标记出病变部位的确切位置,并确定其它重要特征,如大小、体积等。采用基于人工智能的解决方案,能够显著提高这些程序的效率和精准度,大大缩短所需时间。
01
文献速递介绍
骨盆是连接脊柱和下肢的重要结构,在维持身体稳定和保护腹部内部器官方面发挥着至关重要的作用。骨盆的异常,如髋关节发育不良和骨盆骨折等,会对我们的身体健康产生严重影响。例如,骨盆骨折作为最严重和危及生命的骨损伤,会损伤骨折部位的其他器官,在最严重的情况下,骨盆开放性骨折的死亡率可达45%。医学影像学在骨盆损伤患者的诊断和治疗的整个过程中发挥着重要作用。与X射线图像相比,CT保留了实际的解剖结构,包括深度信息,为外科医生提供了更多关于损伤部位的细节,因此常用于三维重建,以便进行后续手术计划和术后效果评估。在这些应用中,准确的骨盆骨分割对于评估骨盆损伤的严重程度至关重要,有助于外科医生做出正确的判断和选择合适的手术入路。过去,外科医生使用Mimics1等软件从CT手动分割骨盆,这既耗时又不可重复。
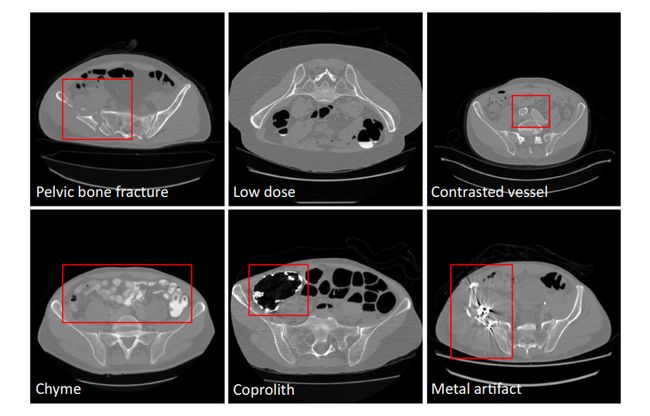
为了满足这些临床需求,本文提出了一种能够准确快速地从CT中分割骨盆骨的自动算法。现有的从CT中分割骨盆骨的方法大多使用简单的阈值、区域生长和手工模型,其中包括可变形模型、统计形状模型、分水岭和其他。这些方法专注于局部灰度信息,由于皮质骨和骨小梁之间的密度差异,精度有限。而骨小梁在纹理和强度方面与周围组织相似。如果存在骨折,则进一步导致弱边缘。最近,基于深度学习的方法在图像分割方面取得了巨大的成功;然而,它们对CT骨盆骨分割的有效性还不完全清楚。虽然有一些与骨盆骨相关的数据集,但其中只有少数是开源的,并且大小较小(小于5张图像或200张切片),远远少于其他器官。虽然进行了基于深度学习的实验,但结果并不是很好(骰子=0.92),数据集只有200个CT片。对于深度学习方法的鲁棒性,拥有一个包括尽可能多的真实场景的综合数据集至关重要。本文通过策划一个大规模的CT数据集来弥补这一差距,并探索深度学习在这一任务中的应用,据我们所知,这是该领域的第一次真正尝试,具有更多的统计意义和参考价值。为了构建一个全面的数据集,我们必须处理由于成像分辨率和视野(FOV)的差异、不同部位产生的域移位、造影血管、粪便和食糜、骨折、低剂量、金属伪影等因素引起的各种图像外观变化。图1给出了这些不同条件的一些示例。在上述外观变化中,金属伪影的挑战是最难处理的。此外,本文的目标是将骨盆分割为多个骨头,包括腰椎、骶骨、左髋和右髋,而不是简单地从CT中分割出整个骨盆。本文的贡献总结如下:- 从多个领域和不同制造商汇集的骨盆CT数据集,包括1184个CT卷(超过320K CT切片)的不同外观变化(包括75个带有金属伪影的CT)。它们的多骨标签由专家仔细注释。我们将其开源以造福整个社区;- 学习一个深度多类分割网络[14],从多领域标记的图像中获得腰椎、骶骨、左髋和右髋分割的更有效表示,从而获得所需的准确性和鲁棒性;- 一个全自动分析管道,实现了高精度、高效率和鲁棒性,从而使其在临床实践中具有潜在的应用价值。
Title
题目
Deep learning to segment pelvic bones: large-scale CT datasets and baseline models
深度学习分割骨盆骨骼:大规模CT数据集和基线模型
Abstract
摘要
Pelvic bone segmentation in CT has always been an essential step in clinical diagnosis and surgery planning of pelvic bone diseases. Existing methods for pelvic bone segmentation are either hand-crafted or semi-automatic and achieve limited accuracy when dealing with image appearance variations due to the multi-site domain shift, the presence of contrasted vessels, coprolith and chyme, bone fractures, low dose, metal artifacts, etc. Due to the lack of a large-scale pelvic CT dataset with annotations, deep learning methods are not fully explored.
在CT中对骨盆骨骼的分割一直是临床诊断和骨盆骨疾病手术规划中的一个重要步骤。现有的骨盆骨骼分割方法要么是手工制作的,要么是半自动的,当面对由于多站点域转换、对比鲜明的血管、粪石和食糜、骨折、低剂量、金属伪影等因素造成的图像外观变化时,这些方法的准确性有限。由于缺乏一个带有注释的大规模骨盆CT数据集,深度学习方法尚未得到充分探索。
Methods
方法
In this paper, we aim to bridge the data gap by curating a large pelvic CT dataset pooled from multiple sources, including 1184 CT volumes with a variety of appearance variations. Then, we propose for the first time, to the best of our knowledge, to learn a deep multi-class network for segmenting lumbar spine, sacrum, left hip, and right hip, from multiple-domain images simultaneously to obtain more effective and robust feature representations. Finally, we introduce a post-processor based on the signed distance function (SDF).
在本文中,我们旨在通过从多个来源整理一个大型骨盆CT数据集来弥补数据差距,包括1184个CT体积和多种外观变化。然后,据我们所知,我们首次提出学习一个深度多类网络,用于同时从多域图像中分割腰椎、骶骨、左髋和右髋,以获得更有效和稳健的特征表示。最后,我们介绍了一种基于有符号距离函数(SDF)的后处理器。
Results
结果
Extensive experiments on our dataset demonstrate the effectiveness of our automatic method, achieving an average Dice of 0.987 for a metal-free volume. SDF post-processor yields a decrease of 15.1% in Hausdorff distance compared with traditional post-processor.
我们对数据集进行的广泛实验证明了我们自动方法的有效性,对于无金属伪影的体积,达到了平均Dice系数0.987。与传统后处理器相比,SDF后处理器使豪斯多夫距离减少了15.1%。
Conclusions
结论
We believe this large-scale dataset will promote the development of the whole community and open source the images, annotations, codes, and trained baseline models at https://github.com/ICT-MIRACLE-lab/CTPelvic1K. Keywords CT dataset · Pelvic segmentation · Deep learning · SDF post-processing
我们相信这个大规模数据集将促进整个社区的发展,并在https://github.com/ICT-MIRACLE-lab/CTPelvic1K上开源图像、注释、代码和训练好的基线模型。关键词 CT数据集 · 骨盆分割 · 深度学习 · SDF后处理
Figure
图
Fig. 1 Pelvic CT image examples with various conditions
图 1. 不同条件下的骨盆CT图像示例
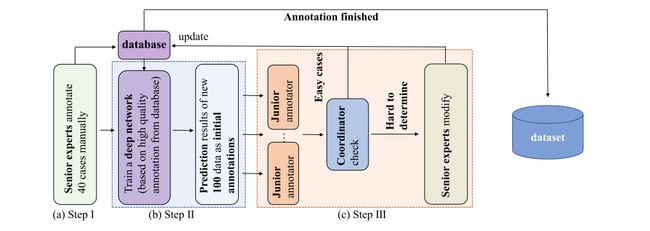
Fig. 2 The designed annotation pipeline based on an AID (Annotation by Iterative Deep Learning) strategy. In Step I, two senior experts first manually annotate 40 cases of data as the initial database. In Step II, we train a deep network based on the human annotated database and use it to predict new data. In Step III, initial annotations from the deep network are checked and modified by human annotators. Step II and Step III are repeated iteratively to refine a deep network to a more and more powerful ‘annotator’. This deep network ‘annotator’ also unifies the annotation standards of different human annotators
图 2 基于AID(通过迭代深度学习的注释)策略设计的注释流程图。在第一步中,两位资深专家首先手动标注40例数据作为初始数据库。在第二步中,我们基于人工注释的数据库训练一个深度网络,并用它来预测新数据。在第三步中,深度网络的初始注释由人类注释者检查和修改。第二步和第三步重复迭代,以精炼深度网络成为一个越来越强大的‘注释者’。这个深度网络‘注释者’也统一了不同人类注释者的标注标准。
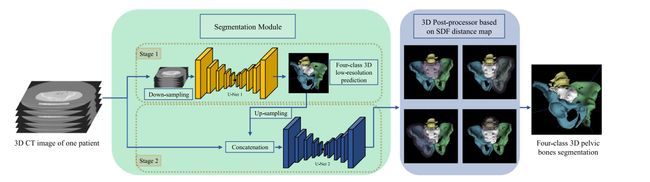
Fig. 3 Overview of our pelvic bones segmentation system, which learns from multi-domain CT images for effective and robust representations. The 3D U-Net cascade is used here to exploit more spatial information in 3D CT images. SDF is introduced to our post-processor to add distance constraint besides size constraint used in traditional MCR-based method
图 3 我们骨盆骨骼分割系统的概览,该系统从多域CT图像中学习,以获得有效和稳健的表示。这里使用了3D U-Net级联,以利用3D CT图像中更多的空间信息。在我们的后处理器中引入了SDF,除了传统的基于MCR方法使用的大小约束之外,还增加了距离约束。
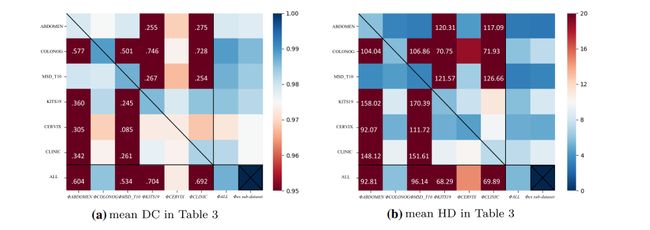
Fig. 4 Heat map of DC & HD results in Table 3. The vertical axis represents different sub-datasets, and the horizontal axis represents different models. In order to show the normal values more clearly, we clip some outliers to the boundary value, i.e., 0.95 in DC and 30 in HD. The values out of range are marked in the grid. The cross in the lower right corner indicates that there is no corresponding experiment
图 4 表 3中DC和HD结果的热图。垂直轴代表不同的子数据集,水平轴代表不同的模型。为了更清楚地显示正常值,我们将一些异常值剪切到边界值,即DC中的0.95和HD中的30。超出范围的值在网格中标记。右下角的叉号表示没有相应的实验。
Fig. 5 Visualization of segmentation results from six datasets tested on different models. Among them, the white, green, blue, and yellow parts of the segmentation results represent the sacrum, left hip bone, right hip bone, and lumbar spine, respectively
图 5 六个数据集在不同模型上测试的分割结果可视化。其中,分割结果的白色、绿色、蓝色和黄色部分分别代表骶骨、左髋骨、右髋骨和腰椎。
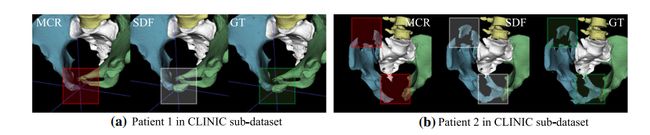
Fig. 6 Comparison between post-processing methods: traditional MCR and the proposed SDF filtering
图 6 后处理方法的比较:传统的MCR和提出的SDF过滤。
Table
表
Table 1 Overview of our large-scale Pelvic CT dataset. ‘#’ represents the number of 3D volumes. ‘Tr/Val/Ts’ denotes training/validation/testing set. Ticks [] in table refer to we can access
the CT images’ acquisition equipment manufacturer[M] information of that sub-dataset. Due to the difficulty of labeling the CLINIC-metal, CLINIC-metal is taken off in our supervised training phase
表 1 我们大规模骨盆CT数据集概览。‘#’代表3D体积的数量。‘Tr/Val/Ts’表示训练/验证/测试集。表格中的勾选框[]表示我们可以访问该子数据集的CT图像采集设备制造商[M]信息。由于标记CLINIC-metal的困难,CLINIC-metal在我们的监督训练阶段被剔除。
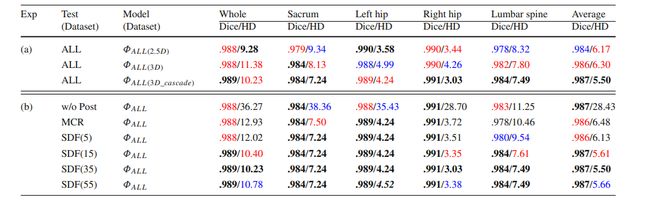
Table 2 (a) The DC and HD results for different models tested on ‘ALL’ dataset. (b) Effect of different post-processing methods on ‘ALL’ dataset. ‘ALL’ refers to the six metal-free sub-datasets. ‘Average’ refers to the mean value of four anatomical structures’ DC/HD. ‘Whole’ refers to treating sacrum, left hip, right hip, and lumbar spine as a whole bone. The top three numbers in each part are marked in bold, red, and blue
表 2 (a) 在‘ALL’数据集上测试的不同模型的DC和HD结果。(b)不同后处理方法在‘ALL’数据集上的效果。‘ALL’指的是六个无金属伪影的子数据集。‘Average’指的是四个解剖结构的DC/HD的平均值。‘Whole’指的是将骶骨、左髋、右髋和腰椎视为一个整体骨骼。每部分前三个数字以粗体、红色和蓝色标记。
Table 3 The ‘Average’ DC and HD results for different models tested on different datasets. Please refer to the Online Resource 1 for details. The top three numbers in each part are marked in bold, red, and blue
表 3 在不同数据集上测试的不同模型的‘平均’DC和HD结果。详情请参阅在线资源1。每部分前三个数字以粗体、红色和蓝色标记。