分子对接软件gnina主要功能与使用案例
分子对接软件gnina主要功能与使用案例
文章目录
- 分子对接软件gnina主要功能与使用案例
- 前言
- 一、关于gnina介绍
- 二、使用方法及案例
-
- 1. gnina usage
- 2. 配体文件与受体文件准备
- 3. gnina 分子对接案例
-
- 3.1 一般用法,在binding site对接
- 3.2 CNN refinement
- 3.3 single CNN
- 3.4 none CNN:Smina
- 3.5 none CNN:vinardo打分
- 总结
- 参考文献:
前言
(1)通过案例介绍gnina常见对接打分功能的使用;
(2)总结gnina的特点及用途。
一、关于gnina介绍
官方描述:gnina (pronounced NEE-na) is a molecular docking program with integrated support for scoring and optimizing ligands using convolutional neural networks. It is a fork of smina, which is a fork of AutoDock Vina.
gnina的安装,可参考前篇博文:卷积神经网络算法分子对接软件-gnina的编译安装
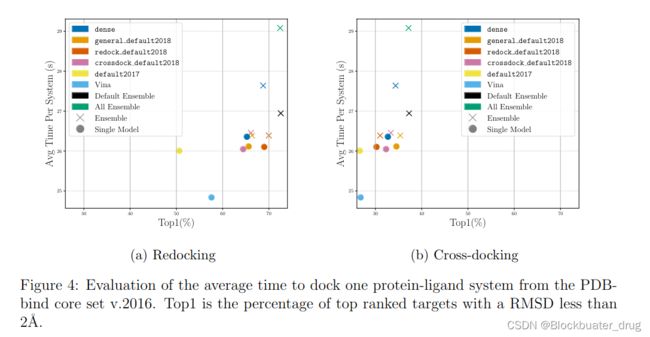
single CNN和 CNN Ensemble效果均优于vina。
在CASF-2016上测试了运行时间,single 和Ensemble模型时间差异不大,与vina相比有增加,但不明显;实际使用中CPU单线程、GPU加速情况下每个分子的对接时间均在30s左右。
从Refinment阶段开始使用CNN打分,相对rescore并没有明显提升对接效果。


二、使用方法及案例
1. gnina usage
场景 1: 要将配体lig.sdf对接到由另一配体orig.sdf定义的rec.pdb上的结合位点
gnina -r rec.pdb -l lig.sdf --autobox_ligand orig.sdf -o docked.sdf
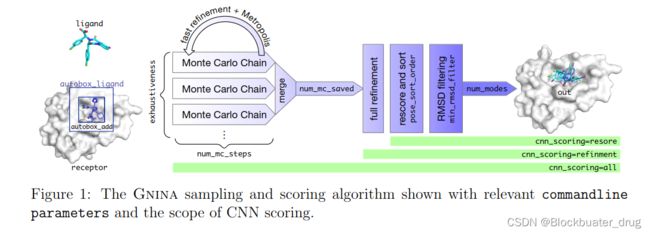
场景 2: 要在对接的refinement步骤能量最小化中使用默认的集成CNN 打分参数(比默认的rescore选项慢10倍)
gnina -r rec.pdb -l lig.sdf --autobox_ligand orig.sdf --cnn_scoring refinement -o cnn_refined.sdf
场景 3: 在docking阶段使用不同的CNN模型(参考help文件,该项可能在特定项目中非常有用):
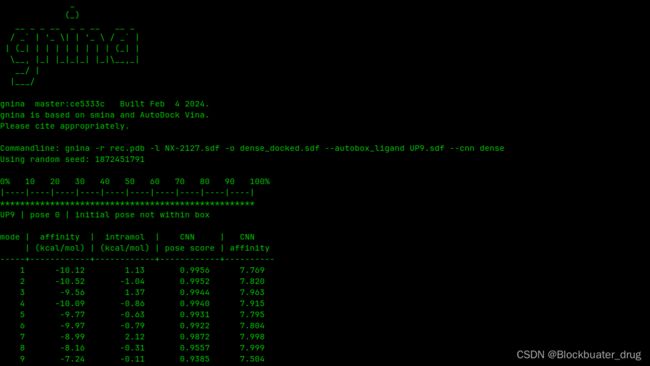
gnina -r rec.pdb -l lig.sdf --autobox_ligand orig.sdf --cnn dense -o dense_docked.sdf
场景 4: 使用Vinardo打分函数评分:
gnina -r rec.pdb -l lig.sdf --autobox_ligand orig.sdf --cnn_scoring none --scoring vinardo -o vinardo_docked.sdf
场景 5: 在pose refinement和rescore均不使用CNN参数(等同于Smina的效果):
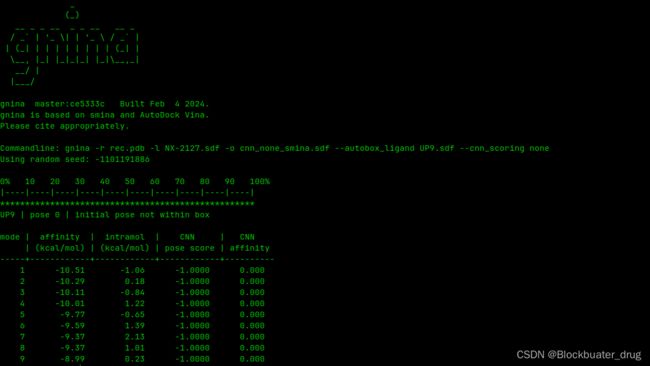
gnina -r rec.pdb -l lig.sdf --autobox_ligand orig.sdf --cnn_scoring none -o cnn_none_smina.sdf
gnina常用选项:(更详细的请参考 gnina --help )
Input:
-r [ --receptor ] arg rigid part of the receptor
-l [ --ligand ] arg ligand(s)
--flexdist_ligand arg Ligand to use for flexdist
--flexdist arg set all side chains within specified
distance to flexdist_ligand to flexible
Search space (required):
--autobox_ligand arg Ligand to use for autobox
Scoring and minimization options:
--scoring arg specify alternative built-in scoring
function: ad4_scoring default dkoes_fast
dkoes_scoring dkoes_scoring_old vina
vinardo
--custom_scoring arg custom scoring function file
--score_only score provided ligand pose
--minimize energy minimization
Convolutional neural net (CNN) scoring:
--cnn_scoring arg (=1) Amount of CNN scoring: none, rescore
(default), refinement, metrorescore
(metropolis+rescore), metrorefine
(metropolis+refine), all
--cnn arg crossdock_default2018
crossdock_default2018_1
crossdock_default2018_2
crossdock_default2018_3
crossdock_default2018_4 default2017 dense
dense_1 dense_2 dense_3 dense_4
general_default2018 general_default2018_1
general_default2018_2
general_default2018_3
general_default2018_4 redock_default2018
redock_default2018_1 redock_default2018_2
redock_default2018_3 redock_default2018_4
Output:
-o [ --out ] arg output file name, format taken from file
extension
Misc (optional):
--cpu arg the number of CPUs to use (the default is
to try to detect the number of CPUs or,
failing that, use 1)
--num_modes arg (=9) maximum number of binding modes to
generate
--addH arg automatically add hydrogens in ligands (on
by default)
gnina可设置固有配体一定范围内残基柔性(–flexdist),也可用于共价对接,此处不再展示,具体请参考帮助文档。
2. 配体文件与受体文件准备
靶点背景:NX-2127是一款具有双重活性的口服小分子靶向蛋白降解剂,可以布鲁顿氏酪氨酸激酶(BTK)蛋白。在与BTK结合的同时,NX-2127还可以招募E3泛素连接酶,使BTK蛋白“泛素化”,从而BTK被蛋白酶体降解。BTK抑制剂作为肿瘤和自身免疫疾病靶点的有效性已在临床上得到了验证,目前已有多款BTK抑制剂获批上市。以分子对接为基础的虚拟筛选可以帮助筛选与靶点蛋白有效结合的小分子。
结构文件来源:
NX-2127与BTK的X-ray共晶结构此前得到解析,pdbid 8U2E,配体NX-2127编号为UP9。
NX-2127.sdf: 将NX-2127结构保存为2D sdf格式文件;
rec.pdb: 将8U2E蛋白主链单独保存为pdb格式文件;
UP9.sdf: 将8U2E中配体UP9单独保存为3D sdf格式文件。
3. gnina 分子对接案例
3.1 一般用法,在binding site对接
在虚拟筛选时,binding site明确的情况是常见的。
CNN默认的打分函数:5个CNN模型dense,general_default2018_3,dense_3,crossdock_default2018和redock_default2018的集成,以平衡pose预测性能和运行时间。
gnina默认参数:配体空间增量autobox_add默认为4A,减小有利于提升pose精度,增加则相反;MC后保留的最佳pose数量num_mc_saved默认50,增加可提升对接性能但是更消耗算力,可根据算力资源而调整;其余默认参数num_modes, min_rmsd_filter和cnn_rotation对结果影响较小。
gnina -r rec.pdb -l NX-2127.sdf -o cnn_rescore.sdf --autobox_ligand UP9.sdf
#调整的参数:
gnina -r rec.pdb -l NX-2127.sdf -o docked_NX-2127_2.5_200.sdf --autobox_ligand UP9.sdf \
--autobox_add 2.5 \
--num_mc_saved 200
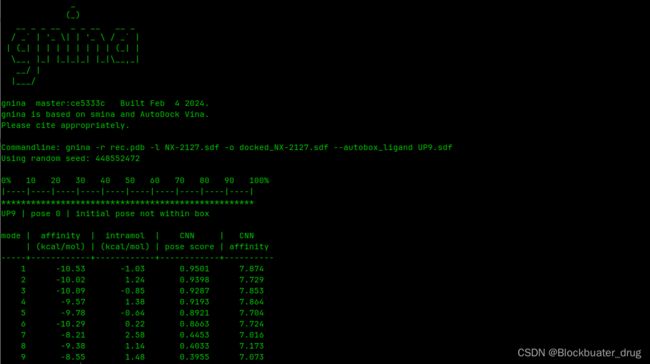
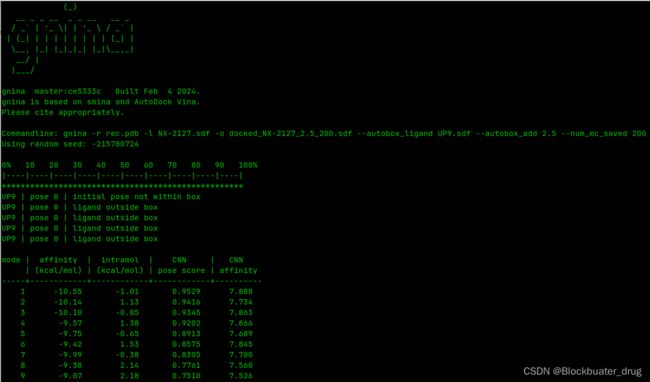 结果中较高的CNN pose score(比如>0.8)可以作为pose质量的一个指标。
结果中较高的CNN pose score(比如>0.8)可以作为pose质量的一个指标。
3.2 CNN refinement
要在对接的refinement步骤能量最小化中使用默认的集成CNN 打分参数(比默认的rescore选项慢10倍)。
gnina -r rec.pdb -l NX-2127.sdf -o cnn_refine.sdf --autobox_ligand UP9.sdf --cnn_scoring refinement
3.3 single CNN
可使用不同的CNN模型(参考help文件);在特定项目中如果找到合适的CNN模型或者其集成,可能会产生更好的效果,这也是Gnina的潜在价值。
gnina -r rec.pdb -l NX-2127.sdf -o default2017_docked.sdf --autobox_ligand UP9.sdf --cnn default2017
gnina -r rec.pdb -l NX-2127.sdf -o dense_docked.sdf --autobox_ligand UP9.sdf --cnn dense
3.4 none CNN:Smina
gnina是Smina的CNN“增强版”,当不使用CNN打分时,就回到了Smina。
gnina -r rec.pdb -l NX-2127.sdf -o cnn_none_smina.sdf --autobox_ligand UP9.sdf --cnn_scoring none
3.5 none CNN:vinardo打分
也可使用Vinardo打分函数。
gnina -r rec.pdb -l NX-2127.sdf -o vinardo_docked.sdf --autobox_ligand UP9.sdf --cnn_scoring none --scoring vinardo
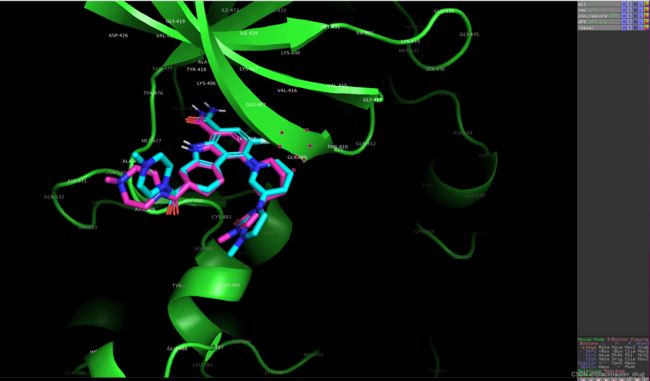
 可通过Pymol等打开对接结果的sdf文件,分子比较对接pose和结合模式的合理性,为进一步分子结构改造提供参考。
可通过Pymol等打开对接结果的sdf文件,分子比较对接pose和结合模式的合理性,为进一步分子结构改造提供参考。
总结
(1)gnina在公共数据集上redock和cross-dock结果显示,性能显著优于vina,合理的CNN Ensemble提供相对最优TopN%性能 。
(2)CNN score可以作为pose质量的评价方式,比如>0.8对应的pose更准确。
(3)single CNN模型的合理组合可能找到适合特定靶点的集成打分函数。
以上就是今天介绍的内容,关于gnina的主要功能的使用。欢迎批评指正与交流。
参考文献:
(1)GNINA 1.0: Molecular docking with deep learning (Primary application citation)
A McNutt, P Francoeur, R Aggarwal, T Masuda, R Meli, M Ragoza, J Sunseri, DR Koes. J. Cheminformatics, 2021
(2)Protein–Ligand Scoring with Convolutional Neural Networks (Primary methods citation)
M Ragoza, J Hochuli, E Idrobo, J Sunseri, DR Koes. J. Chem. Inf. Model, 2017
(3)Three-Dimensional Convolutional Neural Networks and a Cross-Docked Data Set for Structure-Based Drug Design
PG Francoeur, T Masuda, J Sunseri, A Jia, RB Iovanisci, I Snyder, DR Koes. J. Chem. Inf. Model, 2020
(4)Virtual Screening with Gnina 1.0 J Sunseri, DR Koes D. Molecules, 2021