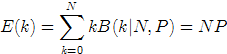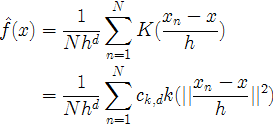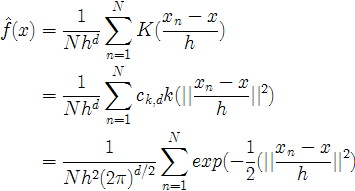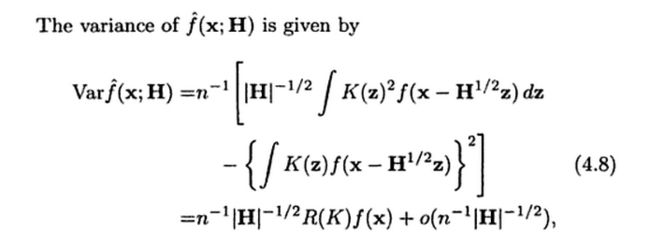mean shift 图像分割(三)
Reference:
[1] Mean shift: A robust approach toward feature space analysis, PAMI, 2002
[2] mean shift,非常好的ppt ,百度文库链接
[3] Pattern Recognition and Machine Learning, Bishop, 2006,Sec 2.5
[4] Computer Vision Algorithms and Applications, Richard Szeliski, 2010, Sec 5.3
[5] Kernel smoothing,MP Wand, MC Jones ,1994, Chapter 4
mean shift 图像分割 (一): 1 总体思想,2 算法步骤
mean shift 图像分割 (二): 3 算法原理,4 延伸
mean shift 图像分割 (三): 5 非参数密度估计
图像分割—mean shift(OpenCV源码注解)
5 非参数密度估计
这一部分说明为什么![]() 处的密度估计
处的密度估计![]() :
:
![]()
其实,我觉得看bishop的那本书[2]就可以了,行云流水,精彩绝伦,其实,这本书的大部分内容都是如此精彩。我是按自己的理解写的,有些地方有改动,也会有错误,望各位看官指正。
如果产生数据的分布形式已知,参数也已知,那么概率密度函数PDF已知,可以直接计算每一点的概率密度,比如高斯分布。如果参数不知道,那么也可以用数据估计参数,比如最小二乘估计,最大似然估计,贝叶斯参数估计等,如果连产生数据的分布形式都不知道,怎么办求概率密度呢?这就是一个非参数问题了,方法:让数据说话。
5.1 猜一下
对于上图中2维的情况,要估计蓝色圆域的概率密度,我相信大多数人都能凭直觉想到一种方法,那就用蓝色圈圈内的数据点个数![]() ,除以总的数据点个数
,除以总的数据点个数![]() ,即
,即![]() 。如果圆圈足够小,那么蓝色圈圈内部的概率密度就可以看成近似相等,那么蓝色点的概率密度应该是,
。如果圆圈足够小,那么蓝色圈圈内部的概率密度就可以看成近似相等,那么蓝色点的概率密度应该是,![]() 是蓝色圈圈的面积。当然,也可以推广到
是蓝色圈圈的面积。当然,也可以推广到![]() 维空间。这种算法,虽然直观,但缺乏理论支撑,下面证明,大伙的确猜对了。
维空间。这种算法,虽然直观,但缺乏理论支撑,下面证明,大伙的确猜对了。
5.2理论推导
首先说下,为什么![]() 可以用
可以用![]() 估计。
估计。
设是一个维的数据,密度函数为,则![]() 空间中的一个区域
空间中的一个区域![]() 的概率密度,即数据点落在区域
的概率密度,即数据点落在区域![]() 的概率:
的概率:
![]()
现在假设,依据某种未知概率分布得到了N个数据点(非参数并不是无法参数化,理论上任何分布都可以参数化,毕达哥拉斯说"万物皆数",只是参数无限维,只能当做非参数处理),则落在![]() 中的点的个数可能是
中的点的个数可能是![]() ,是否落在区域
,是否落在区域![]() 中就是一个二项分布:
中就是一个二项分布:
![]()
二项分布的期望:
二项分布的方差:
![]()
![]()
当![]() 时,
时,![]() ,从参数估计角度说,前者说明
,从参数估计角度说,前者说明![]() 是的无偏估计,后者说明是
是的无偏估计,后者说明是![]() 的一致估计。总之,说明
的一致估计。总之,说明![]() ,是一个很好的估计量。
,是一个很好的估计量。
因此,
![]()
进一步假设,比较小,那么![]() 内的
内的![]() 可近似相等,于是:
可近似相等,于是:
![]() 为
为![]() 的体积
的体积
![]()
注意:是有偏估计,下面再说。
由此推出,估计![]() ,有两种方法,第一种是固定
,有两种方法,第一种是固定![]() 看的数目,这就是kernel估计的本质(个人认为,直方图估计,Parzen windows 也是)。另外一种方法是固定
看的数目,这就是kernel估计的本质(个人认为,直方图估计,Parzen windows 也是)。另外一种方法是固定![]() 看包含
看包含![]() 个数据点所需要的体积,这就是K最近邻估计。
个数据点所需要的体积,这就是K最近邻估计。
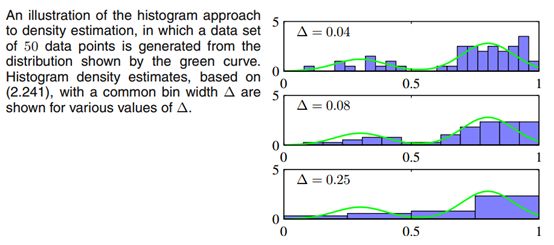
5.3直方图密度估计
将数据范围划分为若干个宽度为的小栅格(bin)(也可以不等长哦),然后统计落在每个区间内的数据点个数,那么,每个区间的密度,为整个数据范围内的数据点个数。
这个方法有很多缺陷:
(1)第一个bin起始位置的选择会影响到结果(与bin的个数无关)
(2)估计出来的概率密度有好多毛刺,不是连续光滑的曲线。
(3)适合一两维的情况。维是需要的bin个数为(假设每一维都需要划分成![]() 个bin),而且大多数bin的值为0,造成维度灾难(Curse of dimensionality)
个bin),而且大多数bin的值为0,造成维度灾难(Curse of dimensionality)
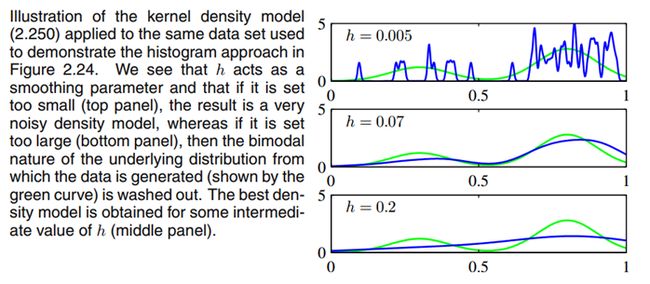
此外,对![]() 的大小特别敏感,小了,过拟合,不光滑,大了,太光滑,不过这是参数估计的普遍现象,前面提到的也是如此。
的大小特别敏感,小了,过拟合,不光滑,大了,太光滑,不过这是参数估计的普遍现象,前面提到的也是如此。
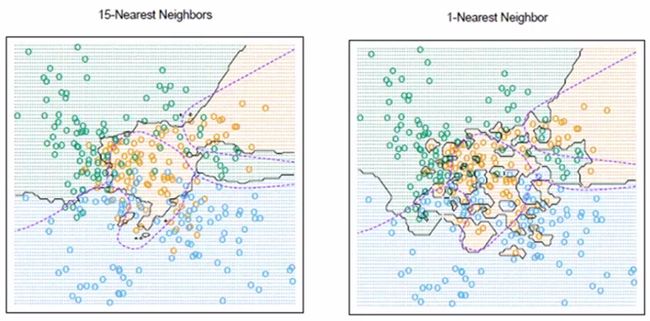
5.4 K近邻密度估计(K-nearest neighbours,KNN)
上面已经提过,,固定![]() ,看需要多大的
,看需要多大的![]() 。
。
这里我们用KNN密度估计+贝叶斯 推导下KNN分类器的原理。至于怎么分类的,很简单,如果不知道的话,哈哈,看我以前写的KNN (Related部分)。
样本![]() 属于哪一类就看它属于哪一类的可能性最大,即:
属于哪一类就看它属于哪一类的可能性最大,即:
![]()
![]() 很简单,基本的先验概率转后验概率:
很简单,基本的先验概率转后验概率:
![]()
利用上面的结论,则![]() ,,
,,![]()
所以,比较属于哪一类时,很公正,先在训练集中找K个最近的数据点,哪一类人多势众,测试样本就属于哪一类。
3类的情况
5.5 核密度估计(kernel density estimation, KDE)
5.5.1 Parzen windows
![]() 点处的密度估计值
点处的密度估计值![]() ,
,![]() 为落在以为中心的超球体的数据点个数。这与我们最开始猜测时的思想一致,只不过将超球体,换成超立方体。下面用数学符号形式化表示一下:
为落在以为中心的超球体的数据点个数。这与我们最开始猜测时的思想一致,只不过将超球体,换成超立方体。下面用数学符号形式化表示一下:
好了,我们用核函数的形式表示了![]() ,这里
,这里![]() ,
,![]() ,
,![]() 为总的样本数。这种方法本质上和直方图方法没有太大的区别,Parzen windows方法是以数据点为中心,而直方图是我们自己固定好的点为中心。因此,它也会有直方图的一些缺点。比如估计的概率密度不是连续,维度灾难。
为总的样本数。这种方法本质上和直方图方法没有太大的区别,Parzen windows方法是以数据点为中心,而直方图是我们自己固定好的点为中心。因此,它也会有直方图的一些缺点。比如估计的概率密度不是连续,维度灾难。
5.5.2 Kernel smoothing
很自然的,如果利用的数据量越大,估计出来的值就会越好,因为,我们综合的信息越多,于是我们使用所有数据点估计。采用所有样本估计的话,自然得要用加权的方法,越靠近估计点的数据点权重越大,反之,越是远离数据点,权重越小。
前面已经介绍过具有这样属性的两种核函数。Epanechnikov Kernel和 Normal Kernel。我们可以直接替换掉,则:
由于这两个核函数都是径向对称(radially symmetric),所以稍作了变化。
一开始,我并不理解为什么可以这么做,因为这样就已经不是窗口内的数据点个数,而是所有数据点都参合进来了,意义已经不一样了。后面我们可以通过求它的期望来进一步说明。
此外,bishop说,这个式子既可以看成,只有一个以![]() 为中心的窗口,也可以看成
为中心的窗口,也可以看成![]() 个以
个以![]() 为中心的窗口,后一种介绍,我一直理解不了,但是,原文都是
为中心的窗口,后一种介绍,我一直理解不了,但是,原文都是![]() 而不是
而不是![]() ,所以应该是第二种解释,才会这样写,我觉得第一种解释挺好,所以我都换过来了。
,所以应该是第二种解释,才会这样写,我觉得第一种解释挺好,所以我都换过来了。
比如,我们使用高斯核,就有:
注意:在靠近左边/右边的估计值有很大偏差,这是因为数据不对称,所以主要以右边/左边的数据为主,如果是回归就不会参数这种现象了。
下面啰嗦一下上式中的![]()
![]()
而,所以。至于为什么要保证![]() ,下面就会知道了。
,下面就会知道了。
现在看看估计值的期望
我们先做一次变量替换,。
假设足够光滑,各阶导数都存在,我们在对![]() 在
在![]() 处泰勒展开一下:
处泰勒展开一下:
这里只推导1维的情况,![]() 维太复杂了……
维太复杂了……
注意:无穷小项![]() 直接被我忽略了。
直接被我忽略了。
第一项当然希望等于![]() ,于是我们就希望,得到
,于是我们就希望,得到![]() 第一个条件。不过,对于模式搜索来说,
第一个条件。不过,对于模式搜索来说,![]() 都可以,只要不影响到我们比较大小就好。
都可以,只要不影响到我们比较大小就好。
第二项,等于0最好,所以我们希望![]()
第三项,不能发散,所以![]() 还得满足第三个条件:,原文还提到一个条件:,这个条件怎么来的,还没想清楚,很多论文也不提这个条件。
还得满足第三个条件:,原文还提到一个条件:,这个条件怎么来的,还没想清楚,很多论文也不提这个条件。
显然这是一个有偏估计,偏差为。
方差:
因此,要使得期望很小,则要很小,要使方差很小则![]() 要很大。
要很大。
书上的多维推导过程,复杂,矩阵知识严重不够用。
其中:![]() ,为了简便,我上面都是
,为了简便,我上面都是![]() (图像分割中,一般也是如此),
(图像分割中,一般也是如此),![]() 用来控制核函数的形状和方向,比如我们可以将高斯核改成椭圆形状。
用来控制核函数的形状和方向,比如我们可以将高斯核改成椭圆形状。
这里岔开一下,扯一扯目标检测。比如我们要检测图像中的椭圆形物体,用两高斯核作差,得到一个DoG(类似于墨西哥草帽),让它和图像卷积。控制它的形状和方向就能使得特定形状和方向的目标的响应值最大(和卷积核越像的区域其滤波响应值越大),从而能得到一张该目标在任何一点出现的概率图。接下来用mean shift 作模点搜索,这应该就是mean shift用于目标检测的基本原理吧,待验证。
记录几个公式:
,![]() 是方阵
是方阵
![]() ,是缩写……
,是缩写……
![]()
5.5.3直方图估计的kernel 平滑版
参见:《Density Estimation》 Simon J. Sheather, Statistical Science 2004
木有仔细看……
如果假设数据服从正态分布,那么就有最优带宽,还有好多种……
normal reference bandwidth:
![]()
oversmoothed bandwidth
![]()
![]() 数据的标准差,
数据的标准差,
![]() :数据的个数,但是我以前看《Fast Object Detection with Entropy-Driven Evaluation》源码,用的是
:数据的个数,但是我以前看《Fast Object Detection with Entropy-Driven Evaluation》源码,用的是![]() , 它的
, 它的![]() 并不是指实际的数据,它是去掉重复后的数据,但是它论文上还是说就是样本的数目,为什么呢?
并不是指实际的数据,它是去掉重复后的数据,但是它论文上还是说就是样本的数目,为什么呢?
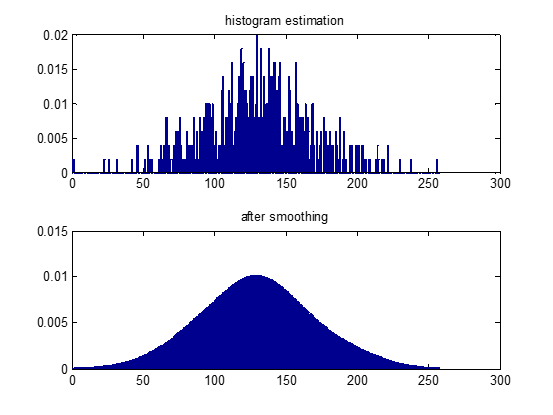
这个用得比较多,我截取了这篇论文的部分代码,做了个小实验,……
matlab代码
close all
ri=round (randn(500,1)*100+50);
nb_UniQueD=numel(unique(ri));
minScore = min(ri(:))-1;
maxScore = max(ri(:))+1;
scoreStd = std(ri);
sigma = 1.44 * scoreStd * nb_UniQueD^(-1/5); % not the number of sample
% sigma = 1.06 * scoreStd * numel(ri)^(-1/5); % not the number of sample
numBins = min(256,10*nb_UniQueD/2);
Sp = linspace(minScore, maxScore, numBins+1);% need to add one
H = histc(ri, Sp);
% normalize by number of samples
Hraw = H / sum(H);
figure, subplot(211);
bar(Hraw);title('histogram estimation')
% discretization factor
discrFactor = (maxScore - minScore) / numBins;
kerSize = round(5 * sigma / discrFactor);
if kerSize(1) < 3
kerSize(1) = 3.0;
end
kerSize = double(kerSize);
% apply parzen window, kernel size such that it gets to 2 sigma
K = fspecial('gaussian', [kerSize 1], double(sigma/discrFactor) );
H = conv( Hraw, K, 'same' );
H = H + 1e-10;
H = H ./sum(H);
subplot(212),bar(H);title('after smooth')