Kmeans聚类Python程序
之前写过两个关于kmeans的博客
kmeans理论介绍
kmeans与dbscan的对比
再说下算法的过程:
1.随机的选取k个聚类中心,这个k是有自己设置的
2.计算数据集到k个聚类中心的距离
3.对一条数据,分配到距k个聚类中心最近的类中
4.得到新的k个数据,计算聚类中心
5.更新2,3,4当类别不再变化时结束迭代
Python程序
计算欧式距离
# 计算欧式距离
def distEclud(vecA,vecB):
return sqrt(sum(power(vecA-vecB,2)))随机的选取初始类中心
# 随机的参数k个类中心
def randCent(dataSet,k):
n = shape(dataSet)[1]
centroids = mat(zeros((k,n)))
# 中心是在每一维的最大值和最小值之间随机选取
for j in range(n):
minJ = min(dataSet[:,j])
rangeJ = float(max(dataSet[:,j]) - minJ)
centroids[:,j] = minJ + rangeJ * random.rand(k,1)
return centroidskmeans算法过程
# 聚类过程 根据欧式距离判定数据所在的类,距离越近越好
def kMeans0(dataSet,k,distMeas = distEclud,creatCent = randCent):
# m个样本,n个特征
m,n = shape(dataSet)
print m,n
# 记录每个数据的类别,以及到所在类别中心的距离
clusterAssment = mat(zeros((m,2)))
centroids = creatCent(dataSet,k)
clusterChanged = True
# 当类别不在变化时候停止迭代
while clusterChanged:
clusterChanged = False
# 遍历所有的样本数据
for i in range(m):
minDist = inf
minIndex = -1
# 计算 第i条样本到k个类中心的距离
# 以最小的类别,更新该样本所在的类别
for j in range(k):
disJI = distMeas(centroids[j,:],dataSet[i,:])
if disJI < minDist:
minDist = disJI
minIndex = j
if clusterAssment[i,0] != minIndex:
clusterChanged = True
clusterAssment[i,:] = minIndex ,minDist
# print centroids
# 计算结束的k个类的类中心
for cent in range(k):
ptInClust = dataSet[nonzero(clusterAssment[:,0].A==cent)[0]]
centroids[cent,:] = mean(ptInClust,axis=0)
return centroids,clusterAssment
缺点:
1.随机的选取类中心,可能影响聚类的效果,这个博客有详解
2.每次计算的都是欧式距离,于是乎,可以更换其他距离的公式试试效果如何
下面采用的是余弦相识度类来计算一个样本到类中心的距离。
余弦相识度计算公式:
这其实就是两个向量的夹角的余弦值,在[-1.1]之间,至于如何衡量距离的好坏,可以认为在1 和-1附近是好的,也可以认为是1附近是最好的,毕竟在-1附近是向量方向相反了。
更改上面的程序,认为余弦相识度越接近1认为越可能是同一类。
Python程序:
# 计算余弦相识度
def cosineSimilarity(vecA,vecB):
vecA = array(vecA)
vecB = array(vecB)
a = sum(vecA*vecB)
b = sqrt(sum(vecA*vecA))
c = sqrt(sum(vecB*vecB))
if b!=0 and c!=0:
return a/(b*c)
if b!=0 and c==0 :
return a/(b*0.001)
if b==0 and c!=0:
return a/(c*0.001)上面当两个向量中有一个模为0的时候认为是0.01
聚类程序:
# 聚类过程 根据余弦相识度判定 数据与类中心相识度越高 作为所在的类
def kMeans1(dataSet,k,distMeas = cosineSimilarity,creatCent = randCent):
m,n = shape(dataSet)
print m,n
# 记录每个数据的类别,以及到所在类别中心的距离
clusterAssment = mat(zeros((m,2)))
centroids = creatCent(dataSet,k)
clusterChanged = True
# 当类别不在变化时候停止迭代
while clusterChanged:
clusterChanged = False
# 遍历所有的样本数据
for i in range(m):
maxDist = -1
maxIndex = -1
# 计算 第i条样本到k个类中心的距离
# 以最小的类别,更新该样本所在的类别
for j in range(k):
disJI = distMeas(centroids[j,:],dataSet[i,:])
if disJI > maxDist:
maxDist = disJI
maxIndex = j
if clusterAssment[i,0] != maxIndex:
clusterChanged = True
clusterAssment[i,:] = maxIndex ,maxDist
# print centroids
# 计算结果的k个类的类中心
for cent in range(k):
ptInClust = dataSet[nonzero(clusterAssment[:,0].A==cent)[0]]
centroids[cent,:] = mean(ptInClust,axis=0)
return centroids,clusterAssment
载入数据:
# -*- coding: utf-8 -*-
from numpy import *
import sys
sys.setrecursionlimit(5000) #例如这里设置为一百万
def loadDataSet(filename):
dataMat=[]
fr = open(filename)
for line in fr.readlines():
curLine = line.strip().split('\t')
fltLine = map(float,curLine)
dataMat.append(fltLine)
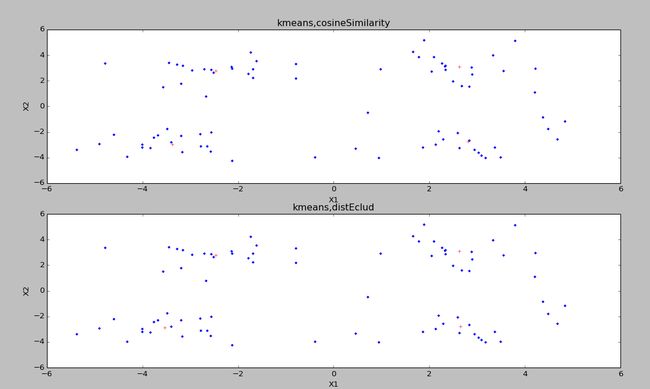
return mat(dataMat)这是最好的聚类结果,运行多长发现,根据余弦相识度的聚类也可能出现不好的结果,但是整体比欧拉距离的好点。
画图程序:
def plotKmeans(k):
import matplotlib.pyplot as plt
dataSet = loadDataSet('testSet.txt')
# 余弦相识度
center1,cluserAssment = kMeans1(dataSet,k,distMeas = cosineSimilarity,creatCent = randCent)
print center1
plt.figure(1)
plt.subplot(211)
plt.plot(array(dataSet[:,0]),array(dataSet[:,1]),'.')
plt.plot(array(center1[:,0]),array(center1[:,1]), 'r+')
# plt.annotate('local max', xy=(2, 1), xytext=(3, 1.5),
# arrowprops=dict(facecolor='red', shrink=0.05),)
plt.xlabel('X1')
plt.ylabel('X2')
plt.title('kmeans,cosineSimilarity')
plt.subplot(212)
# 欧拉距离
center0,cluserAssment = kMeans0(dataSet,k,distMeas = distEclud,creatCent = randCent)
print center0
plt.plot(array(dataSet[:,0]),array(dataSet[:,1]),'.')
plt.plot(array(center0[:,0]),array(center0[:,1]), 'r+')
plt.xlabel('X1')
plt.ylabel('X2')
plt.title('kmeans,distEclud')
plt.show()