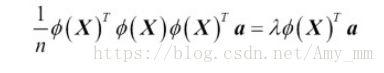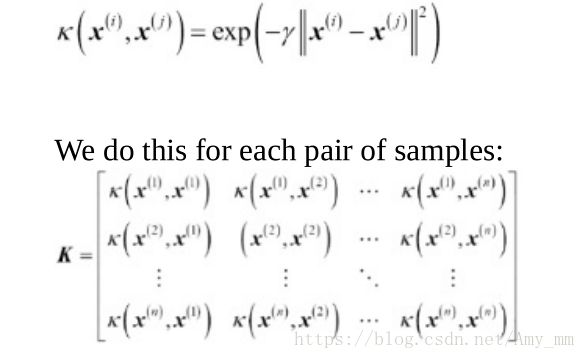数据压缩之降维(三)——KPDA
学习《python machine learning》chapter5——Compressing data via dimensionality reduction
PCA链接 https://blog.csdn.net/Amy_mm/article/details/79812241
主要内容如下:
(1)主要成分分析 Principal Component Analysis (PCA) ——非监督学习
(2)线性判别分析 Linear Discriminant Analysis (LDA) ——监督学习
(3)核主成分分析 Kernel Principal Component Analysis ——非线性降维
源码 git 地址:https://github.com/xuman-Amy/compressing-data
对于非线性数据的降维有以下方法:
方法一:首先将非线性数据映射到一个新的高维特征空间,然后运用标准PCA对数据进行降维,从而将数据集转换为线性可分的数据集。
但是这样不高效,computationally very expensive
方法二就是KPDA~~
KPDA可以计算高维数据集中两个特征向量在原始数据集中的相似性。
【KPDA】
定义映射函数
在PCA中计算两个特征值的协方差如下:
因为数据标准化后,均值为0,所以公式简化为
一般的协方差计算公式:
计算非线性数据集的协方差矩阵,将样本特征向量换成经过![]() 函数转换的特征向量:
函数转换的特征向量:
![]() 为特征值,v为特征向量,a可以通过从kernel 矩阵K中提取特征向量得到
为特征值,v为特征向量,a可以通过从kernel 矩阵K中提取特征向量得到
【kernel matrix】
其中,![]() 为n*k维矩阵
为n*k维矩阵
等式可以写成:
RBF kernel (Radial Basis Function)也称为Gaussian kernel
计算RBF Kernel PCA步骤:
(1)计算kernel 矩阵K
(2)中心化K矩阵
![]() 为n*n的矩阵与K矩阵同维度,每个值都是1/n
为n*n的矩阵与K矩阵同维度,每个值都是1/n
(3)区top k 个向量对应top k 大的特征值,进行降序排列
【python 实现KPCA】
from scipy.spatial.distance import pdist, squareform#pdist 计算成对距离,squareform 转换为方阵形式
from scipy import exp
from scipy.linalg import eigh #Return the eigenvalues and eigenvectors of a Hermitian or symmetric matrix.(by ascending)
import numpy as np
def rbf_kernel_pca(X, gamma, n_components):
"""
RBF kernel PCA implementation.
Parameters
------------
X: {NumPy ndarray}, shape = [n_samples, n_features]
gamma: float
Tuning parameter of the RBF kernel
n_components: int
Number of principal components to return
Returns
------------
X_pc: {NumPy ndarray}, shape = [n_samples, k_features]
Projected dataset
"""
#计算M*N矩阵中两两向量的平方欧几里得距离
sq_dists = pdist(X, 'sqeuclidean')
#将距离转换为方阵
mat_sq_dists = squareform(sq_dists)
#计算对称kernel 矩阵
K = exp(- gamma * mat_sq_dists)
# Center the kernel matrix
N = K.shape[0]
one_n = np.ones((N, N)) / N
K = K - one_n.dot(K) - K.dot(one_n) + one_n.dot(K).dot(one_n)
#提取 K 的向量和特征值
eigvals, eigvecs = eigh(K)
eigvals, eigvecs = eigvals[::-1], eigvecs [::-1]
#选择top K 向量
X_pc = np.column_stack(eigenvecs[:,i]
for i in range(n_components))
return X_pc
【examples ——half moons】
import half moon 数据集,非线性数据集
# half moons datasets
from sklearn.datasets import make_moons
import matplotlib.pyplot as plt
X, y = make_moons(n_samples = 100, random_state = 123)
plt.scatter(X[y == 0, 0], X[y == 0, 1], color='red', marker='^', alpha=0.5)
plt.scatter(X[y == 1, 0], X[y == 1, 1], color='blue', marker='o', alpha=0.5)
plt.tight_layout()
plt.show()
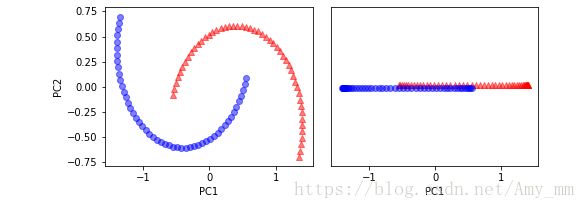
【pca对于half moons 数据的作用】
看下PCA对于非线性数据集的效果
#看下PCA的效果
from sklearn.decomposition import PCA
pca = PCA(n_components = 2)
X_spca = pca.fit_transform(X)
fig, ax = plt.subplots(nrows=1,ncols=2, figsize=(7,3))
ax[0].scatter(X_spca[y == 0, 0], X_spca[y == 0, 1],
color='red', marker='^', alpha=0.5)
ax[0].scatter(X_spca[y == 1, 0], X_spca[y == 1, 1],
color='blue', marker='o', alpha=0.5)
ax[1].scatter(X_spca[y == 0, 0], np.zeros((50, 1)) + 0.02,
color='red', marker='^', alpha=0.5)
ax[1].scatter(X_spca[y == 1, 0], np.zeros((50, 1)) - 0.02,
color='blue', marker='o', alpha=0.5)
ax[0].set_xlabel('PC1')
ax[0].set_ylabel('PC2')
ax[1].set_ylim([-1, 1])
ax[1].set_yticks([])
ax[1].set_xlabel('PC1')
plt.tight_layout()
# plt.savefig('images/05_13.png', dpi=300)
plt.show()显然,线性分类器不能将PCA后的数据进行很好的分类
【Kpca对于half moons 的作用】
看下Kernel PCA 的效果
# kpca
X_kpca = rbf_kernel_pca(X, gamma = 15, n_components = 2)
fig, ax = plt.subplots(nrows=1,ncols=2, figsize=(7,3))
ax[0].scatter(X_kpca[y==0, 0], X_kpca[y==0, 1],
color='red', marker='^', alpha=0.5)
ax[0].scatter(X_kpca[y==1, 0], X_kpca[y==1, 1],
color='blue', marker='o', alpha=0.5)
ax[1].scatter(X_kpca[y==0, 0], np.zeros((50,1))+0.02,
color='red', marker='^', alpha=0.5)
ax[1].scatter(X_kpca[y==1, 0], np.zeros((50,1))-0.02,
color='blue', marker='o', alpha=0.5)
ax[0].set_xlabel('PC1')
ax[0].set_ylabel('PC2')
ax[1].set_ylim([-1, 1])
ax[1].set_yticks([])
ax[1].set_xlabel('PC1')
plt.tight_layout()
plt.show()
【examples 2——concentric circle】
import非线性数据集
#concentric cirles 同心圆
from sklearn.datasets import make_circles
# factor内部圆的比例 noise 高斯噪声
X, y = make_circles(n_samples = 1000, random_state = 123, noise = 0.1, factor = 0.2)
plt.scatter(X[y == 0, 0], X[y == 0, 1], color='red', marker='^', alpha=0.5)
plt.scatter(X[y == 1, 0], X[y == 1, 1], color='blue', marker='o', alpha=0.5)
plt.tight_layout()
plt.show()
【pca对于同心圆的作用】
#看下PCA的效果
from sklearn.decomposition import PCA
pca = PCA(n_components = 2)
X_spca = pca.fit_transform(X)
fig, ax = plt.subplots(nrows=1,ncols=2, figsize=(7,3))
ax[0].scatter(X_spca[y == 0, 0], X_spca[y == 0, 1],
color='red', marker='^', alpha=0.5)
ax[0].scatter(X_spca[y == 1, 0], X_spca[y == 1, 1],
color='blue', marker='o', alpha=0.5)
ax[1].scatter(X_spca[y == 0, 0], np.zeros((500, 1)) + 0.02,
color='red', marker='^', alpha=0.5)
ax[1].scatter(X_spca[y == 1, 0], np.zeros((500, 1)) - 0.02,
color='blue', marker='o', alpha=0.5)
ax[0].set_xlabel('PC1')
ax[0].set_ylabel('PC2')
ax[1].set_ylim([-1, 1])
ax[1].set_yticks([])
ax[1].set_xlabel('PC1')
plt.tight_layout()
# plt.savefig('images/05_13.png', dpi=300)
plt.show()显然不能用线性分类器进行数据分类
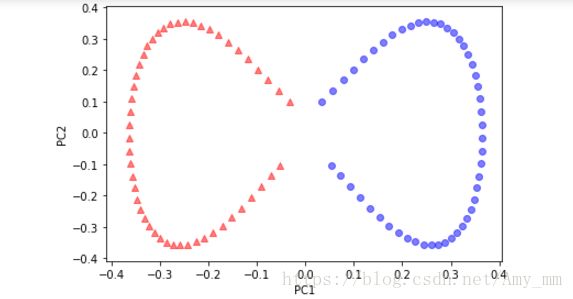
【Kpca对于同心圆的作用】
# kpca
X_kpca = rbf_kernel_pca(X, gamma = 15, n_components = 2)
fig, ax = plt.subplots(nrows=1,ncols=2, figsize=(7,3))
ax[0].scatter(X_kpca[y==0, 0], X_kpca[y==0, 1],
color='red', marker='^', alpha=0.5)
ax[0].scatter(X_kpca[y==1, 0], X_kpca[y==1, 1],
color='blue', marker='o', alpha=0.5)
ax[1].scatter(X_kpca[y==0, 0], np.zeros((500,1))+0.02,
color='red', marker='^', alpha=0.5)
ax[1].scatter(X_kpca[y==1, 0], np.zeros((500,1))-0.02,
color='blue', marker='o', alpha=0.5)
ax[0].set_xlabel('PC1')
ax[0].set_ylabel('PC2')
ax[1].set_ylim([-1, 1])
ax[1].set_yticks([])
ax[1].set_xlabel('PC1')
plt.tight_layout()
plt.show()【sklearn 实现Kpca】
from sklearn.decomposition import KernelPCA
kpca = KernelPCA(n_components = 2, kernel = 'rbf', gamma = 15)
X, y = make_moons(n_samples = 100, random_state = 123)
X_kpca =kpca.fit_transform(X)
plt.scatter(X_kpca[y == 0, 0], X_kpca[y == 0, 1],
color='red', marker='^', alpha=0.5)
plt.scatter(X_kpca[y == 1, 0], X_kpca[y == 1, 1],
color='blue', marker='o', alpha=0.5)
plt.xlabel('PC1')
plt.ylabel('PC2')
plt.tight_layout()
plt.show()