- 【LangChain编程:从入门到实践】LangChain与其他框架的比较
AI天才研究院
AgenticAI实战计算AI人工智能与大数据计算科学神经计算深度学习神经网络大数据人工智能大型语言模型AIAGILLMJavaPython架构设计AgentRPA
【LangChain编程:从入门到实践】LangChain与其他框架的比较1.背景介绍1.1人工智能发展现状在当今时代,人工智能(AI)已经成为科技领域中最热门和最具革命性的话题之一。随着计算能力的不断提升和算法的持续优化,AI系统正在不断扩展其应用范围,包括自然语言处理、计算机视觉、决策系统等各个领域。1.2LangChain概述在这种背景下,LangChain作为一个新兴的AI框架应运而生。L
- happy-llm 第一章 NLP 基础概念
weixin_38374194
自然语言处理人工智能学习
文章目录一、什么是NLP?二、NLP发展三大阶段三、NLP核心任务精要四、文本表示演进史1.传统方法:统计表征2.神经网络:语义向量化课程地址:happy-llmNLP基础概念一、什么是NLP?核心目标:让计算机理解、生成、处理人类语言,实现人机自然交互。现状与挑战:成就:深度学习推动文本分类、翻译等任务达到近人类水平。瓶颈:歧义性、隐喻理解、跨文化差异等。二、NLP发展三大阶段时期代表技术核心思
- Nystromformer:一种基于 Nyström 方法的自注意力近似算法
AI专题精讲
Paper阅读人工智能自然语言处理AI
1.摘要Transformer已经成为广泛自然语言处理任务中的强大工具。推动Transformer展现出卓越性能的一个关键组件是self-attention机制,它对每个token编码了其他token的影响或依赖关系。虽然self-attention机制具有诸多优势,但其在输入序列长度上的二次复杂度限制了其在较长序列上的应用——这是当前社区积极研究的一个主题。为了解决这一限制,我们提出了Nystr
- 供应链风险管理:AI预测潜在风险
AI智能应用
AI大模型应用入门实战与进阶javapythonjavascriptkotlingolang架构人工智能
供应链风险管理,AI预测,机器学习,深度学习,自然语言处理,风险评估,供应链可视化1.背景介绍在当今全球化经济体系中,供应链的复杂性和脆弱性日益凸显。供应链风险是指任何可能对供应链正常运行造成负面影响的事件或因素。这些风险可能来自自然灾害、政治动荡、经济波动、技术故障、供应商违约等方面。一旦供应链风险爆发,可能会导致生产中断、产品短缺、成本飙升、品牌形象受损等严重后果。传统供应链风险管理方法主要依
- 供应链风险管理:AI如何预测供应链风险
AI大模型应用之禅
javapythonjavascriptkotlingolang架构人工智能
供应链风险管理,AI预测,机器学习,深度学习,自然语言处理,时间序列分析,风险评估1.背景介绍在当今全球化经济体系中,供应链风险已成为企业面临的重大挑战。供应链的复杂性和不可预测性使得企业更容易受到各种风险的影响,例如自然灾害、政治动荡、经济波动、疫情爆发等。这些风险可能导致供应中断、成本增加、交付延迟,甚至损害企业声誉。传统供应链风险管理方法主要依赖于经验和专家判断,缺乏数据驱动和预测能力。随着
- 【AI】大语言模型(LLM)& NLP
G皮T
#大语言模型LLMNLP大模型大语言模型AI人工智能
大语言模型(LLM)&NLP1.大语言模型(LLM)1.1一句话解释1.2更形象的比喻1.3为什么叫“大”模型1.4它能做什么1.5现实中的例子2.对比NLP2.1用“汽车进化”比喻NLP→LLM2.2为什么说LLM属于NLP2.3LLM的“革命性突破”在哪里2.4总结1.大语言模型(LLM)1.1一句话解释大语言模型(LargeLanguageModel,LLM)是一个“超级文字预测器”,它通过
- 使用Python爬虫与自然语言处理技术抓取并分析网页内容
Python爬虫项目
python爬虫自然语言处理javascript数据分析人工智能
1.引言在如今数据驱动的时代,网页爬虫(WebScraping)和自然语言处理(NLP)已成为处理大量网页数据的重要工具。利用Python爬虫抓取网页内容,结合NLP技术进行文本分析和信息抽取,能够从大量网页中提取有价值的信息。无论是新闻文章的情感分析、社交媒体的舆情分析,还是电商网站的商品评论挖掘,这些技术都发挥着至关重要的作用。本文将介绍如何利用Python爬虫与自然语言处理技术抓取并分析网页
- GPT在AI原生应用领域的无限潜力
GPT在AI原生应用领域的无限潜力关键词:GPT、AI原生应用、自然语言处理、无限潜力、应用场景摘要:本文深入探讨了GPT在AI原生应用领域所展现出的无限潜力。首先介绍了相关背景知识,包括GPT的基本概念和AI原生应用的定义。接着详细解释了GPT的核心概念,以及它与AI原生应用的紧密联系。通过数学模型和公式对GPT的工作原理进行了阐述,并给出了实际的代码案例。还探讨了GPT在多个实际应用场景中的表
- 深度学习前置知识全面解析:从机器学习到深度学习的进阶之路
一、引言:人工智能时代的核心技术在当今这个数据爆炸的时代,人工智能(AI)已经成为推动社会进步的核心技术之一。作为AI领域最重要的分支,深度学习(DeepLearning)在计算机视觉、自然语言处理、语音识别等领域取得了突破性进展,彻底改变了我们与机器交互的方式。本教案将从机器学习的基础知识出发,系统性地介绍深度学习的核心概念、数学基础、网络架构和训练方法,为读者构建完整的知识体系框架。无论你是刚
- 计算机视觉中的Transformer:ViT模型详解与代码实现
AI大模型应用工坊
计算机视觉transformer人工智能ai
计算机视觉中的Transformer:ViT模型详解与代码实现关键词:计算机视觉、Transformer、ViT、自注意力机制、图像分块摘要:传统卷积神经网络(CNN)统治计算机视觉领域多年,但2020年一篇《AnImageisWorth16x16Words:TransformersforImageRecognitionatScale》的论文打破了这一格局——它将NLP领域的Transformer
- AI 加持下的智能家居行业:变革、挑战与机遇
低代码老李
人工智能智能家居
在当今科技迅猛发展的浪潮中,人工智能(AI)已深深融入智能家居领域,成为推动其蓬勃发展的关键力量,为人们的生活带来了诸多便利和创新体验,同时也面临着一系列亟待解决的问题。一、AI驱动的智能家居功能升级(1)智能语音交互与控制智能语音助手作为智能家居的核心交互方式,借助自然语言处理(NLP)技术,让用户仅通过简单的语音指令,就能轻松操控家中各类智能设备,如精准控制灯光的开关与亮度调节、窗帘的开合、电
- 视觉表征和多模态融合
一只齐刘海的猫
语言模型
视觉表征和多模态融合是当前人工智能领域的研究热点,特别是在计算机视觉和自然语言处理的交叉领域。视觉表征是指将图像或视频信息转化为模型可以处理的向量形式,而多模态融合则是将不同类型的数据(如视觉、文本、音频等)进行整合,以实现更全面、准确的信息理解和处理。视觉表征(VisualRepresentation)目的:将图像或视频数据转化为深度学习模型可以理解的特征向量。方法:卷积神经网络(CNN):传
- NLTK库全解析:用Python打开自然语言处理的第一把钥匙
引言你是否好奇过,手机里的智能助手是如何“听懂”你说的话?电商平台的差评分析又是怎样精准提取“物流慢”“质量差”这些关键词?这些看似神奇的自然语言处理(NLP)功能,背后都藏着一个“入门神器”——NLTK(NaturalLanguageToolkit)。作为Python生态中最经典的NLP库,NLTK就像一本“NLP百科全书”,从最基础的文本拆分到复杂的语义理解,它用简单的代码接口,带我们推开自然
- VLA模型
一介绍在机器人领域,视觉-语言-动作(VLA)模型的发展经历了显著的演变,这得益于计算机视觉和自然语言处理领域的进步。VLA模型代表了一类旨在处理多模态输入的模型,整合了来自视觉、语言和动作的信息。这些模型对于实现具身智能至关重要,使机器人能够理解物理世界并与之互动。以下是VLA模型发展的时间线:早期阶段:计算机视觉和自然语言处理的集成大约在2015年开始,随着视觉问答(VQA)系统的出现。这些系
- Python 爬虫实战:微博话题讨论数趋势爬取与分析全流程
西攻城狮北
python爬虫开发语言
1.项目背景与目标微博话题(#话题#)是社交媒体舆情监测、品牌营销、热点追踪的重要数据源。本实战要完成以下目标:爬取指定话题在7天内的讨论数、阅读量、热搜排名等关键指标。将数据存入MySQL,并每日增量更新。用Pandas+Matplotlib绘制趋势图,直观呈现热度变化。基于SnowNLP做情感倾向分析,输出正面/负面占比。生成一份可分享的HTML可视化报告。2.环境搭建与依赖2.1安装核心库p
- 循环神经网络(RNN):序列数据处理的强大工具
LNL13
rnn人工智能深度学习
在人工智能和机器学习的广阔领域中,处理和理解序列数据一直是一个重要且具有挑战性的任务。循环神经网络(RecurrentNeuralNetwork,RNN)作为一类专门设计用于处理序列数据的神经网络,在诸多领域展现出了强大的能力。从自然语言处理中的文本生成、机器翻译,到时间序列分析中的股票价格预测、天气预测等,RNN都发挥着关键作用。本文将深入探讨RNN的工作原理、架构特点、训练方法、常见类型以及其
- 大语言模型应用指南:Gemini简介
AI大模型应用之禅
人工智能数学基础计算科学神经计算深度学习神经网络大数据人工智能大型语言模型AIAGILLMJavaPython架构设计AgentRPA
1.背景介绍自然语言处理(NLP)一直是人工智能领域的热门话题之一。在NLP中,语言模型是一个重要的概念,它可以用来预测下一个单词或字符的概率。近年来,随着深度学习技术的发展,大型语言模型的研究和应用也越来越受到关注。其中,Gemini是一种新型的大型语言模型,它在多项任务上取得了优异的表现。本文将介绍Gemini的核心概念、算法原理、数学模型和公式、项目实践、实际应用场景、工具和资源推荐、未来发
- 深入了解Transformer模型及其优缺点
目录前言1Transformer结构特点1.1注意力机制(Self-Attention)1.2编码器-解码器架构1.3位置编码和基于注意力的损失函数2Transformer模型优缺点分析2.1Transformer模型的优点2.2Transformer模型的缺点3应用领域结语前言在当今人工智能领域,自然语言处理的关键问题之一是解决文本理解和生成中的挑战。传统的循环神经网络虽然在处理序列数据方面取得
- Anconda环境下Vscode安装Python
Java后时代
程序员python学习面试
最后Python崛起并且风靡,因为优点多、应用领域广、被大牛们认可。学习Python门槛很低,但它的晋级路线很多,通过它你能进入机器学习、数据挖掘、大数据,CS等更加高级的领域。Python可以做网络应用,可以做科学计算,数据分析,可以做网络爬虫,可以做机器学习、自然语言处理、可以写游戏、可以做桌面应用…Python可以做的很多,你需要学好基础,再选择明确的方向。这里给大家分享一份全套的Pytho
- 蒙特卡罗方法与深度学习的关系
AGI大模型与大数据研究院
AI大模型应用开发实战计算科学神经计算深度学习神经网络大数据人工智能大型语言模型AIAGILLMJavaPython架构设计AgentRPA
蒙特卡罗方法与深度学习的关系作者:禅与计算机程序设计艺术/ZenandtheArtofComputerProgramming1.背景介绍1.1问题的由来蒙特卡罗方法和深度学习都是近年来在计算科学和人工智能领域取得重大突破的技术。蒙特卡罗方法是一种基于随机抽样的数值计算方法,广泛应用于物理、工程、金融等领域。深度学习则是一种基于人工神经网络的学习方法,在图像识别、语音识别、自然语言处理等领域取得了显
- 新华妙笔:AI智能写作助手,让高效写作触手可及
东风西巷
AI写作android软件需求智能手机
在当今快节奏的时代,无论是职场人士、学生还是创作者,都面临着大量的写作任务。从工作总结、调研报告到公文写作、商业文案,高效且高质量的写作能力成为了提升个人竞争力的关键。然而,写作不仅需要丰富的知识储备和扎实的文字功底,还需要大量的时间和精力去打磨。为了帮助用户更高效地完成写作任务,新华妙笔APP应运而生。它是一款功能强大的AI智能写作助手,依托自然语言处理(NLP)和大数据分析技术,能够快速生成各
- Day44
1.预训练概念:在大规模数据上训练模型学习通用知识,再迁移到下游任务微调2.常见模型:图像有AlexNet、ResNet、ViT;NLP有BERT、GPT3.图像模型发展:从手工特征到深度学习,从CNN到Transformer、多模态4.预训练策略:数据增强、自监督/监督训练、模型微调、多模态学习作业1.importtorchimporttorch.nnasnnimporttorch.optima
- Python, Rust 开发机关事业单位公文写作助手APP
#机关事业单位公文写作助手应用设计基于Python和Rust开发机关事业单位公文写作APP的方案如下:##系统架构设计```桌面端/Web应用(Tauri/Vue.js)|RESTAPI/WebSocket|Rust核心服务(Actix-web/Axum)←───PythonNLP服务(FastAPI)|公文要素提取/模板生成/智能校对PostgreSQL数据库|Redis缓存(模板缓存/会话管理
- 【大模型面试】大模型Prompt Engineer面试题及参考答案
大模型知识
prompt人工智能开发语言pythonchatgpt深度学习大模型
一、基础概念类1.什么是大模型?大模型通常指具有庞大参数规模的机器学习模型,尤其是在自然语言处理(NLP)和计算机视觉等领域。这些模型能够学习到大量数据中的复杂模式和特征,具备强大的泛化能力,可在多种任务上表现出色,如GPT系列、BERT等。2.大模型与传统机器学习模型的区别是什么?传统机器学习模型参数规模相对较小,往往针对特定任务进行设计和训练,需要较多人工特征工程。而大模型参数数量庞大,通过在
- !LangChain链的并行执行与异步处理深度解析(32)
Android 小码蜂
测试专栏langchain
LangChain链的并行执行与异步处理深度解析一、LangChain链的基础概念与执行逻辑1.1LangChain链的定义与作用LangChain链(Chain)是LangChain框架的核心组件之一,它通过将多个组件(如提示模板、大语言模型、输出解析器等)串联起来,形成一个完整的处理流程,以实现特定的自然语言处理任务。例如,在问答系统中,链可以先使用提示模板构建问题,然后调用大语言模型生成回答
- AI产品经理技术篇:AI领域常用术语解析
让我看看好学吗
人工智能产品经理机器学习深度学习学习
作为AI产品经理,深入理解人工智能领域的核心术语是高效沟通、需求定义和产品落地的关键。无论是与算法工程师协作优化模型,还是向业务方解释技术方案,准确掌握专业术语能显著提升决策效率,避免因概念混淆导致的开发偏差。本文系统梳理了模型与算法、NLP(自然语言处理)、CV(计算机视觉)、数据处理、核心评估指标等领域的核心术语,帮助产品经理快速构建AI技术认知框架。目录1.基础概念2.模型与算法3.自然语言
- 大模型微调到底有没有技术含量?
DeepSeek-大模型系统教程
人工智能语言模型ai大模型大模型微调程序员AI大模型
今天给大家带来知乎好友@ybq的一篇回答-大模型微调到底有没有技术含量,或者说技术含量到底有多大?老生常谈的一句话吧:有没有技术含量取决于这个工作你怎么做,尤其是llm方向,上手门槛相比传统NLP变得更低了。我举一些例子吧,针对大模型微调的几个重要环节,我列举的每一种做法大概率都能完成最终目标,甚至说训出来的模型效果都没什么差别。但对个人能力成长的帮助就大不相同了。数据工作做法1:继承实验室或者同
- AI大模型的2种模型能力Function call 和ReAct
DeepSeek-大模型系统教程
人工智能react.js前端gitai语言模型
近年来,随着AI大模型的快速发展,如何让这些模型更好地与现实世界交互成为了一个重要课题。FunctionCall和ReAct作为两种重要的模型能力,为大模型提供了更强大的工具调用和任务执行能力。我们将深入探讨这两种能力的背景、原理、应用场景以及它们之间的对比。帮助你深入了解他们的价值。01背景介绍AI大模型(如GPT-4、PaLM等)在自然语言处理、文本生成等任务中表现出色,但它们的能力往往局限于
- 大模型算法工程师面试宝典:精选面试题及参考答案全解析,助你备战AI算法工程师岗位!
大模型入门学习
人工智能产品经理大数据机器学习程序员大模型大模型学习
大模型应该算是目前当之无愧的最有影响力的AI技术。它正在革新各个行业,包括自然语言处理、机器翻译、内容创作和客户服务等,正成为未来商业环境的重要组成部分。截至目前大模型已超过200个,在大模型纵横的时代,不仅大模型技术越来越卷,就连大模型相关面试也是越来越卷。我今天给大家分享一篇大模型的面试题总结,内容较长,喜欢记得收藏、关注、点赞。ii.为什么会出现LLMs复读机问题?出现LLMs复读机问题可能
- 百度颠覆了自己,飞算JavaAI造福了中国程序员!
飞算JavaAI开发助手
百度
在当今这个科技日新月异的时代,企业纷纷寻求技术突破,以期在激烈的市场竞争中脱颖而出。百度,作为中国互联网行业的领军企业之一,凭借其强大的科技实力和创新能力,在人工智能等多个领域取得了显著成就,并正在逐步颠覆自身的传统形象。百度自成立之初,就将技术创新视为企业的生命线。从最初的搜索引擎技术,到如今的深度学习、自然语言处理、计算机视觉等前沿领域,百度始终走在技术革新的前沿。其自主研发的飞桨深度学习平台
- Algorithm
香水浓
javaAlgorithm
冒泡排序
public static void sort(Integer[] param) {
for (int i = param.length - 1; i > 0; i--) {
for (int j = 0; j < i; j++) {
int current = param[j];
int next = param[j + 1];
- mongoDB 复杂查询表达式
开窍的石头
mongodb
1:count
Pg: db.user.find().count();
统计多少条数据
2:不等于$ne
Pg: db.user.find({_id:{$ne:3}},{name:1,sex:1,_id:0});
查询id不等于3的数据。
3:大于$gt $gte(大于等于)
&n
- Jboss Java heap space异常解决方法, jboss OutOfMemoryError : PermGen space
0624chenhong
jvmjboss
转自
http://blog.csdn.net/zou274/article/details/5552630
解决办法:
window->preferences->java->installed jres->edit jre
把default vm arguments 的参数设为-Xms64m -Xmx512m
----------------
- 文件上传 下载 解析 相对路径
不懂事的小屁孩
文件上传
有点坑吧,弄这么一个简单的东西弄了一天多,身边还有大神指导着,网上各种百度着。
下面总结一下遇到的问题:
文件上传,在页面上传的时候,不要想着去操作绝对路径,浏览器会对客户端的信息进行保护,避免用户信息收到攻击。
在上传图片,或者文件时,使用form表单来操作。
前台通过form表单传输一个流到后台,而不是ajax传递参数到后台,代码如下:
<form action=&
- 怎么实现qq空间批量点赞
换个号韩国红果果
qq
纯粹为了好玩!!
逻辑很简单
1 打开浏览器console;输入以下代码。
先上添加赞的代码
var tools={};
//添加所有赞
function init(){
document.body.scrollTop=10000;
setTimeout(function(){document.body.scrollTop=0;},2000);//加
- 判断是否为中文
灵静志远
中文
方法一:
public class Zhidao {
public static void main(String args[]) {
String s = "sdf灭礌 kjl d{';\fdsjlk是";
int n=0;
for(int i=0; i<s.length(); i++) {
n = (int)s.charAt(i);
if((
- 一个电话面试后总结
a-john
面试
今天,接了一个电话面试,对于还是初学者的我来说,紧张了半天。
面试的问题分了层次,对于一类问题,由简到难。自己觉得回答不好的地方作了一下总结:
在谈到集合类的时候,举几个常用的集合类,想都没想,直接说了list,map。
然后对list和map分别举几个类型:
list方面:ArrayList,LinkedList。在谈到他们的区别时,愣住了
- MSSQL中Escape转义的使用
aijuans
MSSQL
IF OBJECT_ID('tempdb..#ABC') is not null
drop table tempdb..#ABC
create table #ABC
(
PATHNAME NVARCHAR(50)
)
insert into #ABC
SELECT N'/ABCDEFGHI'
UNION ALL SELECT N'/ABCDGAFGASASSDFA'
UNION ALL
- 一个简单的存储过程
asialee
mysql存储过程构造数据批量插入
今天要批量的生成一批测试数据,其中中间有部分数据是变化的,本来想写个程序来生成的,后来想到存储过程就可以搞定,所以随手写了一个,记录在此:
DELIMITER $$
DROP PROCEDURE IF EXISTS inse
- annot convert from HomeFragment_1 to Fragment
百合不是茶
android导包错误
创建了几个类继承Fragment, 需要将创建的类存储在ArrayList<Fragment>中; 出现不能将new 出来的对象放到队列中,原因很简单;
创建类时引入包是:import android.app.Fragment;
创建队列和对象时使用的包是:import android.support.v4.ap
- Weblogic10两种修改端口的方法
bijian1013
weblogic端口号配置管理config.xml
一.进入控制台进行修改 1.进入控制台: http://127.0.0.1:7001/console 2.展开左边树菜单 域结构->环境->服务器-->点击AdminServer(管理) &
- mysql 操作指令
征客丶
mysql
一、连接mysql
进入 mysql 的安装目录;
$ bin/mysql -p [host IP 如果是登录本地的mysql 可以不写 -p 直接 -u] -u [userName] -p
输入密码,回车,接连;
二、权限操作[如果你很了解mysql数据库后,你可以直接去修改系统表,然后用 mysql> flush privileges; 指令让权限生效]
1、赋权
mys
- 【Hive一】Hive入门
bit1129
hive
Hive安装与配置
Hive的运行需要依赖于Hadoop,因此需要首先安装Hadoop2.5.2,并且Hive的启动前需要首先启动Hadoop。
Hive安装和配置的步骤
1. 从如下地址下载Hive0.14.0
http://mirror.bit.edu.cn/apache/hive/
2.解压hive,在系统变
- ajax 三种提交请求的方法
BlueSkator
Ajaxjqery
1、ajax 提交请求
$.ajax({
type:"post",
url : "${ctx}/front/Hotel/getAllHotelByAjax.do",
dataType : "json",
success : function(result) {
try {
for(v
- mongodb开发环境下的搭建入门
braveCS
运维
linux下安装mongodb
1)官网下载mongodb-linux-x86_64-rhel62-3.0.4.gz
2)linux 解压
gzip -d mongodb-linux-x86_64-rhel62-3.0.4.gz;
mv mongodb-linux-x86_64-rhel62-3.0.4 mongodb-linux-x86_64-rhel62-
- 编程之美-最短摘要的生成
bylijinnan
java数据结构算法编程之美
import java.util.HashMap;
import java.util.Map;
import java.util.Map.Entry;
public class ShortestAbstract {
/**
* 编程之美 最短摘要的生成
* 扫描过程始终保持一个[pBegin,pEnd]的range,初始化确保[pBegin,pEnd]的ran
- json数据解析及typeof
chengxuyuancsdn
jstypeofjson解析
// json格式
var people='{"authors": [{"firstName": "AAA","lastName": "BBB"},'
+' {"firstName": "CCC&
- 流程系统设计的层次和目标
comsci
设计模式数据结构sql框架脚本
流程系统设计的层次和目标
- RMAN List和report 命令
daizj
oraclelistreportrman
LIST 命令
使用RMAN LIST 命令显示有关资料档案库中记录的备份集、代理副本和映像副本的
信息。使用此命令可列出:
• RMAN 资料档案库中状态不是AVAILABLE 的备份和副本
• 可用的且可以用于还原操作的数据文件备份和副本
• 备份集和副本,其中包含指定数据文件列表或指定表空间的备份
• 包含指定名称或范围的所有归档日志备份的备份集和副本
• 由标记、完成时间、可
- 二叉树:红黑树
dieslrae
二叉树
红黑树是一种自平衡的二叉树,它的查找,插入,删除操作时间复杂度皆为O(logN),不会出现普通二叉搜索树在最差情况时时间复杂度会变为O(N)的问题.
红黑树必须遵循红黑规则,规则如下
1、每个节点不是红就是黑。 2、根总是黑的 &
- C语言homework3,7个小题目的代码
dcj3sjt126com
c
1、打印100以内的所有奇数。
# include <stdio.h>
int main(void)
{
int i;
for (i=1; i<=100; i++)
{
if (i%2 != 0)
printf("%d ", i);
}
return 0;
}
2、从键盘上输入10个整数,
- 自定义按钮, 图片在上, 文字在下, 居中显示
dcj3sjt126com
自定义
#import <UIKit/UIKit.h>
@interface MyButton : UIButton
-(void)setFrame:(CGRect)frame ImageName:(NSString*)imageName Target:(id)target Action:(SEL)action Title:(NSString*)title Font:(CGFloa
- MySQL查询语句练习题,测试足够用了
flyvszhb
sqlmysql
http://blog.sina.com.cn/s/blog_767d65530101861c.html
1.创建student和score表
CREATE TABLE student (
id INT(10) NOT NULL UNIQUE PRIMARY KEY ,
name VARCHAR
- 转:MyBatis Generator 详解
happyqing
mybatis
MyBatis Generator 详解
http://blog.csdn.net/isea533/article/details/42102297
MyBatis Generator详解
http://git.oschina.net/free/Mybatis_Utils/blob/master/MybatisGeneator/MybatisGeneator.
- 让程序员少走弯路的14个忠告
jingjing0907
工作计划学习
无论是谁,在刚进入某个领域之时,有再大的雄心壮志也敌不过眼前的迷茫:不知道应该怎么做,不知道应该做什么。下面是一名软件开发人员所学到的经验,希望能对大家有所帮助
1.不要害怕在工作中学习。
只要有电脑,就可以通过电子阅读器阅读报纸和大多数书籍。如果你只是做好自己的本职工作以及分配的任务,那是学不到很多东西的。如果你盲目地要求更多的工作,也是不可能提升自己的。放
- nginx和NetScaler区别
流浪鱼
nginx
NetScaler是一个完整的包含操作系统和应用交付功能的产品,Nginx并不包含操作系统,在处理连接方面,需要依赖于操作系统,所以在并发连接数方面和防DoS攻击方面,Nginx不具备优势。
2.易用性方面差别也比较大。Nginx对管理员的水平要求比较高,参数比较多,不确定性给运营带来隐患。在NetScaler常见的配置如健康检查,HA等,在Nginx上的配置的实现相对复杂。
3.策略灵活度方
- 第11章 动画效果(下)
onestopweb
动画
index.html
<!DOCTYPE html PUBLIC "-//W3C//DTD XHTML 1.0 Transitional//EN" "http://www.w3.org/TR/xhtml1/DTD/xhtml1-transitional.dtd">
<html xmlns="http://www.w3.org/
- FAQ - SAP BW BO roadmap
blueoxygen
BOBW
http://www.sdn.sap.com/irj/boc/business-objects-for-sap-faq
Besides, I care that how to integrate tightly.
By the way, for BW consultants, please just focus on Query Designer which i
- 关于java堆内存溢出的几种情况
tomcat_oracle
javajvmjdkthread
【情况一】:
java.lang.OutOfMemoryError: Java heap space:这种是java堆内存不够,一个原因是真不够,另一个原因是程序中有死循环; 如果是java堆内存不够的话,可以通过调整JVM下面的配置来解决: <jvm-arg>-Xms3062m</jvm-arg> <jvm-arg>-Xmx
- Manifest.permission_group权限组
阿尔萨斯
Permission
结构
继承关系
public static final class Manifest.permission_group extends Object
java.lang.Object
android. Manifest.permission_group 常量
ACCOUNTS 直接通过统计管理器访问管理的统计
COST_MONEY可以用来让用户花钱但不需要通过与他们直接牵涉的权限
D
![基于实体BIO标签嵌入以及多任务学习的不均衡数据关系提取[ACL 2019]阅读笔记_第2张图片](http://img.e-com-net.com/image/info8/cf94c9afaef446f8a3138519ec88a680.jpg)
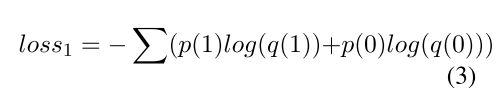
![基于实体BIO标签嵌入以及多任务学习的不均衡数据关系提取[ACL 2019]阅读笔记_第3张图片](http://img.e-com-net.com/image/info8/05917858f3b64d80b55703385f584cfc.jpg)
![基于实体BIO标签嵌入以及多任务学习的不均衡数据关系提取[ACL 2019]阅读笔记_第4张图片](http://img.e-com-net.com/image/info8/f5c7a893ad144130b8331c2e0518fdfe.jpg)

![基于实体BIO标签嵌入以及多任务学习的不均衡数据关系提取[ACL 2019]阅读笔记_第6张图片](http://img.e-com-net.com/image/info8/8998a2ec83bf43caa1ec3b41da5ce789.jpg)
![基于实体BIO标签嵌入以及多任务学习的不均衡数据关系提取[ACL 2019]阅读笔记_第7张图片](http://img.e-com-net.com/image/info8/edc64385e1564a4db41a0c2b1a4a6da4.jpg)
![基于实体BIO标签嵌入以及多任务学习的不均衡数据关系提取[ACL 2019]阅读笔记_第1张图片](http://img.e-com-net.com/image/info8/79d296bdd8cb4a70aa96b96f71198840.jpg)
![基于实体BIO标签嵌入以及多任务学习的不均衡数据关系提取[ACL 2019]阅读笔记_第5张图片](http://img.e-com-net.com/image/info8/1ff9ae80228747329318679f4e216052.jpg)