Lasso Regression
Lasso Regression
标签(空格分隔): 监督学习
在数据挖掘和机器学习算法的模型建立之初,为了尽量的减少因缺少重要变量而出现的模型偏差问题,我们通常会尽可能的多的选择自变量。但是在实际建模的过程中,通常又需要寻找 对响应变量具有解释能力的自变量子集,以提高模型的解释能力与预测精度,这个过程称为特征选择。
还是考虑《线性回归》中的一般线性回归模型 y=wTx ,使用最小二乘估计(OLS)可以得到,模型的参数为:
最小二乘估计虽然有不错的解析性,但是其在大多数情况下的数据分析能力是不够的,主要有两个原因:
预测精度问题:最小二乘法虽然是无偏估计,但是他的方差在自变量存在多重共线性(变量间线性相关)时会非常大,这个可以通过将某些系数压缩到0来改进预测精度,但这个是以一定的有偏为代价来降低预测值的方差。
模型的可解释性:自变量个数很多的时候,我们总是希望能够确定一个较小的变量模型来表现较好的结果
对于以上的问题,就有两种方法可以对最小二乘估计进行改进:子集选择和脊归回。子集选择过程中,对变量要么保留,要么剔除,这很可能使得观测数据的一个微小变动就导致要选择一个新的模型,使得模型变得不稳定,但由于模型的变量少了,使得模型的解释性得到了提高;脊回归是一个连续的方法,它在不抛弃任何一个变量的情况下,缩小了回归系数,使得模型相对而言比较的稳定,但这会使得模型的变量特别多,模型解释性差。
基于以上的问题,才有了现在要说的一种新的变量选择技术:Lasso(Least Absolute Shrinkage and Selection Operator)。这种方法使用模型系数的 l1 范数来压缩模型的系数,使得一些系数变小,甚至还是一些绝对值较小的系数直接变为0,这就使得这种方法同时具有了自己选择和脊回归的优点。
Lasso回归模型,是一个用于估计稀疏参数的线性模型,特别适用于参数数目缩减。基于这个原因,Lasso回归模型在压缩感知(compressed sensing)中应用的十分广泛。从数学上来说,Lasso是在线性模型上加上了一个 l1 正则项,其目标函数为:
也可以表示为:
Lasso回归解法
Lasso 回归主要的解法有两种:坐标轴下降法(coordinate descent)和最小角回归法( Least Angle Regression)。
坐标轴下降法
- 坐标下降优化方法是一种非梯度优化算法。为了找到一个函数的局部极小值,在每次迭代中可以在当前点处沿一个坐标方向进行一维搜索。在整个过程中循环使用不同的坐标方向。一个周期的一维搜索迭代过程相当于一个梯度迭代。 其实,gradient descent 方法是利用目标函数的导数(梯度)来确定搜索方向的,而该梯度方向可能不与任何坐标轴平行。而coordinate descent方法是利用当前坐标系统进行搜索,不需要求目标函数的导数,只按照某一坐标方向进行搜索最小值。坐标下降法在稀疏矩阵上的计算速度非常快,同时也是Lasso回归最快的解法。
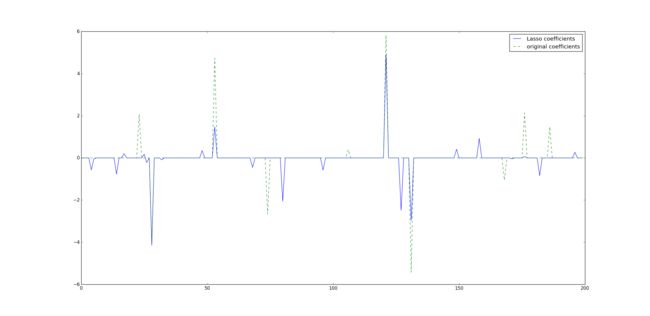
下面这份代码是在稀疏系数上使用Lasso回归,这里Lasso内置的是坐标下降法:
#!/usr/bin/python
# -*- coding: utf-8 -*-
"""
author : [email protected]
time : 2016-06-06_15-41
Lasso 回归应用于稀疏信号
"""
print(__doc__)
import numpy as np
import matplotlib.pyplot as plt
import time
from sklearn.linear_model import Lasso
from sklearn.metrics import r2_score
# 用于产生稀疏数据
np.random.seed(int(time.time()))
# 生成系数数据,样本为50个,参数为200维
n_samples, n_features = 50, 200
# 基于高斯函数生成数据
X = np.random.randn(n_samples, n_features)
# 每个变量对应的系数
coef = 3 * np.random.randn(n_features)
# 变量的下标
inds = np.arange(n_features)
# 变量下标随机排列
np.random.shuffle(inds)
# 仅仅保留10个变量的系数,其他系数全部设置为0
# 生成稀疏参数
coef[inds[10:]] = 0
# 得到目标值,y
y = np.dot(X, coef)
# 为y添加噪声
y += 0.01 * np.random.normal((n_samples,))
# 将数据分为训练集和测试集
n_samples = X.shape[0]
X_train, y_train = X[:n_samples / 2], y[:n_samples / 2]
X_test, y_test = X[n_samples / 2:], y[n_samples / 2:]
# Lasso 回归的参数
alpha = 0.1
lasso = Lasso(max_iter=10000, alpha=alpha)
# 基于训练数据,得到的模型的测试结果
# 这里使用的是坐标轴下降算法(coordinate descent)
y_pred_lasso = lasso.fit(X_train, y_train).predict(X_test)
# 这里是R2可决系数(coefficient of determination)
# 回归平方和(RSS)在总变差(TSS)中所占的比重称为可决系数
# 可决系数可以作为综合度量回归模型对样本观测值拟合优度的度量指标。
# 可决系数越大,说明在总变差中由模型作出了解释的部分占的比重越大,模型拟合优度越好。
# 反之可决系数小,说明模型对样本观测值的拟合程度越差。
# R2可决系数最好的效果是1。
r2_score_lasso = r2_score(y_test, y_pred_lasso)
print("测试集上的R2可决系数 : %f" % r2_score_lasso)
plt.plot(lasso.coef_, label='Lasso coefficients')
plt.plot(coef, '--', label='original coefficients')
plt.legend(loc='best')
plt.show()
最小角回归法
在阐述最小角回归( Least Angle Regression)算法之前,这里需要对两种更为简单直观的前向(Forward)算法做一些说明,最小回归算法是以这两种前向算法为基础的:
前向选择算法
前向选择(Forward Selection)算法,是一种典型的贪心算法。它在自变量 xi,i∈[1,N] 中,选择和目标 y 最为接近的一个自变量 xk ,用 xk 来逼近 y ,得到 y~=βkxk ,其中:
即: y~ 是 y 在 xk 上的投影。那么,可以定义残差(residual): yres=y−y~ 。很容易知道 yres 和 xk 是正交的。再以 yres 为新的因变量,除去 xk 后,剩下的自变量的集合 {xi,i=1,2,3...k−1,k+1,...N} 为新的自变量集合,重复刚才投影和残差的操作,直到残差为0,或者所有的自变量都用完了,才停止算法。
此算法对每个变量只需要执行一次操作,效率高,速度快。但也容易看出,当自变量不是正交的时候,由于每次都是在做投影,所有算法只能给出最优解的一个近似解。
前向梯度算法
前向梯度(Forward Stagewise)和前向选择算法类似,也是每次取相关性最大的一个特征 xk ,然后用 xk 逼近目标 y 。但与之不同的是,前向梯度算法的残差并不是直接使用投影得到的,其计算方式为:
然后和前向选择算法一样,使用 yres 为新的目标,但是不将 xk 从自变量的集合中剔除出去,因为&x_{k}&的相关度可能仍然是最高的。如此进行下去,直到 yres 减小到一定的范围,算法停止。
这个算法在 ϵ 很小的时候,可以很精确的给出最优解,当然,其计算复杂度也随之增加。
最小角回归
计算机领域的很多算法的提出都是这样:先给出两种算法,一种是速度快的,精度低;另一种是精度高的,太复杂。于是(这就像动漫里面一定有两个男主和一个女主,一个能力特别强,但是特别高冷;一个稍弱能力弱,但是2B搞笑,最后两人同时追女主一样。说的大一点,这也算是一个种人生的哲学)计算机领域中,就会出现一个结合前两个算法的第三个算法,是前两种算法的折中,其速度不算特别慢,精度也还不错。在本文,下面就要提出最小角回归(Least Angle Regression)。
LARS算法是结合前两种前向算法的所得到的。
首先,还是找到与因变量 y 相关度最高的自变量 xk ,使用类似于前向梯度算法中的残差计算方法,得到新的目标 yres ,重复这个过程。
直到出现一个 xl ,使得 xl 和 yres 的相关度和 xk 与 yres 的相关度是一样的,此时就在 xl 和 xk 的角分线方向上,继续使用类似于前向梯度算法中的残差计算方法,逼近 y 。
当出现第三个特征 xp 和 yres 的相关度足够大的时候,将其也叫入到 y 的逼近特征集合中,并用 y 的逼近特征集合的共同角分线,作为新的逼近方向。以此循环,直到 yres 足够的小,或者说所有的变量都已经取完了,算法停止。
LARS算法是一个适用于高维数据的回归算法,其主要的优点如下:
对于特征维度 n 远高于样本点数 m 的情况( n≥m ),该算法有极高的数值计算效率
该算法的最坏计算复杂度和最小二乘法(OLS)类似,但是其计算速度几乎和前向选择算法一样
它可以产生分段线性结果的完整路径,这在模型的交叉验证中极为有用
其主要的确点为:
- 由于LARS的迭代方向是根据目标的残差 yres 定的,所以该算法对样本的噪声是极为敏感的
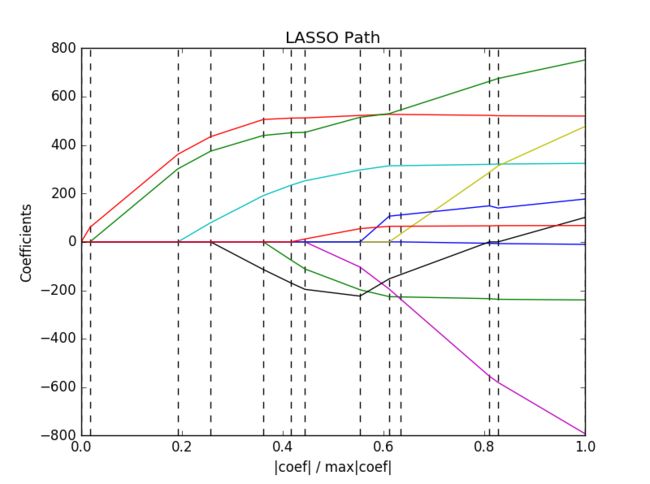
下面这份代码是LARS算法的展示:
#!/usr/bin/python
# -*- coding: utf-8 -*-
"""
author : [email protected]
time : 2016-06-07-10-24
LARS测试代码
这里计算了LARS算法在diabetes数据集上,其正则化参数的路径
最终结果图中的每一个颜色代表参数向量中不同的特征
"""
print(__doc__)
import numpy as np
import matplotlib.pyplot as plt
from sklearn import linear_model
from sklearn import datasets
# 导入数据集
# 这个数据集,总的样本个数为442个,特征维度为10
diabetes = datasets.load_diabetes()
X = diabetes.data
y = diabetes.target
print X.shape
# 所谓参数正则化路径
# 其实就是LARS算法每次迭代的时候,每个参数的数值所组成的曲线
# 其横轴对应着迭代的程度,纵轴是每个特征参数对应的数值
# 这里一共有10个特征,所以有10条特征正则化曲线
print("基于LARS算法计算正则化路径:")
alphas, _, coefs = linear_model.lars_path(X, y, method='lasso', verbose=True)
# 这里讲迭代程度归一化到[0,1]直间
xx = np.sum(np.abs(coefs.T), axis=1)
xx /= xx[-1]
plt.plot(xx, coefs.T)
ymin, ymax = plt.ylim()
plt.vlines(xx, ymin, ymax, linestyle='dashed')
plt.xlabel('|coef| / max|coef|')
plt.ylabel('Coefficients')
plt.title('LASSO Path')
plt.axis('tight')
plt.show()LARS会计算参数正则化路径上的每一个拐点,所以,如果设计矩阵(数据矩阵)的尺寸比较的小,即样本和特征的数目比较的少,那么LARS将会有很高的计算效
当然,即便在没有元参数的情况下,LARS也可以计算出完整的参数路径
坐标轴下降法是在一个事先确定的坐标轴顺序上计算参数路径的
所以,如果坐标轴数目比路径上拐点的数目要少的话,那么坐标轴下降法效率会更高
基于Lasso的特征选择
使用 l1 正则项的线性回归模型,Lasso,其拥有稀疏的回归结果,即:回归后的参数大部分的为0,而那些不为0的参数所对应的变量,可以作为其他的分类模型的特征,这就是基于Lasso的特征选择。
下面这个代码对基于Lasso的特征选择做了说明
#!/usr/bin/python
# -*- coding: utf-8 -*-
"""
author : [email protected]
time : 2016-06-06_16-39
基于lasso的特征选择
这个功能一般和其他的分类器一起使用
或直接内置于其他分类器算中
"""
import numpy as np
import time
from sklearn.svm import LinearSVC
from sklearn.datasets import load_iris
from sklearn.feature_selection import SelectFromModel
from sklearn.metrics import r2_score
np.random.seed(int(time.time()))
# 导入iris数据集
# 这个数据集一共有150个样本,特征维数为4维
iris = load_iris()
X, y = iris.data, iris.target
print '生成矩阵的尺寸:150, 4'
print X.shape
# 对原始样本重排列
inds = np.arange(X.shape[0])
np.random.shuffle(inds)
# 提取训练数据集和测试数据集
X_train = X[inds[:100]]
y_train = y[inds[:100]]
X_test = X[inds[100:]]
y_test = y[inds[100:]]
print '原始特征的维度:', X_train.shape[1]
# 线性核的支持向量机分类器(Linear kernel Support Vector Machine classifier)
# 支持向量机的参数C为0.01,使用l1正则化项
lsvc = LinearSVC(C=0.01, penalty="l1", dual=False).fit(X_train, y_train)
print '原始特征,在测试集上的准确率:', lsvc.score(X_test, y_test)
print '原始特征,在测试集上的R2可决系数:', r2_score(lsvc.predict(X_test), y_test)
# 基于l1正则化的特征选择
model = SelectFromModel(lsvc, prefit=True)
# 将原始特征,转换为新的特征
X_train_new = model.transform(X_train)
X_test_new = model.transform(X_test)
print
print '新特征的维度:', X_train_new.shape[1]
# 用新的特征重新训练模型
lsvc = LinearSVC(C=0.01, penalty="l1", dual=False).fit(X_train_new, y_train)
print '新特征,在测试集上的准确率:', lsvc.score(X_test_new, y_test)
print '新始特征,在测试集上的R2可决系数:', r2_score(lsvc.predict(X_test_new), y_test)
其运行结果为:
生成矩阵的尺寸:150, 4
(150L, 4L)
原始特征的维度: 4
原始特征,在测试集上的准确率: 0.62
原始特征,在测试集上的R2可决系数: 0.506237006237
新特征的维度: 3
新特征,在测试集上的准确率: 0.62
新始特征,在测试集上的R2可决系数: 0.506237006237由运行结果可以看出,基于Lasso的特征选择,在不改变模型测试集准去率的情况下,减小了特征的维度。
在实际测试中,还存在特征维度被较小到了2的情况,但是出现的次数并不多。
- 关于 l1 特征选择的应用中,最具有现实意义的,莫过于文本分类,文本的原始特征维度几乎是整个字典,而一个文本的单词量基本上在100~1000之间,所以文本分类中的设计矩阵是一个极其稀疏的举证,在我实际的测试中,其稀疏度可以达到1%以下。对于这种问题, l1 惩罚会非常的有用,相关的原理和代码示例,会在文本分类的章节中说明。
参数 α 的选择
对于Lasso回归的损失函数而言:
其中 α 控制了稀疏参数估计的惩罚程度。但如何确定其具体应该取值为多少,并没有一个绝对好的算法或者评判标准,这里提供几种常用的模型选择工具来实现 α 的选择:
交叉验证(Cross-validation)
将交叉验证和坐标轴下降法结合,可以有LassoCV算法,和LARS算法结合,可以有LassolarsCV算法。
- 对于变量间存在相关性的高维数据而言,LassoCV往往有更好的效果。但是相对于LassoCV而言,LassolarsCV可以得到更多的 α 相关的参数数值,所以在设计矩阵的维度远大于样本个数的情况下,LassolarsCV比LassoCV有更高的计算效率。
基于信息准则的模型选择
再一个常用的模型选择的方式,就是信息准则,一般有两个信息准则可以使用:
Akaike information criterion (AIC),是衡量统计模型拟合优良性的一种标准,是由日本统计学家赤池弘次创立和发展的。赤池信息量准则建立在熵的概念基础上,可以权衡所估计模型的复杂度和此模型拟合数据的优良性,优先考虑的模型应是AIC值最小的那一个。
AIC=2k−2ln(L)这里, L 是模型极大似然函数的最大值,而 k 是模型参数的个数。
Bayesian Information Criterions(BIC),贝叶斯信息准则(BIC)是在赤池信息量准则(AIC)的基础上建立起来的,它们两非常的相似。相对于AIC而言,BIC对模型的复杂度的惩罚更为重一些。和AIC一样,优先考虑的模型应是BIC值最小的那一个。
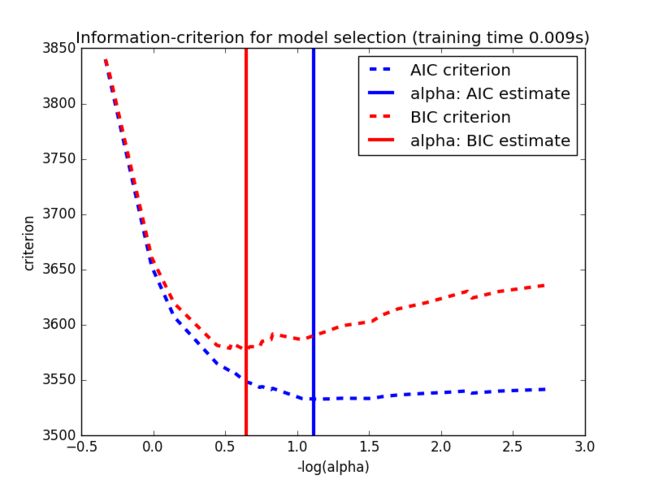
下面这份代码展示了Lasso中参数 α 的模型选择:
#!/usr/bin/python
# -*- coding: utf-8 -*-
"""
author : [email protected]
time : 2016-06-07_13-47
基于CV/AIC/BIC的 Lasso模型选择
CV(cross-validation)交叉验证
AIC(Akaike information criterion)赤池信息准则
BIC(Bayes Information criterion)贝叶斯信息准则
这里AIC和BIC信息准则使用的是LassoLarsIC实现的,使用的是LARS算法
基于信息准则的模型选择的速度是非常的快的
但是,其依赖于模型是正确的基本假设,且对模型自由度需要有恰当的估计,
这样才能在大量的样本上得到一个渐进的结果。
当特征的数量远大于样本的数量的时候,信息准则的模型选择效果并不理想
对于交叉验证而言,基于坐标轴下降算法的交叉验证可以使用LassoCV
基于LARS算法的交叉验证可以使用LassoLarsCV
在实际使用中,这两种算法仅仅是在速度上存在一定的差异,其结果几乎差不多
由于参数的选择对未知的数据可能不是最优的
所以在评价一个使用交叉验证得到的参数的方法的时候
嵌套交叉验证是有必要的
"""
print(__doc__)
import time
import numpy as np
import matplotlib.pyplot as plt
from sklearn.linear_model import LassoCV, LassoLarsCV, LassoLarsIC
from sklearn import datasets
# 加载数据集
# 该数据集有442个样本,特征的维度为10
diabetes = datasets.load_diabetes()
X = diabetes.data
y = diabetes.target
print X.shape
# 选择随机种子
rng = np.random.RandomState(int(time.time())%100)
# 添加14个噪声的特征
X = np.c_[X, rng.randn(X.shape[0], 14)]
# 对每个特征做数据归一化,这个过程是LARS算法需要的
X /= np.sqrt(np.sum(X ** 2, axis=0))
# LassoLarsIC: 使用LARS算法做BIC/AIC信息准则
model_bic = LassoLarsIC(criterion='bic')
t1 = time.time()
model_bic.fit(X, y)
t_bic = time.time() - t1
alpha_bic_ = model_bic.alpha_
model_aic = LassoLarsIC(criterion='aic')
model_aic.fit(X, y)
alpha_aic_ = model_aic.alpha_
# 这里alpha_是最终选择的参数
# alphas_是所有的alpha选择
# alpha_就是alphas_中,对于的信息准则最小的那个值
# criterion_是和alphas_对应的信息准则的结果
def plot_ic_criterion(model, name, color):
alpha_ = model.alpha_
alphas_ = model.alphas_
criterion_ = model.criterion_
plt.plot(-np.log10(alphas_), criterion_, '--', color=color,
linewidth=3, label='%s criterion' % name)
plt.axvline(-np.log10(alpha_), color=color, linewidth=3,
label='alpha: %s estimate' % name)
plt.xlabel('-log(alpha)')
plt.ylabel('criterion')
plt.figure()
plot_ic_criterion(model_aic, 'AIC', 'b')
plot_ic_criterion(model_bic, 'BIC', 'r')
plt.legend()
plt.title('Information-criterion for model selection (training time %.3fs)'
% t_bic)
# LassoCV: 基于坐标轴下降法的Lasso交叉验证
print("使用坐标轴下降法计算参数正则化路径:")
t1 = time.time()
# 这里是用20折的交叉验证
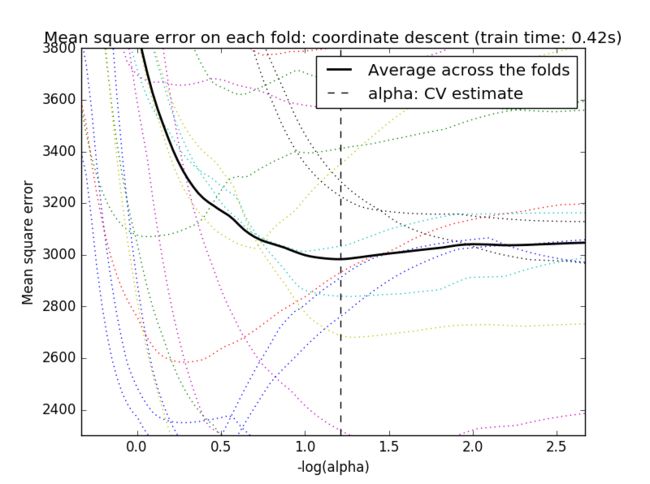
model = LassoCV(cv=20).fit(X, y)
t_lasso_cv = time.time() - t1
# 最终alpha的结果,因为有的alpha实在是太小了
# 所以使用负对数形式表示
m_log_alphas = -np.log10(model.alphas_)
# 由于这里使用的是20折交叉验证
# 所以model.mse_path_有20列
# model.mse_path_中每一列,是对应交叉验证,在alpha选择不同值的时候
# 其对应的均方误差(mean square error)
# 模型最终选择的alpha是所有交叉验证结果的平均值中
# 最小的那个平均的均方误差对应的alpha
plt.figure()
ymin, ymax = 2300, 3800
plt.plot(m_log_alphas, model.mse_path_, ':')
plt.plot(m_log_alphas, model.mse_path_.mean(axis=-1), 'k',
label='Average across the folds', linewidth=2)
plt.axvline(-np.log10(model.alpha_), linestyle='--', color='k',
label='alpha: CV estimate')
plt.legend()
plt.xlabel('-log(alpha)')
plt.ylabel('Mean square error')
plt.title('Mean square error on each fold: coordinate descent '
'(train time: %.2fs)' % t_lasso_cv)
plt.axis('tight')
plt.ylim(ymin, ymax)
# LassoLarsCV: 基于LARS算法的交叉验证
print("使用LARS算法计算参数正则化路径:")
t1 = time.time()
model = LassoLarsCV(cv=20).fit(X, y)
t_lasso_lars_cv = time.time() - t1
# 最终alpha的结果,因为有的alpha实在是太小了
# 所以使用负对数形式表示
m_log_alphas = -np.log10(model.cv_alphas_)
# 参数说明和上面是一样的
plt.figure()
plt.plot(m_log_alphas, model.cv_mse_path_, ':')
plt.plot(m_log_alphas, model.cv_mse_path_.mean(axis=-1), 'k',
label='Average across the folds', linewidth=2)
plt.axvline(-np.log10(model.alpha_), linestyle='--', color='k',
label='alpha CV')
plt.legend()
plt.xlabel('-log(alpha)')
plt.ylabel('Mean square error')
plt.title('Mean square error on each fold: Lars (train time: %.2fs)'
% t_lasso_lars_cv)
plt.axis('tight')
plt.ylim(ymin, ymax)
plt.show()