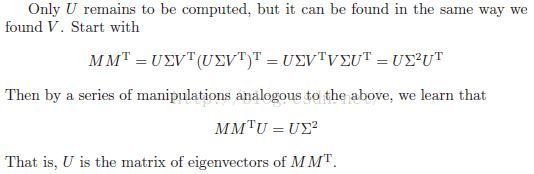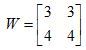海量数据挖掘MMDS week4: 推荐系统之数据降维Dimensionality Reduction
http://blog.csdn.net/pipisorry/article/details/49231919
海量数据挖掘Mining Massive Datasets(MMDs) -Jure Leskovec courses学习笔记 推荐系统Recommendation System之降维Dimensionality Reduction
{博客内容:推荐系统有一种推荐称作隐语义模型(LFM, latent factor model)推荐,这种推荐将在下一篇博客中讲到。这篇博客主要讲隐语义模型的基础:降维技术,包括SVD分解等等}
降维Dimensionality Reduction
降维介绍
数据的低维表示:空间中的点不是完全随机分布的,而是分布lie in在它的一个子空间中。我们的目标就是找到这个可以有效表示所有数据的子空间。
降维示例
customer-day矩阵中行表示data(也就是点),列代表数据属性(也就是点的坐标)。降维就是要减少属性(列)。
这个矩阵实际只有2维,wc-th-fr和sa-su。
数据集的维度
矩阵的秩
矩阵A的秩就是A中列的无关最大组数目。下面的是坐标重新定义后A矩阵的表示。
秩即维数
通过秩来进行坐标重定义,用新坐标重新表示A矩阵,达到降维目的。
降维的实质
实质是找到一个新的数据轴。
这个例子中,我们只考虑数据在红线上的投影,而忽略与红线的距离,存在一定的error。目标就是找到一个新坐标轴让error尽量小。
降维的目的
皮皮blog
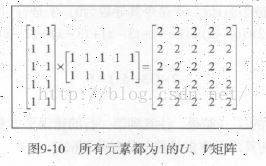
UV分解
UV分解示例
UV分解误差度量RMSE
我们一般通过RMSE(Root-Mean-Square Error, 均方根误差)度量UV和M的相近程度。
UV分解的增量式计算
{寻找具有最小RMSE的UV分解过程:初始任意选择UV,然后反复调整UV使得RMSE越来越小}
初始化
增量计算-对特定元素优化的示例
增量计算-对任意元素的优化。。。
完整UV分解算法的构建
1. 预处理
2.初始化
3. 执行优化
4. 收敛到极小值
5. 避免过拟合
皮皮blog
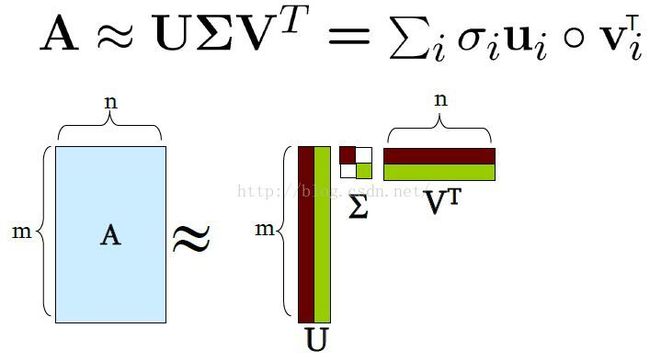
奇异值分解Singular Value Decomposition, SVD
{数据降维技术}
SVD定义
这里假设奇异值对角矩阵中的奇异值是降序排列的。
[矩阵论]
SVD分解的性质
Note: U、V列向量是正交的orthogonal,也就是说向量间内积为0。
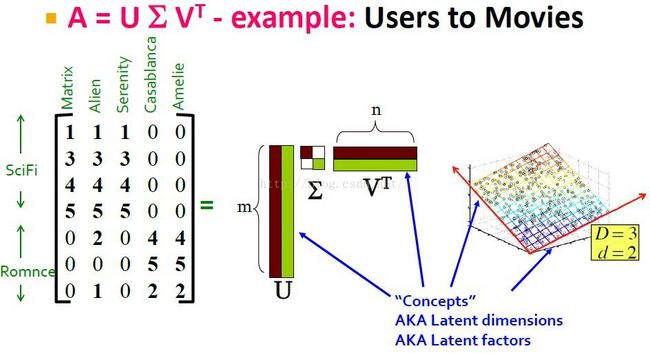
SVD分解实例
users-movies矩阵,其中行代表用户,每列代表一部电影。
concepts就是SVD分解要告诉我们的,用户是sci-fi lover和romance lover类型,电影是sci-fi和romance等类型。也就是不同的genres(流派), or topics。
SVD分解中各分解分量的实际意义解释
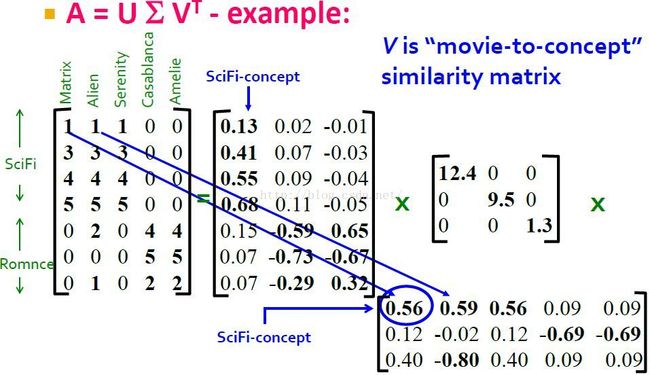
下面是通过matlab或者python对矩阵A进行SVD分解得到的结果。下面分别讲解U V矩阵代表的实际含义(注意这种解释性也是人为解释的,其实SVD分解的解释性并没有那么强)。
我们可以将U的列看成concepts,如U的第一列对应Sci-Fi concept,第二列对应romance concept(第三列可能代表其它的什么,其实不一定能用一个类别来描述和解释,因为SVD其解释性并不是那么强)。我们从这里可以看到,前4个用户衷情于sic-fi,后3个用户衷情于romance。
于是我们可以将U矩阵看成是user to concept matrix(user to concept similarity matrix)。其中元素代表某个用户对某个concepts的感兴趣程度。这里是说第1个用户很喜欢sci-fi concept(0.13),而第5个用户很喜欢romance concept(-0.59)。至于-0.59代表最喜欢的concept,可能是要看它的绝对值?
Sigma矩阵中的值可看做是concepts的强度,如sci-fi concept强度(12.4)就比romance concept的强度(9.5)强。
同样的,我们可以将V矩阵看成是movies to concept matrix(movies to concept similarity matrix)。注意这里还有第三种concepts,但是其强度1.3太小,可以忽略。
从V矩阵第1列我们可以看到,第1部电影与第1个concept和第3个concept相关度高,然而第3个concept的强度过低,它对解释数据并不重要。
[TopicModel主题模型 - LSA(隐性语义分析)模型和其实现的早期方法SVD]
使用concept space进行查询
如果原始矩阵中没有的一个新用户Qurncy看了一部电影The Matrix,评分为4,则Qurncy的向量表示为q=[4,0,0,0,0]。
当然使用协同过滤,不过这里可以通过qV将Qurncy map到concept space中,其中qV = [2.32, 0],说明Qurncy对scifi有很大兴趣,对romance几乎没有兴趣。
map回movie space,qVVT = [1.35, 1.35, 1.35, 0, 0],也就是说Qurncy会喜欢Alien and Star Wars, but not Casablanca or Titanic。
可以将所有用户都map到concept space中,再计算他们的cosin相似度。
SVD分解的计算
Note: m*n矩阵A的奇异值是矩阵乘积AAH的特征值(这些特征值是非负的)的正平方根。
当然可以使用软件来代替手动计算,如scipy中linalg.svd(A)
[SVD Dimension Reduction 奇异值分解 降维]
皮皮blog
CUR分解
SVD分解的缺点在于:
计算比较耗时
存储矩阵比较占空间
CUR分解是另外一个选择,其目标是:找到输入矩阵的一个“尽可能好”的分解为三个矩阵的乘积,SVD分解是完美的分解(通过允许误差来加速计算)。
U矩阵构造
C矩阵构造
计算M所有数据平方和f,取出的行数为r
计算选取列的缩放scale因子
![]()
R矩阵构造
同C,只是概率(缩放因子计算是对行来说的)
CUR分解示例
对下面矩阵进行CUR分解
假设选取列Matrix, Alien,和行Jim, John。
所有元素平方和为243
Matrix, Alien和Star Wars的squared Frobenius norm为1^2 + 3^2 + 4^2 + 5^2 = 51,故缩放因子即概率为51/243 = .210。其它两列的概率为45/243 = .185。
7行的squared Frobenius norms分别为3, 27, 48, 75, 32,50, 8,相应的概率为.012, .111, .198,.309, .132, .206, .033.
C矩阵构造
C矩阵中选择了Alien列和Matrix 列,则
R矩阵的构造
R矩阵选择了行Jim, John。
U矩阵的构造
W矩阵为
W的SVD分解结果为:
u:
[[-0.6-0.8]
[-0.8 0.6]]
e:
[ 7.0711 0. ]
v:
[[-0.7071-0.7071]
[-0.7071 0.7071]]
1/e =
[[0.1414 0. ]
[ 0. 0. ]]
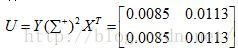
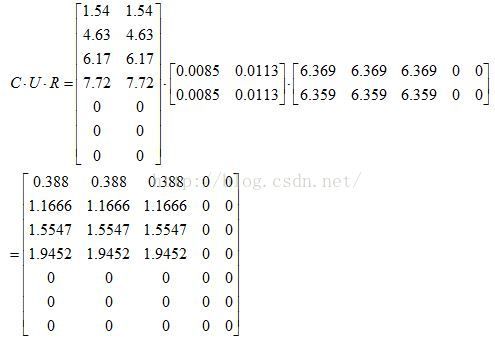
故M矩阵的CUR分解为:
[Anand.Rajaraman-Mining of Massive Datasets-mmds2014:CUR Decomposition]
[CUR Dimension Reduction CUR分解 降维]
皮皮blog
。。。
Review复习
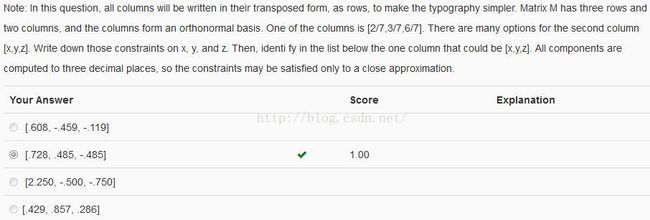
标准正交基
计算出各个选项的长度(2范数)和与[2/7...]的内积为:
0.771042151896
-0.125
1.00021697646
0.000142857142857
2.42383992871
-0.214285714286
1.00014298978
0.735
故选择选项2
Code:
a = np.array([2 / 7, 3 / 7, 6 / 7]) B = [[.608, -.459, -.119], [.728, .485, -.485], [2.250, -.500, -.750], [.429, .857, .286]] for b in B: print(np.linalg.norm(b)) print(a.dot(b)) print()
PCA
皮皮blog
ref: Anand.Rajaraman-大数据:互联网大规模数据挖掘与分布式处理