本文转自生信人https://www.shengxin.ren/article/16
1、简介
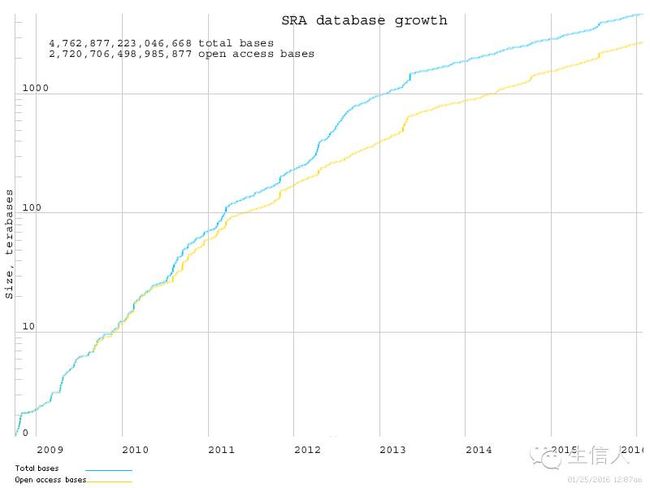
SRA(Sequence ReadArchive)数据库是用于存储二代测序的原始数据,包括 454,Illumina,SOLiD,IonTorrent,Helicos 和 CompleteGenomics。除了原始序列数据外,SRA现在也存在raw reads在参考基因的比对信息。
根据SRA数据产生的特点,将SRA数据分为四类:
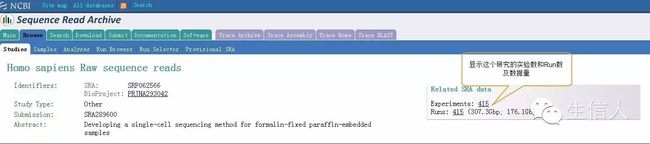
Studies-- 研究课题
Experiments-- 实验设计
Runs-- 测序结果集
Samples-- 样品信息
SRA中数据结构的层次关系为:Studies->Experiments->Samples->Runs.
Studies是就实验目标而言的,一个study 可能包含多个Experiment。
Experiments包含了Sample、DNA source、测序平台、数据处理等信息。
一个Experiment可能包含一个或多个runs。
Runs 表示测序仪运行所产生的reads。
SRA数据库用不同的前缀加以区分:
ERP或SRP表示Studies;
SRS 表示 Samples;
SRX 表示 Experiments;
SRR 表示 Runs;
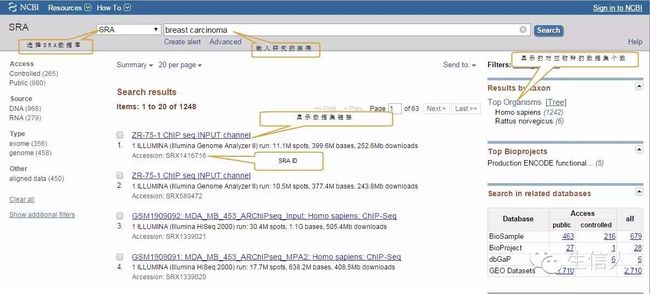
2、使用
3、下载数据
要下载SRA数据,我们需要先安装SRA Toolkit软件包,下载地址:
https://www.ncbi.nlm.nih.gov/Traces/sra/sra.cgi?view=software
根据自己的环境下载相应的软件包。
主要包括:
CentOS 32/64, Ubuntu 32/64, MacOS 32/64, MS Windows 32/64
以CentOS为例:
1、下载安装:
wget "http://ftp-trace.ncbi.nlm.nih.gov/sra/sdk/current/sratoolkit.current-centos_linux64.tar.gz"
tar xzf sratoolkit.current-centos_linux64.tar.gz
2、运行下载
cd sratoolkit.2.5.7-centos_linux64/bin
./prefetch SRR2172038
下载完成后,会在你的工作主目录下生成一个ncbi的文件夹。
cd ncbi/public/sra
查看SRR2172038.sra数据
3、转换fastq
/sratoolkit.2.5.7-centos_linux64/bin/fastq-dump ./SRR2172038.sra
4、转换fasta
/sratoolkit.2.5.7-centos_linux64/bin/fastq-dump --fasta ./SRR2172038.sra
4、数据提交
一个 SRA study 所包含的内容, 应该在一个 LSBI 的项目中提交。 即 SRA study 和 LSBI项目为 1 对 1 关系。 一个 study 的内容可以在一个项目下, 分成几个批次提交, 每次提交不同的内容。
一个批次的 SRA 数据, 包括一个.info 文件和一个名为 DATA, 装有提交原始文件的子文件夹。 子文件夹中内容为描述 metadata 的 xml 文件或者 sff 等格式的数据文件。 一个完整的 study, 包括一个或多个 study.xml, experiment.xml, sample.xml 和 run.xml, 以及一个或多个数据文件。 但是一个批次的提交数据不一定包括所有的文件。
Run.xml 和该 xml 中包括的所有数据文件, 必须要在一个批次中提交。
(1) 请先确认您已是数据中心网站注册的用户, 否则请登陆中心网站,注册。
(2) 登陆中心网站后,点击左侧菜单的 mydata,选择已有项目或创建新项目。
(3) 选择已有批次或创建新批次。 在创建批次时, 选择要提交的数据类型为“SRA”。
(4) 在点击批次下的 submit data 按钮后, 进入提交页面。
(5) 首先下载离线提交附件( subdesc.bch), 作为离线提交的标识文件, 是离线提交必须的附件之一。
(6) 按照 SRA 的数据格式标准, 处理生成数据文件, 连同标识文件一起, 通过提交页面上显示的路径( 为 ftp://shengxin.ren:1221/SRA/****/) 上传至服务器指定目录。