前言
今天(3.12日),想在NCBI上下载lncRNA数据,突然发现软件Aspera的ascp(报错:ascp: Failed to open TCP connection for SSH, exiting)下载不了。
百度一波,这种问题很少。随后问了身边的小伙伴,大家都没及时回复。
然后就问了外校的小伙伴,发现一样的报错,考虑应该是NCBI在维护。
问题来了,除了ascp以外,其他下载方式有哪些呢?
pengzw@super-server:~/data/ncbi$ ascp -T -l 200M -i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh -T --mode recv --host ftp-private.ncbi.nlm.nih.gov --user anonftp --file-list ./download_list.txt ./
ascp: Failed to open TCP connection for SSH, exiting.
Session Stop (Error: Failed to open TCP connection for SSH)
SRA数据库介绍
NCBI目录文件FTP查找网址
ftp://ftp.ncbi.nlm.nih.gov/sra/sra-instant
1.Aspera高速下载
①简介及安装:由于下载时,官方自动检测系统,linux最新版是师弟在ubuntu上下载传给我。
下载地址:https://downloads.asperasoft.com/en/downloads/8?list
wget https://download.asperasoft.com/download/sw/connect/3.8.1/ibm-aspera-connect-3.8.1.161274-linux-g2.12-64.tar.gz
tar zxf ~/downloads/ibm-aspera-connect-3.8.1.161274-linux-g2.12-64.tar.gz -C ~/biosoft/ #指定安装目录
cd ~/biosoft/
bash ibm-aspera-connect-3.8.1.161274-linux-g2.12-64.sh # 安装(我修改为bash解释器)
cd # 去根目录
ls -a # 查看是否有.aspera文件夹,如果看到.aspera文件夹,代表安装成功
echo 'PATH=$PATH:~/.aspera/connect/bin/' >> ~/.bashrc # 永久添加环境变量
source ~/.bashrc #让当前变量生效
ascp --help
②使用
从ncbi上批量下载
ascp -T -l -i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh -T --mode recv --host ftp-private.ncbi.nlm.nih.gov --user anonftp --file-list ./download_list.txt ./
download_list_list.txt内容:
/sra/sra-instant/reads/ByRun/sra/SRR/SRR103/SRR1039508/SRR1039508.sra
/sra/sra-instant/reads/ByRun/sra/SRR/SRR103/SRR1039509/SRR1039509.sra
ascp [options] SRC... DEST
options:
• -T 不进行加密。若不添加此参数,可能会下载不了。
• -i string 输入私钥,安装aspera后在目录~/.aspera/connect/ect/下有私钥,linux服务器一般使用asperaweb_id_dsa.openssh
• --host=string ftp的host名,NCBI为ftp-private.ncbi.nlm.nih.gov;EBI为fasp.sra.ebi.ac.uk
• --user=string用户名,NCBI为anonftp,EBI为era-fasp
• --mode=string 选择模式,上传为send,下载为recv
• -l string 设置最大传输速度,如设置为200M表示最大传输速度为200M/s。不设置则一般可达10M/s
2.prefetch
①prefetch来自于软件NCBI SRA Toolkit(被称为manual最垃圾的软件之一),帮助mannul。
方法一:prefetch SRRXXXXXXX,生成文件直接会指定到~/ncbi/public/sra/,600M不到4min,很快啦!
pengzw@super-server:~$ time prefetch SRR5504363
2019-04-01T07:18:36 prefetch.2.9.2: 1) Downloading 'SRR5504363'...
2019-04-01T07:18:36 prefetch.2.9.2: Downloading via fasp...
SRR5504363
2019-04-01T07:22:04 prefetch.2.9.2: fasp download succeed
2019-04-01T07:22:04 prefetch.2.9.2: 1) 'SRR5504363' was downloaded successfully
2019-04-01T07:22:04 prefetch.2.9.2: 'SRR5504363' has 0 unresolved dependencies
real 3m32.262s
user 0m6.519s
sys 0m13.074s
pengzw@super-server:~/ncbi/public/sra$ ll -h #查看大小,以防下载有错
总用量 646M
drwxrwxr-x 2 pengzw pengzw 4.0K 4月 1 15:22 ./
drwxrwxr-x 3 pengzw pengzw 4.0K 4月 1 15:18 ../
-rw-r--r-- 1 pengzw pengzw 646M 5月 4 2017 SRR5504363.sra
方法二:prefetch --option-file SRR_Acc_List.txt
prefetch --option-file SRR_Acc_List.txt
prefetch -t ascp -a "~/.aspera/connect/bin/ascp|~/.aspera/connect/etc/asperaweb_id_dsa.openssh" --option-file SRR_Acc_List.txt -O ./ncbi
SRR_Acc_List.txt内容为SRRXXXXXXX
注意:
sra数据会下载到家目录下的ncbi/public/sra中,perfetch 默认aspera下载(如果存在于环境变量,否则使用https下载),也可设置aspera,Ex:prefetch -t ascp -a "/opt/aspera/bin/ascp|/opt/aspera/bin/asperaweb_id_dsa.openssh" --option-file file.txt; file.txt 格式为每一行一个SRR#,可以使用下载界面的RunInfo table下载的文件。
3.fasterq-dump and fastq-dump
①fastq-dump也来自于SRA Toolkit,可以转换sra为fq文件,只能单线程(对于我习惯使用ascp,简直无法忍受)。通过整理发现也可以下载SRA数据,帮助manual。
pengzw@super-server:~$ fastq-dump -A SRR390728
②fasterq-dump 是通过都8102年了,还用fastq-dump,快换fasterq-dump吧,这篇文章而了解(因为我真的很懒,不喜欢看英文),虽然调了线程,感觉还是很慢,虽然lncRNA数据大,但是下载加转换花了6-8个小时,严重怀疑这是个假服务器或者假学校!!!
pengzw@super-server:~$ nohup fasterq-dump -e 24 SRR4042230 &
pengzw@super-server:~$ ls
biosoft data downloads fast.tmp ncbi project reference SRR4042230_1.fastq database examples.desktop miniconda3 nohup.out R script SRR4042230_2.fastq
注意:
1.重点参数是-e|threads, 用于选择使用多少线程进行运行,默认6个线程。
2.会在~ 生成一个临时文件夹fast.tmp,下载完成以后跳转到~有_1.fastq,_2.fastq。
3.下载SRA数据是直接accession号,fastq-dump 有-A。
问题:reads的名字会被加入SRR4042230.1等在@后面,相当于内容增加。
pengzw@super-server:~$ cat -n SRR4042230_1.fastq |head -n 8
1 @SRR4042230.1 HWI-ST1336:101:C3BDPACXX:1:1101:1412:2175 length=101
2 CCGTCCAATAATTTGATCAACGGAACAAGTTANCCTAGGGATAACAGCGCAATTCTATTCTAGAGTCCATATCGACAATAGAGNNNACGACCTCGATGTTG
3 +SRR4042230.1 HWI-ST1336:101:C3BDPACXX:1:1101:1412:2175 length=101
4 :=?+A@DFDFHD?FFEIIIIGBGIIIIFG?9C#1:BB;>GGHIIIIIIIII>AGEHGEEEHBECFBDDFB>CEB?;@?CCCCC###,,80<@BCB@CCAA>
5 @SRR4042230.2 HWI-ST1336:101:C3BDPACXX:1:1101:1497:2197 length=101
6 CTCTTCCGATCTTGTCAGCATCTTCCTCATCAACATCAAGCAGGAGAACATGGAAACGGAGCAGAAGCTCAGTGGGAAGCTATATCTGGTGGACCTGGCAG
7 +SRR4042230.2 HWI-ST1336:101:C3BDPACXX:1:1101:1497:2197 length=101
8 @@@DFFFFHHHHG>AEFHIHHHIGHHIJJJJIEHJGIJJJJIJIIIJGICGJIJGJJJJDHIIHEHHHBEED?CEBDC@ACDDDDA@CDCCBACDCDBBDD
fasterq-dump and fastq-dump两者的异同(文章末尾):
https://github.com/ncbi/sra-tools/wiki/HowTo:-fasterq-dump
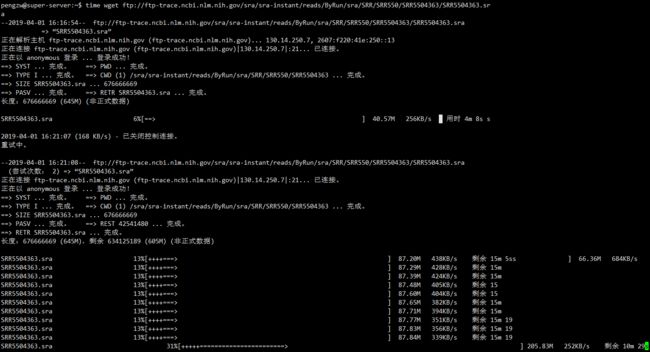
4.wget(亲测真的很断点啊,理解很深刻),看实际网速啦~我这边花了 26min。
wget -c ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR550/SRR5504363/SRR5504363.sra
太看得起我农的日常网速!!!
如果按SRR下载文件的话,可见ftp构成:
ftp://ftp.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR+登陆号前三位数字(550)+/SRR+完整登陆号(554360)
5.参考
http://www.cnblogs.com/ywliao/p/7356528.html
https://blog.csdn.net/qq_29300341/article/details/76137939
https://www.jianshu.com/p/19ae29e54c59
http://www.chenlianfu.com/?p=2319
https://www.jianshu.com/p/cf0a7b937413
Aspera connect 官方问答:https://support.asperasoft.com/hc/en-us/articles/216125898-Downloading-data-