- 迁移学习基础知识
zhooooooou
深度学习迁移学习人工智能机器学习
简介使用迁移学习的优势:1、能够快速的训练出一个理想的结果2、当数据集较小时也能训练出理想的效果。注意:在使用别人预训练的参数模型时,要注意别人的预处理方式。原理:对于浅层的网络结构,他们学习到的角点信息和纹理信息都是通用的,将学习好的浅层网络的信息迁移到新的网络中,这样新的网络也拥有了识别底层通用特征的能力,从而能加快网络学习新的数据集的高维特征。常见的迁移学习的方式:1、载入权重后训练所有参数
- 并发编程面试题四
2301_76231794
面试
1、ReentrantLock和synchronized的区别及使用的场景synchronized关键字(是悲观锁):自动管理:synchronized是Java提供的一种内置锁机制,使用简单,不需要显式地获取和释放锁。可重入性:同一个线程可以多次获取同一个锁而不被阻塞。不可中断:一旦一个线程开始等待获取锁,它不能被其他线程中断。内存可见性:synchronized块或方法提供了内存可见性的保证,
- 主从dns+nfs+nginx综合实验
睡不够觉
centoslinux运维
目录1.实验要求2.配置主DNS1安装bind软件编辑主配置文件编辑区域数据文件检查配置文件和区域文件是否有误重启服务测试dns1是否生效3.配置从DNS2安装bind编辑从DNS2的主配置文件防火墙放行测试dns24.配置nfs服务创建共享目录写入指定内容,并编辑共享配置文件5.web服务配置下载nginx软件挂载共享目录到nginx的默认页面6.客服端配置安装bind-utils客户端测试7.
- AUTOSAR与arxml的文档解析
dont__cry
c语言autosar
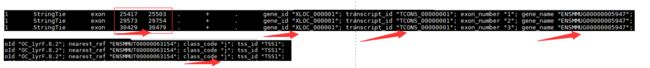
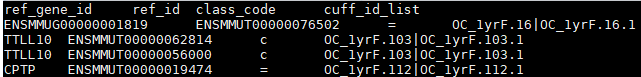
如下是文档脑图一、文档概述该文档是AUTOSAR经典平台的应用接口用户指南,主要解释**AlTable(应用接口表)**的结构、方法论及相关技术细节,帮助开发者理解如何通过标准化接口实现软件组件的互操作性。关键内容目的:定义并发布稳定、广泛接受的汽车电子系统应用接口,支持跨厂商软件组件的通信。结构:包含元模型表示、兼容性管理、生命周期状态、视图概念及XML生成规则等。二、核心概念与示例1.AUTO
- 提到一个项目的“验证LOV”属性?提到lov和list项目有什么区别?思维导图 代码示例(java 架构)
用心去追梦
listjava架构
验证LOV(ListofValues)属性在OracleForms中,LOV(ListofValues)是一种用于显示可供选择的值列表的组件。它通常与字段或项关联,允许用户从预定义的选项列表中选择一个值,而不是手动输入。验证LOV属性确保用户只能从LOV提供的选项中选择值,从而增强了数据输入的准确性和一致性。验证LOV属性定义:当设置为“是”时,表示该字段必须从LOV中选择值;如果用户尝试输入不在
- python 游戏开发cocos2d库安装与使用
范哥来了
pythoncocos2d开发语言
Cocos2d-x是一个广泛使用的开源游戏开发框架,支持多种编程语言,包括Python。对于Python开发者来说,通常使用的是Cocos2d-py或者更现代的Cocos2d-x的Python绑定版本。这里我将指导你如何安装和开始使用Cocos2d-py。安装步骤安装Python:确保你的系统上已经安装了Python3.x版本。你可以从Python官方网站下载最新版的Python。安装pip:pi
- 迁移学习入门
EmbodiedTech
人工智能大模型迁移学习人工智能机器学习
迁移学习1迁移学习的概念预训练模型定义:简单来说别人训练好的模型。一般预训练模型具备复杂的网络模型结构;一般是在大量的语料下训练完成的预训练语言模型的类别现在我们接触到的预训练语言模型,基本上都是基于transformer这个模型迭代而来的因此划分模型类别的时候,以transformer架构来划分:Encoder-Only:只有编码器部分的模型,代表:BERTDecoder-Only:只要解码器部
- https握手过程
给我个面子中不
https网络协议http
HTTPS(HyperTextTransferProtocolSecure)是HTTP的安全版本,它通过SSL/TLS协议对通信内容进行加密,确保数据在传输过程中的机密性、完整性和身份验证。以下是HTTPS握手过程的详细说明,以及它是如何保证信息安全的。1.HTTPS的核心机制HTTPS的安全性主要依赖于以下技术:加密:使用对称加密和非对称加密结合的方式,确保数据在传输过程中不被窃听。身份验证:通
- 通用具身智能机器人首次进厂造芯!
量子位
在全球半导体产业竞争日益激烈的背景下,半导体生产在部分高度依赖人力的生产环节,传统的智能化生产模式仍暴露出效率瓶颈与污染问题。人工操作易导致污染,且效率不高、污染控制难度大。如今,随着通用智能机器人的应用,这一难题正迎来全新解决方案。3月18日,国内领先的具身智能机器人公司智平方与全球知名科技创新企业吉利科技集团旗下浙江晶能微电子有限公司正式签署战略合作协议。双方将基于智平方自研的端到端具身大模型
- HarmonyOS TEXT 语音搜索场景学习和总结
架构教育
在HarmonyOS中实现语音搜索功能时,涉及到麦克风权限的申请、音频数据的采集、编码和传输等多个步骤。以下是对上述代码的详细解析和补充说明:麦克风权限的申请与检查在HarmonyOS中,使用麦克风需要申请ohos.permission.MICROPHONE权限。在代码中,通过GRPermissionsUtils.checkPermissions方法来检查和申请权限。如果权限被授予,则可以继续进行
- 代练代打护航系统小程序源码:游戏生态的“双刃剑”
vue.jsmysql
近年来,随着游戏产业的蓬勃发展,代练代打行业也逐渐兴起。代练代打护航系统小程序的出现,为这一行业提供了一个新的平台,也引发了关于其利弊的讨论。本文将深入分析代练代打护航系统小程序源码,探讨其对游戏生态的影响。源码获取地址!!!!请点击!!!代练代打护航系统小程序通常包含以下功能模块:用户模块:用户注册、登录、个人信息管理、实名认证等。订单模块:玩家发布订单、代练接单、订单管理、交易担保等。评价模块
- YashanDB 运行日志管理
数据库
本文内容来自YashanDB官网,原文内容请见https://doc.yashandb.com/yashandb/23.3/zh/%E6%95%B0%E6%8D%AE%...运行日志通过日志级别控制记录开关和记录策略,级别为OFF时不记录运行日志。通过设置RUN\_LOG\_FILE\_COUNT参数修改运行日志的保留个数,当达到该参数设置的值(默认10)时,系统会自动删除时间靠前的日志。日志文件
- SciER:首个大规模科学文档中的实体和关系抽取数据集
数据集
2024-10-28,为科学文档中的实体和关系抽取领域带来了突破,提供了一个包含106篇完整科学出版物、超过24,000个实体和12,000个关系的大规模数据集,这对于构建科学知识图谱和促进科学信息抽取技术的发展具有重要意义。数据集地址:SciER|科学信息提取数据集|人工智能数据集一、研究背景:在科学文档中,实体(如数据集、方法、任务)和它们之间的关系对于理解科学发现和推动研究进展至关重要。然而
- YashanDB表的紧急恢复
数据库
#表的紧急恢复对于无法通过闪回进行快速恢复的表,可使用YashanDB的物理备份恢复功能和导入、导出功能进行紧急恢复。操作流程如下:备份现有数据库的所有数据文件,以防止在此过程的剩余步骤中出错。将数据库备份还原到备库环境。至少应恢复以下内容:SYSTEM和SYSAUX表空间包含还原或回退段的表空间包含要检索的数据的独立表空间使用还原的备份控制文件对该备份执行不完全恢复,直到删除表之前。从数据库的临
- 程序员996写bug?这个AI工具让你头发越秃代码越香
后端
凌晨三点的写字楼里,小王第18次按下F5刷新浏览器,控制台又跳出了新的报错信息。咖啡杯底的褐色痕迹在显示器蓝光下格外刺眼,他突然想起入职时主管说的"程序员越秃越强",摸了摸发际线苦笑——原来这句话的潜台词是"用头发换代码"啊。直到上个月团建时,我发现隔壁工位的老张居然在团建现场掏出笔记本写代码。凑近一看,他正在用DeepSeek的智能提示功能自动补全单元测试。更气人的是,这厮今年居然还长出了新发茬
- HarmonyNext实战:基于ArkTS的高性能网络请求与缓存优化案例详解
harmonyos-next
HarmonyNext实战:基于ArkTS的高性能网络请求与缓存优化案例详解在现代移动应用开发中,网络请求是不可避免的核心功能之一。然而,频繁的网络请求不仅会增加服务器负担,还会影响应用的性能和用户体验。HarmonyOSNext提供了强大的网络请求框架和缓存机制,帮助开发者优化网络请求性能。本文将深入探讨如何在HarmonyOSNext中使用ArkTS实现高性能网络请求与缓存优化。通过一个完整的
- Promise.all() 和 Promise.allSettled()的区别
风无雨
#JavaScript前端javascript开发语言
特性Promise.all()Promise.allSettled()失败时行为任意一个Promise失败后立即终止,返回第一个错误等待所有Promise完成(无论成功或失败)返回值成功时返回结果数组;失败时返回第一个错误始终返回对象数组,每个对象包含status(状态)和value(成功值)或reason(错误)适用场景需要所有Promise必须成功(如并行请求依赖全部结果的场景)需要记录所有P
- HarmonyNext实战:基于ArkTS的跨设备文件同步与冲突解决案例详解
harmonyos-next
HarmonyNext实战:基于ArkTS的跨设备文件同步与冲突解决案例详解在现代多设备协同的场景中,文件同步是一个常见的需求。然而,跨设备文件同步往往会面临冲突问题,例如同一文件在不同设备上被同时修改。HarmonyOSNext提供了强大的分布式文件系统和冲突解决机制,帮助开发者实现高效、可靠的跨设备文件同步。本文将深入探讨如何在HarmonyOSNext中使用ArkTS实现跨设备文件同步与冲突
- 第十天-字符串:编程世界的文本基石
大橙子房
aipythonjava
在编程的广阔领域中,字符串是极为重要的数据类型,它就像一座桥梁,连接着人类的自然语言和计算机能够理解与处理的数字信息。下面,让我们深入探索字符串的世界。一、字符串简介字符串是由零个或多个字符组成的有序序列,它在程序中用于表示文本信息。在Python语言环境下,创建字符串简洁直观,例如:str="HelloWorld"。这里,str作为字符串变量名,就如同给一个装着文本内容的盒子贴上了标签;Hell
- 学习第十一天-树
大橙子房
ai学习
一、树的基础概念1.定义树是一种非线性数据结构,由n个有限节点组成层次关系集合。特点:有且仅有一个根节点其余节点分为若干互不相交的子树节点间通过父子关系连接2.关键术语术语定义节点包含数据和子节点引用的单元根节点树的起始节点,没有父节点子节点直接连接到父节点的节点叶子节点没有子节点的节点度节点拥有的子树数目树的高度从根节点到最远叶子节点的最长路径边数树的深度从根节点到当前节点的层数路径从根到某节点
- HarmonyNext实战:基于ArkTS的高性能数据流处理系统开发
harmonyos-next
HarmonyNext实战:基于ArkTS的高性能数据流处理系统开发引言在HarmonyNext生态系统中,数据流处理是一个关键的技术领域。本文将深入探讨如何使用ArkTS构建一个高性能的数据流处理系统,重点讲解流式数据处理、并发模型、内存管理以及性能优化等高级主题。我们将通过一个实际的案例——实时日志分析系统,来展示如何将这些技术应用到实际工程中。系统架构设计我们的实时日志分析系统需要处理来自多
- Linux修改/设置服务器ip地址
大橙子房
Linuxlinux服务器centos
1.用户切换到root用户su-#普通用户切换到root用户2.cd到network-scripts目录下cd/etc/sysconfig/network-scriptsll#ll查看文件目录#找到ifcfg-exx这个格式的文件,我这里的是ifcfg-ens33#大家可能都不相同,但是前面的ifcfg-exx这些是一样的3.编辑ifcfg-ens33(每个人的文件名都不一样,要自己看文件名是什么
- 如何在Linux中运行脚本
大橙子房
Linuxlinux运维服务器
1.首先在windos下,新建一个txt文件,2,编写脚本#!/bin/bash#编写helloworld脚本echo"HelloWorld!"3.将.txt后缀改成.sh4.上传到Linux5,执行,./hello.sh6.若显示权限不足,赋予权限chmod777./hello.sh7.若是报错坏的解释器:没有那个文件或目录的解决方法sed's/\r//'-ihello.sh原因:由于windo
- 工业机器人:现代工业的智能力量
机器人之树小风
机器人科技经验分享
在当今科技飞速发展的时代,工业机器人已成为现代工业不可或缺的重要组成部分。它如同一位不知疲倦、高度精准且功能强大的“智能工匠”,以其独特的魅力和卓越的能力,深刻地改变着工业生产的面貌。工业机器人是一种能够自动执行任务的可编程机械装置。它通常由机械臂、控制系统和编程装置等部分组成。机械臂是机器人的主体结构,能够实现多自由度的灵活运动,模拟人类手臂的各种动作。控制系统则犹如机器人的“大脑”,负责指挥和
- 鸿蒙Next,图片上传01(扩展02截图保存到相册)-组件截图另通过沙箱保存到相册
harmonyos
1.componentSnapShot组件截图的get方法,得到PixelMap像素图像数据@ohos.arkui.componentSnapshot(组件截图)-UI界面-ArkTSAPI-ArkUI(方舟UI框架)-应用框架-华为HarmonyOS开发者//进行截图constpixelMap=awaitcomponentSnapshot.get('share')2.ImagePacker打包P
- 使用LoRA微调LLaMA3
想胖的壮壮
深度学习人工智能
使用LoRA微调LLaMA3的案例案例概述在这个案例中,我们将使用LoRA微调LLaMA3模型,进行一个文本分类任务。我们将使用HuggingFace的Transformers库来完成这个过程。步骤一:环境搭建安装必要的Python包pipinstalltransformersdatasetstorch配置GPU环境确保你的环境中配置了CUDA和cuDNN,并验证GPU是否可用。importtor
- 复旦:LLM不同层位置编码缩放
大模型任我行
大模型-结构原理人工智能自然语言处理语言模型论文笔记
标题:Layer-SpecificScalingofPositionalEncodingsforSuperiorLong-ContextModeling来源:arXiv,2503.04355摘要尽管大型语言模型(LLM)在处理长上下文输入方面取得了重大进展,但它们仍然存在“中间丢失”问题,即上下文中间的关键信息往往不足或丢失。我们广泛的实验表明,这个问题可能源于旋转位置嵌入(RoPE)的快速长期衰
- SQL注入之ORDER BY注入
是小七呀呀
sql数据库网络安全
前言主要介绍本人在学习SQL注过程中遇到关于orderby这个字段的使用心得。一、ORDERBY是什么?orderby在mysql中就是用来对特定字段进行排序的,可以通过字段名进行指定,也可以通过数字1,2,3等进行指定,1就代表第一个字段,2就代表第二个字段,以此类推。因此,我我们就可以通过数字的方式,来判断查询的表格有几个字段,这样就避免了要知道表的字段名称的处境,为后续的sql注入打下基础。
- 元戎启行最新战略RoadAGI:所有移动智能体都将被AI驱动
量子位
2025年3月18日(北京时间),元戎启行作为国内人工智能企业代表,出席由NVIDIA主办的GTC大会。会上,公司CEO周光发表了技术主题演讲,展示了公司的最新战略布局RoadAGI,并发布道路通用人工智能平台——AISpark(以下简称”Spark平台”)。RoadAGI是元戎启行实现物理世界通用人工智能的关键一步,旨在让包括智能驾驶汽车在内的移动智能体,都具有在道路上自主行驶、与物理世界深度交
- 【python】可变、不可变数据类型
qianx77
pythonpythonnumpy开发语言
文章目录python可变、不可变数据类型一、什么是可变和不可变的数据类型?二、不可变类型1.数字2.字符3.元组三、可变类型4.列表需要注意的点5.集合5.字典6.补充-深拷贝和浅拷贝总结python可变、不可变数据类型用于记录python数据类型python我个人常用的数据就是数字、字符串、元组、列表、集合、字典,分为可变类型和不可变类型。一、什么是可变和不可变的数据类型?可变就是说在相同内存地
- jsonp 常用util方法
hw1287789687
jsonpjsonp常用方法jsonp callback
jsonp 常用java方法
(1)以jsonp的形式返回:函数名(json字符串)
/***
* 用于jsonp调用
* @param map : 用于构造json数据
* @param callback : 回调的javascript方法名
* @param filters : <code>SimpleBeanPropertyFilter theFilt
- 多线程场景
alafqq
多线程
0
能不能简单描述一下你在java web开发中需要用到多线程编程的场景?0
对多线程有些了解,但是不太清楚具体的应用场景,能简单说一下你遇到的多线程编程的场景吗?
Java多线程
2012年11月23日 15:41 Young9007 Young9007
4
0 0 4
Comment添加评论关注(2)
3个答案 按时间排序 按投票排序
0
0
最典型的如:
1、
- Maven学习——修改Maven的本地仓库路径
Kai_Ge
maven
安装Maven后我们会在用户目录下发现.m2 文件夹。默认情况下,该文件夹下放置了Maven本地仓库.m2/repository。所有的Maven构件(artifact)都被存储到该仓库中,以方便重用。但是windows用户的操作系统都安装在C盘,把Maven仓库放到C盘是很危险的,为此我们需要修改Maven的本地仓库路径。
- placeholder的浏览器兼容
120153216
placeholder
【前言】
自从html5引入placeholder后,问题就来了,
不支持html5的浏览器也先有这样的效果,
各种兼容,之前考虑,今天测试人员逮住不放,
想了个解决办法,看样子还行,记录一下。
【原理】
不使用placeholder,而是模拟placeholder的效果,
大概就是用focus和focusout效果。
【代码】
<scrip
- debian_用iso文件创建本地apt源
2002wmj
Debian
1.将N个debian-506-amd64-DVD-N.iso存放于本地或其他媒介内,本例是放在本机/iso/目录下
2.创建N个挂载点目录
如下:
debian:~#mkdir –r /media/dvd1
debian:~#mkdir –r /media/dvd2
debian:~#mkdir –r /media/dvd3
….
debian:~#mkdir –r /media
- SQLSERVER耗时最长的SQL
357029540
SQL Server
对于DBA来说,经常要知道存储过程的某些信息:
1. 执行了多少次
2. 执行的执行计划如何
3. 执行的平均读写如何
4. 执行平均需要多少时间
列名 &
- com/genuitec/eclipse/j2eedt/core/J2EEProjectUtil
7454103
eclipse
今天eclipse突然报了com/genuitec/eclipse/j2eedt/core/J2EEProjectUtil 错误,并且工程文件打不开了,在网上找了一下资料,然后按照方法操作了一遍,好了,解决方法如下:
错误提示信息:
An error has occurred.See error log for more details.
Reason:
com/genuitec/
- 用正则删除文本中的html标签
adminjun
javahtml正则表达式去掉html标签
使用文本编辑器录入文章存入数据中的文本是HTML标签格式,由于业务需要对HTML标签进行去除只保留纯净的文本内容,于是乎Java实现自动过滤。
如下:
public static String Html2Text(String inputString) {
String htmlStr = inputString; // 含html标签的字符串
String textSt
- 嵌入式系统设计中常用总线和接口
aijuans
linux 基础
嵌入式系统设计中常用总线和接口
任何一个微处理器都要与一定数量的部件和外围设备连接,但如果将各部件和每一种外围设备都分别用一组线路与CPU直接连接,那么连线
- Java函数调用方式——按值传递
ayaoxinchao
java按值传递对象基础数据类型
Java使用按值传递的函数调用方式,这往往使我感到迷惑。因为在基础数据类型和对象的传递上,我就会纠结于到底是按值传递,还是按引用传递。其实经过学习,Java在任何地方,都一直发挥着按值传递的本色。
首先,让我们看一看基础数据类型是如何按值传递的。
public static void main(String[] args) {
int a = 2;
- ios音量线性下降
bewithme
ios音量
直接上代码吧
//second 几秒内下降为0
- (void)reduceVolume:(int)second {
KGVoicePlayer *player = [KGVoicePlayer defaultPlayer];
if (!_flag) {
_tempVolume = player.volume;
- 与其怨它不如爱它
bijian1013
选择理想职业规划
抱怨工作是年轻人的常态,但爱工作才是积极的心态,与其怨它不如爱它。
一般来说,在公司干了一两年后,不少年轻人容易产生怨言,除了具体的埋怨公司“扭门”,埋怨上司无能以外,也有许多人是因为根本不爱自已的那份工作,工作完全成了谋生的手段,跟自已的性格、专业、爱好都相差甚远。
- 一边时间不够用一边浪费时间
bingyingao
工作时间浪费
一方面感觉时间严重不够用,另一方面又在不停的浪费时间。
每一个周末,晚上熬夜看电影到凌晨一点,早上起不来一直睡到10点钟,10点钟起床,吃饭后玩手机到下午一点。
精神还是很差,下午像一直野鬼在城市里晃荡。
为何不尝试晚上10点钟就睡,早上7点就起,时间完全是一样的,把看电影的时间换到早上,精神好,气色好,一天好状态。
控制让自己周末早睡早起,你就成功了一半。
有多少个工作
- 【Scala八】Scala核心二:隐式转换
bit1129
scala
Implicits work like this: if you call a method on a Scala object, and the Scala compiler does not see a definition for that method in the class definition for that object, the compiler will try to con
- sudoku slover in Haskell (2)
bookjovi
haskellsudoku
继续精简haskell版的sudoku程序,稍微改了一下,这次用了8行,同时性能也提高了很多,对每个空格的所有解不是通过尝试算出来的,而是直接得出。
board = [0,3,4,1,7,0,5,0,0,
0,6,0,0,0,8,3,0,1,
7,0,0,3,0,0,0,0,6,
5,0,0,6,4,0,8,0,7,
- Java-Collections Framework学习与总结-HashSet和LinkedHashSet
BrokenDreams
linkedhashset
本篇总结一下两个常用的集合类HashSet和LinkedHashSet。
它们都实现了相同接口java.util.Set。Set表示一种元素无序且不可重复的集合;之前总结过的java.util.List表示一种元素可重复且有序
- 读《研磨设计模式》-代码笔记-备忘录模式-Memento
bylijinnan
java设计模式
声明: 本文只为方便我个人查阅和理解,详细的分析以及源代码请移步 原作者的博客http://chjavach.iteye.com/
import java.util.ArrayList;
import java.util.List;
/*
* 备忘录模式的功能是,在不破坏封装性的前提下,捕获一个对象的内部状态,并在对象之外保存这个状态,为以后的状态恢复作“备忘”
- 《RAW格式照片处理专业技法》笔记
cherishLC
PS
注意,这不是教程!仅记录楼主之前不太了解的
一、色彩(空间)管理
作者建议采用ProRGB(色域最广),但camera raw中设为ProRGB,而PS中则在ProRGB的基础上,将gamma值设为了1.8(更符合人眼)
注意:bridge、camera raw怎么设置显示、输出的颜色都是正确的(会读取文件内的颜色配置文件),但用PS输出jpg文件时,必须先用Edit->conv
- 使用 Git 下载 Spring 源码 编译 for Eclipse
crabdave
eclipse
使用 Git 下载 Spring 源码 编译 for Eclipse
1、安装gradle,下载 http://www.gradle.org/downloads
配置环境变量GRADLE_HOME,配置PATH %GRADLE_HOME%/bin,cmd,gradle -v
2、spring4 用jdk8 下载 https://jdk8.java.
- mysql连接拒绝问题
daizj
mysql登录权限
mysql中在其它机器连接mysql服务器时报错问题汇总
一、[running]
[email protected]:~$mysql -uroot -h 192.168.9.108 -p //带-p参数,在下一步进行密码输入
Enter password: //无字符串输入
ERROR 1045 (28000): Access
- Google Chrome 为何打压 H.264
dsjt
applehtml5chromeGoogle
Google 今天在 Chromium 官方博客宣布由于 H.264 编解码器并非开放标准,Chrome 将在几个月后正式停止对 H.264 视频解码的支持,全面采用开放的 WebM 和 Theora 格式。
Google 在博客上表示,自从 WebM 视频编解码器推出以后,在性能、厂商支持以及独立性方面已经取得了很大的进步,为了与 Chromium 现有支持的編解码器保持一致,Chrome
- yii 获取控制器名 和方法名
dcj3sjt126com
yiiframework
1. 获取控制器名
在控制器中获取控制器名: $name = $this->getId();
在视图中获取控制器名: $name = Yii::app()->controller->id;
2. 获取动作名
在控制器beforeAction()回调函数中获取动作名: $name =
- Android知识总结(二)
come_for_dream
android
明天要考试了,速速总结如下
1、Activity的启动模式
standard:每次调用Activity的时候都创建一个(可以有多个相同的实例,也允许多个相同Activity叠加。)
singleTop:可以有多个实例,但是不允许多个相同Activity叠加。即,如果Ac
- 高洛峰收徒第二期:寻找未来的“技术大牛” ——折腾一年,奖励20万元
gcq511120594
工作项目管理
高洛峰,兄弟连IT教育合伙人、猿代码创始人、PHP培训第一人、《细说PHP》作者、软件开发工程师、《IT峰播》主创人、PHP讲师的鼻祖!
首期现在的进程刚刚过半,徒弟们真的很棒,人品都没的说,团结互助,学习刻苦,工作认真积极,灵活上进。我几乎会把他们全部留下来,现在已有一多半安排了实际的工作,并取得了很好的成绩。等他们出徒之日,凭他们的能力一定能够拿到高薪,而且我还承诺过一个徒弟,当他拿到大学毕
- linux expect
heipark
expect
1. 创建、编辑文件go.sh
#!/usr/bin/expect
spawn sudo su admin
expect "*password*" { send "13456\r\n" }
interact
2. 设置权限
chmod u+x go.sh 3.
- Spring4.1新特性——静态资源处理增强
jinnianshilongnian
spring 4.1
目录
Spring4.1新特性——综述
Spring4.1新特性——Spring核心部分及其他
Spring4.1新特性——Spring缓存框架增强
Spring4.1新特性——异步调用和事件机制的异常处理
Spring4.1新特性——数据库集成测试脚本初始化
Spring4.1新特性——Spring MVC增强
Spring4.1新特性——页面自动化测试框架Spring MVC T
- idea ubuntuxia 乱码
liyonghui160com
1.首先需要在windows字体目录下或者其它地方找到simsun.ttf 这个 字体文件。
2.在ubuntu 下可以执行下面操作安装该字体:
sudo mkdir /usr/share/fonts/truetype/simsun
sudo cp simsun.ttf /usr/share/fonts/truetype/simsun
fc-cache -f -v
- 改良程序的11技巧
pda158
技巧
有很多理由都能说明为什么我们应该写出清晰、可读性好的程序。最重要的一点,程序你只写一次,但以后会无数次的阅读。当你第二天回头来看你的代码 时,你就要开始阅读它了。当你把代码拿给其他人看时,他必须阅读你的代码。因此,在编写时多花一点时间,你会在阅读它时节省大量的时间。
让我们看一些基本的编程技巧:
尽量保持方法简短
永远永远不要把同一个变量用于多个不同的
- 300个涵盖IT各方面的免费资源(下)——工作与学习篇
shoothao
创业免费资源学习课程远程工作
工作与生产效率:
A. 背景声音
Noisli:背景噪音与颜色生成器。
Noizio:环境声均衡器。
Defonic:世界上任何的声响都可混合成美丽的旋律。
Designers.mx:设计者为设计者所准备的播放列表。
Coffitivity:这里的声音就像咖啡馆里放的一样。
B. 避免注意力分散
Self Co
- 深入浅出RPC
uule
rpc
深入浅出RPC-浅出篇
深入浅出RPC-深入篇
RPC
Remote Procedure Call Protocol
远程过程调用协议
它是一种通过网络从远程计算机程序上请求服务,而不需要了解底层网络技术的协议。RPC协议假定某些传输协议的存在,如TCP或UDP,为通信程序之间携带信息数据。在OSI网络通信模型中,RPC跨越了传输层和应用层。RPC使得开发