环状序列(紫书例题3-6)
问题描述:
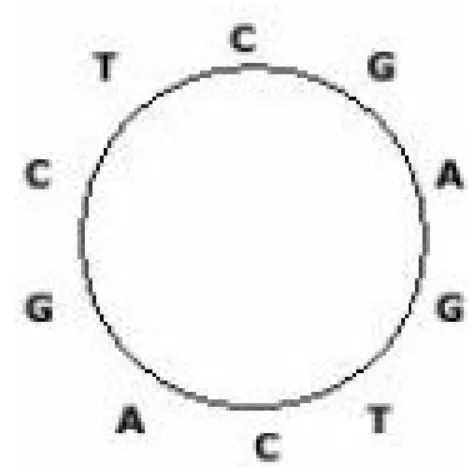
长度为n的环状串有n种表示法,分别为某个位置开始顺时针得到。CGAGTCAGCT,GAGTCAGCTC,AGTCAGCTCG等。在这些表示法中,字典序最小的称为“最小表示”。输入一个长度为n(n<=100)的环状DNA串(只包含A、C、G、T这4种字符)的一种表示法,你的任务是输出该环状串的最小表示。例如,CTCC的最小表示是CCCT,CGAGTCAGCT的最小表示为AGCTCGAGTC。
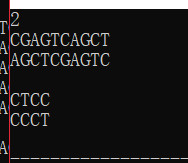
样例输入:
2
CGAGTCAGCT
CTCC
样例输出:
AGCTCGAGTC
CCCT
分析:
一开始理解这道题的时候很吃力,看了好久都不知道它想要干嘛,后面才知道原来是在一串成环的 DNA像字典排序一样找出其中最小的表示,也就是要按照字母排列寻找正确的起点。
难点:
1.当找到的起点并非是输入的DNA序列的第一个字母,怎么让“断掉“”的重新“续上”:
(起点s+增值i)%输入DNA的长度
2.当符合/不符合时,应怎么递增继续判断/切换起点:
符合时,记录当前i的值作为起点,用此起点继续跟该点以后做起点的序列作比较;不符合时,直接跳过;直到到达该DNA的结尾。
判断以不同起点的序列的字典序,从起点开始比较,当增加相同单位处的字母不同时,字母小的序列就小,相同时继续判断两序列的下一个位置字母的大小关系,直到回到原起点。
代码如下:
#include
#include
using namespace std;
int comp(string s,int s1,int s2){
int num=s.size();
for(int i=0;is[(s2+i)%num];
}
return 0;
}
int main(){
int n;
string dna;
cin>>n;
while(n--){
cin>>dna;
int start=0,num=dna.size();
for(int i=1;i 相似的,还有:
DNA序列(紫书习题3-7)
题目描述:
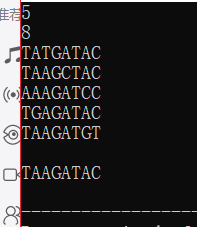
输入m个长度均为n的DNA序列,求一个DNA序列,到所有序列的总Hamming距离尽量小。两个等长字符串的Hamming距离等于字符不同的位置个数,例如,ACGT和GCGA的Hamming距离为2(左数第1,4字符不同)。
输入整数m和n(4<=m<=50,4<=n<=1000),以及m个长度为n的DNA序列(只是包含字母A,C,G,T),输出到m个序列的Hamming距离和最小的DNA序列和对应的距离。如有多解,要求为字典序最小的解。例如,对于下面5个DNA序列,最优解为TAAGATAC。
分析:
在上面那题对字典序有了了解以后,理解这道题就很简单了。所谓Hamming距离和最小意思就是跟所给出的所有序列的字符不同的位置个数总和最小,直接寻找在所有序列相同位置中出现次数最多的字符就可以找出这个序列了。又由于规定了多个解的时候,题目要求字典序最小,因此判断时可以用if-else结构按照字典顺序进行判断。
代码如下:
#include
#include
using namespace std;
int main(){
int m,n,max,a,c,g,t;
string dna;
cin>>m>>n;
char str[m][n];
for(int i=0;i>str[i];
}
for(int i=0;imax)max=c;
if(g>max)max=g;
if(t>max)max=t;
if(max==a)dna+='A';
else if(max==c)dna+='C';
else if(max==g)dna+='G';
else if(max==t)dna+='T';
}
cout<<"\n"<