单细胞测序数据的降维方法及细胞亚型鉴定聚类方法总结

随着单细胞测序技术的发展,每个研究或实验中测定的细胞数量在显著增加。现在很多单细胞研究中,少则产生几百,多则产生几十万的细胞数量,甚至更多。其中,细胞亚型(cell subtype or cell subpopulations)的鉴定是单细胞测序技术一个非常重要的基础应用。但由于单细胞测序数据通常涉及到很多细胞,而每个细胞中的基因数量又可能是几万个,所以,单细胞测序数据是一个高维的复杂数据。
为了有效地对单细胞测序数据进行各种处理分析,特别是细胞亚型的鉴定,通常需要首先对单细胞测序数据进行降维。单细胞测序数据的降维方法主要可分为两大类(微信公众号:AIPuFuBio):
1、Dimensionality reduction(降维)。降维方法通常是把高维数据通过优化保留原始数据中的关键特征后投射到低维空间,从而可以通过二维或三维的形式把数据展示出来。
常用的降维方法有:
1)PCA(Principle Component Analysis),主成分分析,是一种线性的降维方法;
2)t-SNE(T-distributed stochastic neighbor embedding),是一种非线性的降维方法;
3)UMAP (uniform manifold approximation and projection) (Becht et al., 2018, Nat. Biotechnol.),
4)scvis (Ding et al., 2018, Nat. Commun.)
其中PCA和t-SNE被广泛应用于已发表的单细胞测序相关文章中。特别注意,PCA和t-SNE是降维的方法,并不是聚类方法。

2、Feature selection(特征选择),主要是通过去除信息含量少的基因而保留信息含量最多的基因来降低数据的维度。
常用的Feature selection的方法有:
1)基于先验信息的方法(如已知细胞的亚型)。比如通过SCDE软件鉴定已知不同细胞亚型间的差异表达基因,然后再基于差异表达基因来聚类分析等。
2)非监督方法。又可细分为:
(i) 基于highly variable genes (HVG) ;
(ii) 基于spike-in,如scLVM (Buettner et al., 2015)和BASiCS (Vallejos et al., 2015)等;
(iii)基于 dropout,如M3Drop (Andrews and Hemberg, 2018)。
单细胞测序数据细胞亚型鉴定方法(更多请见AIPuFu:www.aipufu.com)
1、监督的方法。比如基于特定细胞亚型的已知marker基因来聚类分析。
2、非监督的方法(unsupervised clustering)。又可细分为:
(i) k-means,通常可结合PCA和t-SNE等来使用;
(ii) hierarchical clustering,运行速度比K-means要慢;
(iii) density-based clustering,需要基于大样本才能提高聚类的精度;
(iv) graph-based clustering,是density-based clustering的一个延伸,可以应用于上百万的细胞数量。
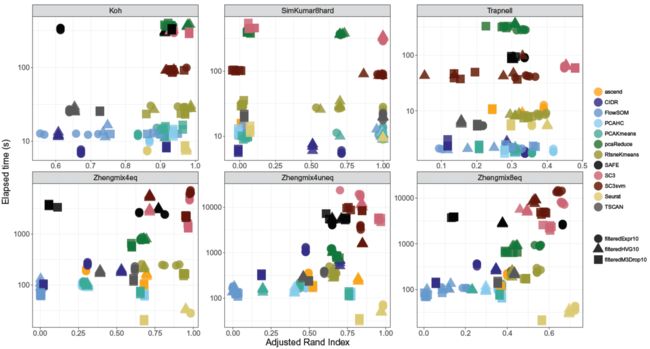
***不同细胞亚型鉴定聚类方法运行时间和具体性能的比较***
因此,从上面的图中可知,不同的聚类方法所具备的特点可能不一样,有些聚类方法运行时间短,有些聚类方法的结果更准确。可根据具体的数据情况,选择相应的软件。建议选择最新发表、且发表在高质量期刊的软件哦~(**更多经典知识,可关注大型免费综合生物信息学资源和工具平台AIPuFu:www.aipufu.com)。
希望今天的内容对大家有用哦,会持续更新的,欢迎留言~~