数据分析入门之使用scikit-learn构建模型
文章目录
- 1、使用sklearn转换器处理数据
- 1.1、加载datasets模块中数据集
- 1.1.1、加载波士顿房价信息
- 1.2、将数据集划分为训练集和测试集
- 1.2.1、常用划分方式
- 1.2.2、K折交叉验证法
- 1.2.3、train_test_split函数
- 1.3、使用sklearn转换器进行数据预处理与降维
- 1.3.1、sklearn转换器三个方法
- 1.3.2、sklearn转换器
- 1.3.3、sklearn部分预处理函数与其作用
- 1.3.4、PCA降维算法函数
- 1.3.5、PCA降维算法函数常用参数及其作用
- 2、构建并评价聚类模型
- 2.1、使用sklearn估计器构建聚类模型
- 2.1.1、聚类
- 2.1.2、聚类方法类别
- 2.1.3、cluster提供的聚类算法及其适用范围
- 2.1.4、sklearn估计器
- 2.2、评价聚类模型
- 2.2.1、聚类模型评价指标
- 3、构建并评价分类模型
- 3.1、使用sklearn估计器构建分类模型
- 3.1.1、分类算法的实现过程
- 3.1.2、sklearn库常用分类算法函数
- 3.2、评价分类模型
- 3.2.1、分类模型的评价指标
- 3.2.2、ROC曲线
- 4、构建并评价回归模型
- 4.1、回归分析方法
- 4.2、常用的回归模型
- 4.3、sklearn库常用回归算法函数
- 4.4、回归模型评价指标
1、使用sklearn转换器处理数据
1.1、加载datasets模块中数据集
- sklearn库的datasets模块集成了部分数据分析的经典数据集,可以使用这些数据集进行数据预处理,建模等操作,熟悉sklearn的数据处理流程和建模流程。
- datasets模块常用数据集的加载函数与解释如下表所示:
| 数据集加载函数 | 数据集任务类型 |
|---|---|
| load_ boston | 回归 |
| fetch_california_housing | 回归 |
| load_digits | 分类 |
| load_wine | 分类 |
| load_breast_cancer | 分类,聚类 |
| load_iris | 分类,聚类 |
1.1.1、加载波士顿房价信息
from sklearn.datasets import load_boston
data = load_boston()
(1)查看键值对:
data.keys()
dict_keys(['data', 'target', 'feature_names', 'DESCR', 'filename'])
(2)查看数据:
data['data']
array([[6.3200e-03, 1.8000e+01, 2.3100e+00, ..., 1.5300e+01, 3.9690e+02,
4.9800e+00],
[2.7310e-02, 0.0000e+00, 7.0700e+00, ..., 1.7800e+01, 3.9690e+02,
9.1400e+00],
[2.7290e-02, 0.0000e+00, 7.0700e+00, ..., 1.7800e+01, 3.9283e+02,
4.0300e+00],
...,
[6.0760e-02, 0.0000e+00, 1.1930e+01, ..., 2.1000e+01, 3.9690e+02,
5.6400e+00],
[1.0959e-01, 0.0000e+00, 1.1930e+01, ..., 2.1000e+01, 3.9345e+02,
6.4800e+00],
[4.7410e-02, 0.0000e+00, 1.1930e+01, ..., 2.1000e+01, 3.9690e+02,
7.8800e+00]])
1.2、将数据集划分为训练集和测试集
1.2.1、常用划分方式
- 在数据分析过程中,为了保证模型在实际系统中能够起到预期作用,一般需要将样本分成独立的三部分:
- 训练集(train set):用于估计模型。
- 验证集(validation set):用于确定网络结构或者控制模型复杂程度的参数。
- 测试集(test set):用于检验最优的模型的性能。
- 典型的划分方式是训练集占总样本的50%,而验证集和测试集各占25%。
1.2.2、K折交叉验证法
- 当数据总量较少的时候,使用上面的方法将数据划分为三部分就不合适了。
- 常用的方法是留少部分做测试集,然后对其余N个样本采用K折交叉验证法,基本步骤如下:
- 将样本打乱,均匀分成K份。
- 轮流选择其中K-1份做训练,剩余的一份做验证。
- 计算预测误差平方和,把K次的预测误差平方和的均值作为选择最优模型结构的依据。
1.2.3、train_test_split函数
- sklearn的model_selection模块提供了train_test_split函数,能够对数据集进行拆分,其使用格式如下。
- sklearn.model_selection.train_test_split(*arrays, **options)
- train_test_split函数根据传入的数据,分别将传入的数据划分为训练集和测试集。
- 如果传入的是1组数据,那么生成的就是这一组数据随机划分后训练集和测试集,总共2组。如果传入的是2组数据,则生成的训练集和测试集分别2组,总共4组。
from sklearn.model_selection import train_test_split
# 分别返回这四个值:train_data,test_data,train_target,test_target
#取20%作为测试集和训练集
train_test_split(data['data'], data['target'], test_size=0.2)
- 结果分别对应这四个值:train_data,test_data,train_target,test_target
[array([[4.15292e+01, 0.00000e+00, 1.81000e+01, ..., 2.02000e+01,
3.29460e+02, 2.73800e+01],
...,
[8.05579e+00, 0.00000e+00, 1.81000e+01, ..., 2.02000e+01,
3.52580e+02, 1.81400e+01],
array([[1.7090e-02, 9.0000e+01, 2.0200e+00, ..., 1.7000e+01, 3.8446e+02,
4.5000e+00],
...,
[2.7290e-02, 0.0000e+00, 7.0700e+00, ..., 1.7800e+01, 3.9283e+02,
4.0300e+00]]),
array([ 8.5, 17.5, 24.3, 24.8, 24.1, 22.3, 16.5, 19.4, 46. , 15.6, 14.3,
33.2, 11. , 44.8, 19.1, 24.8, 50. , 5.6, 15.6, 23. , 22.6, 31.7,
...,
13.3, 19.4, 50. , 21.2, 13.1, 20.6, 23.1, 24.8, 11.8, 21.4, 34.9,
15. , 13.1, 23.9, 19.4, 13.9, 13.8, 14.9, 23.9]),
array([30.1, 24.3, 34.6, 10.4, 15.7, 16.2, 14.1, 22.9, 21.1, 19.5, 18.5,
27.9, 28.7, 20.8, 14.1, 28.7, 19.6, 20.4, 43.1, 31.1, 15.4, 18. ,
...,
20.6, 10.5, 22.8, 18.8, 25. , 25.3, 50. , 13.8, 37. , 18.3, 23.9,
11.9, 23.3, 34.7])]
- train_test_split是最常用的数据划分方法,在model_selection模块中还提供了其他数据集划分的函数,如PredefinedSplit,ShuffleSplit等。
1.3、使用sklearn转换器进行数据预处理与降维
1.3.1、sklearn转换器三个方法
- sklearn把相关的功能封装为转换器(transformer)。使用sklearn转换器能够实现对传入的NumPy数组进行标准化处理,归一化处理,二值化处理,PCA降维等操作。转换器主要包括三个方法:
| 方法名称 | 说明 |
|---|---|
| fit | fit方法主要通过分析特征和目标值,提取有价值的信息,这些信息可以是统计量,也可以是权值系数等 |
| transform | transform方法主要用来对特征进行转换。从可利用信息的角度可分为无信息转换和有信息转换。无信息转换是指不利用任何其他信息进行转换,比如指数和对数函数转换等。有信息转换根据是否利用目标值向量又可分为无监督转换和有监督转换。无监督转换指只利用特征的统计信息的转换,比如标准化和PCA降维等。有监督转换指既利用了特征信息又利用了目标值信息的转换,比如通过模型选择特征和LDA降维等 |
| fit_transform | fit_transform方法就是先调用fit方法,然后调用transform方法 |
1.3.2、sklearn转换器
- 在数据分析过程中,各类特征处理相关的操作都需要对训练集和测试集分开操作,需要将训练集的操作规则,权重系数等应用到测试集中。
- 如果使用pandas,则应用至测试集的过程相对烦琐,使用sklearn转换器可以解决这一困扰。
train_data,test_data,train_target,test_target = train_test_split(data['data'], data['target'], test_size=0.2)
from sklearn.preprocessing import MinMaxScaler
model = MinMaxScaler().fit(train_data)
train_data_mms = model.transform(train_data)
test_data_mms = model.transform(test_data)
model.min_
array([-7.10352762e-05, 0.00000000e+00, -2.74074074e-02, 0.00000000e+00,
-7.92181070e-01, -6.89581720e-01, -2.98661174e-02, -1.02719857e-01,
-4.34782609e-02, -3.56870229e-01, -1.34042553e+00, -6.38977636e-03,
-4.77373068e-02])
1.3.3、sklearn部分预处理函数与其作用
| 函数名称 | 说明 |
|---|---|
| MinMaxScaler | 对特征进行离差标准化 |
| StandardScaler | 对特征进行标准差标准化 |
| Normalizer | 对特征进行归一化 |
| Binarizer | 对定量特征进行二值化处理 |
| OneHotEncoder | 对定性特征进行独热编码处理 |
| FunctionTransformer | 对特征进行自定义函数变换 |
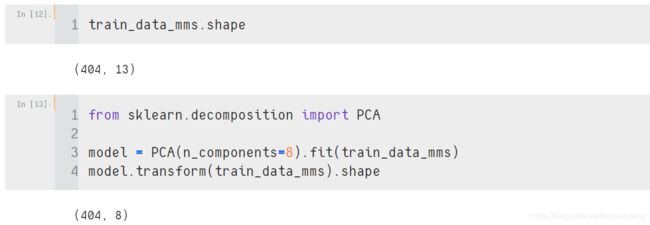
1.3.4、PCA降维算法函数
- sklearn除了提供基本的特征变换函数外,还提供了降维算法,特征选择算法,这些算法的使用也是通过转换器的方式。
from sklearn.decomposition import PCA
model = PCA(n_components=8).fit(train_data_mms)
model.transform(train_data_mms).shape
1.3.5、PCA降维算法函数常用参数及其作用
| 函数名称 | 说明 |
|---|---|
| n_components | 接收None,int,float或string。未指定时,代表所有特征均会被保留下来;如果为int,则表示将原始数据降低到n个维度;如果为float,同时svd_solver参数等于full;赋值为string,比如n_components=‘mle’,将自动选取特征个数n,使得满足所要求的方差百分比。默认为None |
| copy | 接收bool。代表是否在运行算法时将原始数据复制一份,如果为True,则运行后,原始数据的值不会有任何改变;如果为False,则运行PCA算法后,原始训练数据的值会发生改变。默认为True |
| whiten | 接收boolean。表示白化,所谓白化,就是对降维后的数据的每个特征进行归一化,让方差都为1。默认为False |
| svd_solver | 接收string {‘auto’, ‘full’, ‘arpack’, ‘randomized’}。代表使用的SVD算法。randomized一般适用于数据量大,数据维度多,同时主成分数目比例又较低的PCA降维,它使用了一些加快SVD的随机算法。full是使用SciPy库实现的传统SVD算法。arpack和randomized的适用场景类似,区别是randomized使用的是sklearn自己的SVD实现,而arpack直接使用了SciPy库的sparse SVD实现。auto则代表PCA类会自动在上述三种算法中去权衡,选择一个合适的SVD算法来降维。默认为auto |
2、构建并评价聚类模型
2.1、使用sklearn估计器构建聚类模型
2.1.1、聚类
from sklearn.datasets import load_iris
from sklearn.cluster import KMeans
data = load_iris()
data.keys()
dict_keys(['data', 'target', 'target_names', 'DESCR', 'feature_names', 'filename'])
(2)降维聚类
model = KMeans(n_clusters=3).fit(data['data'])
KMeans(algorithm='auto', copy_x=True, init='k-means++', max_iter=300,
n_clusters=3, n_init=10, n_jobs=None, precompute_distances='auto',
random_state=None, tol=0.0001, verbose=0)
(3)查看聚类标签
array([1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 2, 2, 0, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 0, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2,
2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 0, 2, 0, 0, 0, 0, 2, 0, 0, 0,
0, 0, 0, 2, 2, 0, 0, 0, 0, 2, 0, 2, 0, 2, 0, 0, 2, 2, 0, 0, 0, 0,
0, 2, 0, 0, 0, 0, 2, 0, 0, 0, 2, 0, 0, 0, 2, 0, 0, 2])
(4)聚类中心
model.cluster_centers_
array([[6.85 , 3.07368421, 5.74210526, 2.07105263],
[5.006 , 3.428 , 1.462 , 0.246 ],
[5.9016129 , 2.7483871 , 4.39354839, 1.43387097]])
(5) 查看聚类效果
import matplotlib.pyplot as plt
for i in range(3):
plt.scatter(data['data'][model.labels_ == i, 0], data['data'][model.labels_ == i, 1])
plt.show()
2.1.2、聚类方法类别
| 算法类别 | 包括的主要算法 |
|---|---|
| 划分(分裂)方法 | K-Means算法(K-平均),K-MEDOIDS算法(K-中心点)和CLARANS算法(基于选择的算法) |
| 层次分析方法 | BIRCH算法(平衡迭代规约和聚类),CURE算法(代表点聚类)和CHAMELEON算法(动态模型) |
| 基于密度的方法 | DBSCAN算法(基于高密度连接区域),DENCLUE算法(密度分布函数)和OPTICS算法(对象排序识别) |
| 基于网格的方法 | STING算法(统计信息网络),CLIOUE算法(聚类高维空间)和WAVE-CLUSTER算法(小波变换) |
2.1.3、cluster提供的聚类算法及其适用范围
- sklearn常用的聚类算法模块cluster提供的聚类算法及其适用范围如下所示:
| 参数名称 | 参数 | 适用范围 | 距离度量 |
|---|---|---|---|
| KMeans | 簇数 | 可用于样本数目很大,聚类数目中等的场景 | 点之间的距离 |
| Spectral clustering | 簇数 | 可用于样本数目中等,聚类数目较小的场景 | 图距离 |
| Ward hierarchical clustering | 簇数 | 可用于样本数目较大,聚类数目较大的场景 | 点之间的距离 |
| Agglomerative clustering | 簇数,链接类型,距离 | 可用于样本数目较大,聚类数目较大的场景 | 任意成对点线图间的距离 |
| DBSCAN | 半径大小,最低成员数目 | 可用于样本数目很大,聚类数目中等的场景 | 最近的点之间的距离 |
| Birch | 分支因子,阈值,可选全局集群 | 可用于样本数目很大,聚类数目较大的场景 | 点之间的欧式距离 |
2.1.4、sklearn估计器
- 聚类算法实现需要sklearn估计器(estimator)。sklearn估计器和转换器类似,拥有fit和predict两个方法。两个方法的作用如下:
| 方法名称 | 说明 |
|---|---|
| fit | fit方法主要用于训练算法。该方法可接收用于有监督学习的训练集及其标签两个参数,也可以接收用于无监督学习的数据 |
| predict | predict用于预测有监督学习的测试集标签,亦可以用于划分传入数据的类别 |
2.2、评价聚类模型
2.2.1、聚类模型评价指标
- 聚类评价的标准是组内的对象相互之间是相似的(相关的),而不同组中的对象是不同的(不相关的)。即组内的相似性越大,组间差别越大,聚类效果就越好。sklearn的metrics模块提供的聚类模型评价指标。
| 方法名称 | 真实值 | 最佳值 | sklearn函数 |
|---|---|---|---|
| ARI评价法(兰德系数) | 需要 | 1.0 | adjusted_rand_score |
| AMI评价法(互信息) | 需要 | 1.0 | adjusted_mutual_info_score |
| V-measure评分 | 需要 | 1.0 | completeness_score |
| FMI评价法 | 需要 | 1.0 | fowlkes_mallows_score |
| 轮廓系数评价法 | 不需要 | 畸变程度最大 | silhouette_score |
| Calinski-Harabasz指数评价法 | 不需要 | 相较最大 | calinski_harabaz_score |
from sklearn.metrics import silhouette_score
for k in range(2,9):#聚类必须要两类以上
model = KMeans(n_clusters=k).fit(data['data'])
print (k, silhouette_score(data['data'], model.labels_))
2 0.681046169211746
3 0.5528190123564091
4 0.4980505049972867
5 0.4887488870931048
6 0.3678464984712235
7 0.34974797998286666
8 0.3519340715227998
- k = 2 的聚类效果最好,值越接近1,效果越理想。
3、构建并评价分类模型
3.1、使用sklearn估计器构建分类模型
3.1.1、分类算法的实现过程
- 在数据分析领域,分类算法有很多,其原理千差万别,有基于样本距离的最近邻算法,有基于特征信息熵的决策树,有基于bagging的随机森林,有基于boosting的梯度提升分类树,但其实现的过程相差不大。过程如图所示:

3.1.2、sklearn库常用分类算法函数
- sklearn中提供的分类算法非常多,分别存在于不同的模块中。常用的分类算法如下表所示:
| 模块名称 | 函数名称 | 算法名称 |
|---|---|---|
| linear_model | LogisticRegression | 逻辑斯蒂回归 |
| svm | SVC | 支持向量机 |
| neighbors | KNeighborsClassifier | K最近邻分类 |
| naive_bayes | GaussianNB | 高斯朴素贝叶斯 |
| tree | DecisionTreeClassifier | 分类决策树 |
| ensemble | RandomForestClassifier | 随机森林分类 |
| ensemble | GradientBoostingClassifier | 梯度提升分类树 |
(1) 导入乳腺癌数据
from sklearn.datasets import load_breast_cancer
data = load_breast_cancer()
data.keys()
dict_keys(['data', 'target', 'target_names', 'DESCR', 'feature_names', 'filename'])
(2) 切分训练集和测试集
from sklearn.model_selection import train_test_split
x = data['data']
y = data['target']
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2)
import numpy as np
a = x_train.max(axis=0)
b = np.int32(a)
print (a)
print (b)
[2.742e+01 3.381e+01 1.869e+02 2.501e+03 1.447e-01 3.454e-01 4.268e-01
2.012e-01 3.040e-01 9.744e-02 2.547e+00 4.885e+00 1.865e+01 5.422e+02
3.113e-02 1.354e-01 3.038e-01 4.090e-02 7.895e-02 1.792e-02 3.604e+01
4.954e+01 2.512e+02 4.254e+03 2.184e-01 1.058e+00 1.252e+00 2.910e-01
6.638e-01 2.075e-01]
[ 27 33 186 2501 0 0 0 0 0 0 2 4 18 542
0 0 0 0 0 0 36 49 251 4254 0 1 1 0
0 0]
(5)模型预处理
from sklearn.preprocessing import StandardScaler#标准化处理
model = StandardScaler().fit(x_train)#转化器
x_train_ss = model.transform(x_train)#数据转换
x_test_ss = model.transform(x_test)
x_train_ss.max(axis=0)
array([ 3.75279933, 3.40078459, 3.87612308, 5.22160261, 3.47708666,
4.46981795, 4.23702302, 3.86941019, 4.32316651, 5.17184343,
8.08552814, 6.46636386, 8.20460134, 11.59481831, 7.99107585,
6.19064439, 10.06666234, 4.79952833, 6.94486675, 6.79949363,
4.04728935, 3.8607151 , 4.23197577, 5.78700996, 3.76910474,
4.91271971, 4.65125398, 2.63951827, 5.7811745 , 6.88724735])
(6)分类模型构建
#分类模型构建
from sklearn.svm import SVC
model = SVC().fit(x_train_ss, y_train)#SVC估计器,对模型进行训练
model.predict(x_test_ss)#用测试集来预测模型
array([1, 1, 1, 0, 1, 1, 1, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 0, 0, 0, 0,
1, 1, 0, 1, 1, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 0, 0, 0, 1,
0, 1, 1, 0, 0, 0, 0, 1, 1, 1, 1, 1, 0, 1, 1, 1, 1, 1, 1, 0, 1, 1,
1, 1, 1, 1, 0, 1, 0, 1, 1, 1, 0, 1, 1, 1, 0, 1, 0, 1, 0, 0, 1, 0,
1, 0, 1, 0, 1, 1, 0, 0, 0, 1, 0, 1, 0, 0, 0, 1, 0, 1, 0, 1, 0, 1,
0, 1, 1, 1])
model.score(x_test_ss,y_test)
0.9473684210526315
3.2、评价分类模型
3.2.1、分类模型的评价指标
- 分类模型对测试集进行预测而得出的准确率并不能很好地反映模型的性能,为了有效判断一个预测模型的性能表现,需要结合真实值,计算出精确率、召回率、F1值和Cohen’s Kappa系数等指标来衡量。常规分类模型的评价指标如表所示。分类模型评价方法前4种都是分值越高越好,其使用方法基本相同。
- sklearn的metrics模块还提供了一个能够输出分类模型评价报告的函数classfication_report。
| 方法名称 | 最佳值 | sklearn函数 |
|---|---|---|
| Precision(精确率) | 1.0 | metrics.precision_score |
| Recall(召回率) | 1.0 | metrics.recall_score |
| F1值 | 1.0 | metrics.f1_score |
| Cohen’s Kappa系数 | 1.0 | metrics.cohen_kappa_score |
| ROC曲线 | 最靠近y轴 | metrics. roc_curve |
from sklearn.metrics import recall_score, precision_score, f1_score, roc_curve
y_pre = model.predict(x_test_ss)#用测试集来预测模型
print ('召回率:',recall_score(y_test, y_pre)) #召回率
print ('准确率:',precision_score(y_test, y_pre))#准确率
print ('F1值:',f1_score(y_test, y_pre))#F1值
召回率: 0.9746835443037974
准确率: 1.0
F1值: 0.9871794871794872
3.2.2、ROC曲线
- 除了使用数值,表格形式评估分类模型的性能,还可通过绘制ROC曲线的方式来评估分类模型。
- ROC曲线横纵坐标范围为[0,1],通常情况下ROC曲线与X轴形成的面积越大,表示模型性能越好。但是当ROC曲线处于下图中蓝色虚线的位置,就表明了模型的计算结果基本都是随机得来的,在此种情况下模型起到的作用几乎为零。故在实际中ROC曲线离图中蓝色虚线越远表示模型效果越好。
fpr, tpr, thresholds = roc_curve(y_test, y_pre)#返回的值分别为假证率,真证率,预计值
import matplotlib.pyplot as plt
plt.plot(fpr, tpr)
plt.show()
4、构建并评价回归模型
4.1、回归分析方法
- 从19世纪初高斯提出最小二乘估计算起,回归分析的历史已有200多年。从经典的回归分析方法到近代的回归分析方法。
- 按照研究方法划分,回归分析研究的范围大致如图所示:

- 回归算法的实现步骤和分类算法基本相同,分为学习和预测2个步骤。学习是通过训练样本数据来拟合回归方程;预测则是利用学习过程中拟合出的回归方程,将测试数据放入方程中求出预测值。
4.2、常用的回归模型
| 回归模型名称 | 适用条件 | 算法描述 |
|---|---|---|
| 线性回归 | 因变量与自变量是线性关系 | 对一个或多个自变量和因变量之间的线性关系进行建模,可用最小二乘法求解模型系数 |
| 非线性回归 | 因变量与自变量之间不都是线性关系 | 对一个或多个自变量和因变量之间的非线性关系进行建模。如果非线性关系可以通过简单的函数变换转化成线性关系,用线性回归的思想求解;如果不能转化,用非线性最小二乘方法求解 |
| Logistic回归 | 因变量一般有1和0(是与否)两种取值 | 是广义线性回归模型的特例,利用Logistic函数将因变量的取值范围控制在0和1之间,表示取值为1的概率 |
| 岭回归 | 参与建模的自变量之间具有多重共线性 | 是一种改进最小二乘估计的方法 |
| 主成分回归 | 参与建模的自变量之间具有多重共线性 | 主成分回归是根据主成分分析的思想提出来的,是对最小二乘法的一种改进,它是参数估计的一种有偏估计。可以消除自变量之间的多重共线性 |
4.3、sklearn库常用回归算法函数
- sklearn内部提供了不少回归算法,常用的函数如下表所示:
| 模块名称 | 函数名称 | 算法名称 |
|---|---|---|
| linear_model | LinearRegression | 线性回归 |
| svm | SVC | 支持向量机 |
| neighbors | KNeighborsRegressor | 最近邻回归 |
| tree | DecisionTreeRegressor | 回归决策树 |
| ensemble | RandomForestClassifier | 随机森林分类 |
| ensemble | GradientBoostingClassifier | 梯度提升分类树 |
- 可以利用预测结果和真实结果画出折线图作对比,以便更直观看出线性回归模型效果。
(1)导入数据
from sklearn.datasets import load_boston
data = load_boston()
x = data['data']
y = data['target']
(2)用训练集拟合回归方差
from sklearn.model_selection import train_test_split
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2)
(3)用训练集建模
from sklearn.linear_model import LinearRegression
model = LinearRegression().fit(x_train, y_train)
(4)预测值
y_pre = model.predict(x_test)
(5)绘制折线图
import matplotlib.pyplot as plt
plt.figure(figsize=(10,6))
plt.plot(range(len(y_test)), y_test)
plt.plot(range(len(y_pre)), y_pre)
plt.legend(['real','predict'])
plt.show()
4.4、回归模型评价指标
- 回归模型的性能评估不同于分类模型,虽然都是对照真实值进行评估,但由于回归模型的预测结果和真实值都是连续的,所以不能够求取Precision、Recall和F1值等评价指标。回归模型拥有一套独立的评价指标。
- 平均绝对误差、均方误差和中值绝对误差的值越靠近0,模型性能越好。可解释方差值和R方值则越靠近1,模型性能越好。
| 方法名称 | 最优值 | sklearn函数 |
|---|---|---|
| 平均绝对误差 | 0.0 | metrics. mean_absolute_error |
| 均方误差 | 0.0 | metrics. mean_squared_error |
| 中值绝对误差 | 0.0 | metrics. median_absolute_error |
| 可解释方差值 | 1.0 | metrics. explained_variance_score |
| R方值 | 1.0 | metrics. r2_score |
from sklearn.metrics import mean_squared_error,r2_score#均方误差,R方值
print ('均方误差:',mean_squared_error(y_true=y_test, y_pred=y_pre))#越接近0越好
print ('R方差:',r2_score(y_true=y_test, y_pred=y_pre))
均方误差: 19.027472855809343
R方差: 0.7606396627204572