Step by step
3鉴定一系列感兴趣的基因。
典型的是,这些基因是对你的实验调节反应比较强烈的基因(也就是差异基因)。下面讲描述三种和这些基因相关的输入网络数据到cytoscape的方法:
A:querying相互作用数据库
B:通过文本挖掘计数建立关系网络
C:加载自己的网络数据(从text tile)
究竟选取哪一种方式基于那种是最适合你的案例的。想跟从下面步骤的话下载galFiltered.sif文件,继续步骤。这个文件中,最有效的网络的建立至少有250个interacitons。为了获取这样的一个网络,至少得有25个gene,也可以增加更多的基因和更多的关系获取最理想的size。
(A)从cPath获取蛋白相互作用数据
(i)cPath允许用户从很多数据库获取蛋白相互作用数据,包括intAct和MINT.通过选择File-new-network-construct network using cPath来激活cPath。
(ii)在cpath插件对话框的左上角输入框中输入一个或多个感兴趣的基因
关键步骤:对这个功能才开始熟悉的新用户或许希望一次只输入一个基因名字。而有经验的用户则希望输入一串基因,就像sample.input.genes.txt中的内容
(iii)在speciees下拉菜单选择species,默认是所有生物,对于样例sample.input.genes.txt则选择酿酒酵母
(iv)记录的最大数目默认值是10,把这个数值改为500.如果感兴趣的基因是一个的话,默认设置对你探寻需要的列表还可以,但如果你有很多个感兴趣的基因的话,就会获取更多的记录来建立连接。许多数据库记录里包含了一个蛋白的多个关系,因此所获取的连接会比限制的大。为了获取基因列表的所有相互作用,选择No limit,不过要知道,limit数越高,载入需要的时间越长。
(v)点击search button。稍等一会,cytoscape画布就会显示蛋白质(nodes)之间的相互作用网络,并且以grid形式排列,连接方式是获取的interacitons(edges)。默认情况下,cytoscape显示网络10,000或稍少的节点,而对于更大的网络,用户或许需要在network tree viewer的network lable上 右击,然后在出现的create view上查看
(vi)在network tree viewer上注意你的网络entry,并且要看下nodes和edges的数目
(B)产生关联网络
(i)在插件菜单下,选择Agilent literature search
关键步骤:这个插件第一次执行的时候,会让用户选择接受许可协议。接下来,Agilent literature search窗口会出现。
(ii)窗口左上部的terms panel里,输入你感兴趣的基因的最多100个基因或蛋白
关键步骤:为了达到最好的效果,搜索的时候最好使用HUGO gene symbol或其他合适的官方基因名称。这个搜索过程大概每个基因需要3s,为了符合pubmed使用限制,我们建议从一个短列表开始(10或更少的基因)。注意,查询编辑面板和基因列表相似,panel中的每一行代表将会被发往pubmed的one query。
(iii)在提取控制下,转到concept lexicon(概念词典) 下拉菜单,选择物种species。对于样本数据,选择saccharomyces cerevisiae。保留相互作用词典设置limited,句子选择的要提取文本的物种之间更容易发生相互作用,并且这是一个推荐设置,除非你的案例文献很少。
(iv)通过设置Max engine mathes可以提高每次查询获得的出版物的数量,fileld,up to 1,000查询may be issued in total,可以符合pubmed对用户的限制。对于样本例子,max engine matche 可以提高的50.总体来说,每个搜索条目需要更多的出版文献比更多的搜索条目效率更高。
(v)点击use aliase按钮。会看到query editor区域变化了
(vi)在contex区域设置可以精简搜索,这里可以添加额外的描述,比如组织或疾病类型,点击use context复选框。对于样本数据,可以输入galactose,如果描述文字大于一个单词,要用”。比如(”transcriptional regulation”)。注意,query editor区域会陪陪你contet中的term,用AND连接。这会限制pubmed搜寻范围,当然是和search terms 和context term 相关的。
(viii)检查query mathes面板的文章列表,右击链接选择delete match即可删除文件,同时network中的相应nodes和edges也会被移除,当然如果他们被其他文章也支持的话,这些nodes和edge就不会被移除了。
(ix)这个功能使得用户得以查看参考文献。具体是(i)用鼠标选择网络中的一个或多个节点和边,(ii)在select菜单,选择evidence from literature-show sentences from the literature。
这会显示一个有从文献来的带句子列表的窗口,当然都和nodes和edges有关,搜索条目会以黑体显示。如果别名匹配,那么正式名字会以方括号显示。如果用户选择了多个nodes或edges,这个列表会包含所有选择nodes和edges的句子。鼠标点击任何一个句子,都会在用户默认浏览器下有摘要。通过右击delete可以删除句子。如果所有的支持一个edge的句子都被删除,这个edge也会从网络中被删除。
(x)用户可以通过右击node来扩充网络。右击 select-evidence from litreatrue-extended network from the literature。这会有一个新的node。如果新搜索产生任何新nodes,edges或句子,就会被假如已经存在的网络中。对nodes来说,如果这个node没有包含在list中,这是非常有效的
(c)从text file输入网络数据
(i)按样本sif文件,组织你的数据
(ii)file-import-network
4探索网络并展示
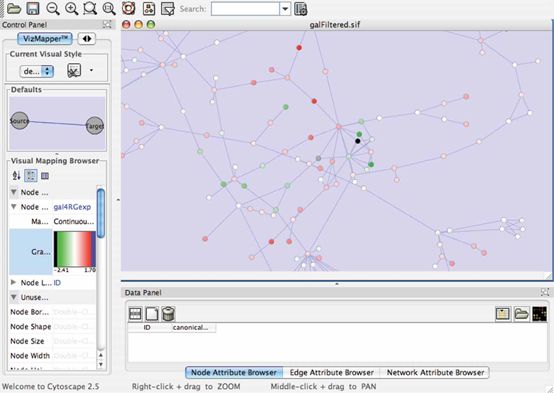
通过layout才看可以重新组织网络,yfles-organic。这种算法执行的算法是force-directed paradigm,其中nodes被当作相互排斥的力,edges在他们连接的nodes之间产生吸引的力。Nodes被排放的原则是使得这些力的总和最小。这种layout会揭露网络中的内含结构。具体来说,它有助于紧密连接点的识别,这往往意味着功能模块,还有揭示hub nodes,也就是被参与了很多相互关系,往往代表功能非常重要的蛋白。注意,cytoscape提供了非常多的layout方式,包括登记,环形,基于属性的输出。可以多体验。
5布局设置
为了让网络画布产生更大的空间,可以让control面板和data面板漂浮,方式是点击float window control图标,它在每个面板的右下部。这会把panel玻璃成单独的窗口。通过拖曳network 画布的边缘可以进行缩放。
6.鼠标左键可以选择任何node。
注意,选择的node会变成黄色。摁住鼠标左键不放进行唾液可以定义一个矩形区域进行选择。
7.注意,data panel列表了所有选择的nodes的“身份”。
这也会显示node属性。从cpath获得的nodes有一下属性,‘FULL-NAME’,描述性蛋白名字。从Agilent literature search插件来的nodes有一下属性比如’Nbrconnections‘,node的度。
8.点击select attributes 按钮
可以选择属性中的一个展示,它在data panel 工具栏的最左边。这会展示可获得的属性的列表,并且选择的属性会高亮显示。选择或去选择一个属性,都是通过点击name。鼠标右击退出
9.可以拖曳矩形区域来选择一个或多个edges。
从cpath获得的edges有以下属性,包括‘EXPERIMENTAL-SYSTEM-NAME’,支持这个关系的实验证据。从Agilent Literature Search 插件获得的Edges有包括Nbrsentences的属性,支持这个关系的不同的句子的数目。
10.data panel 同样可以看edge属性。和第8步差不多
11.工具栏的zoom in 按钮
可以放大网络,符号是放大镜符号+。注意到control panel,网络总览中的蓝色的方框会收缩面会网络的部分。移动这个框可以寻找整个网络。其他控制有zoom out(缩小)。
12确定网络中的node的ID name,
比如在galFiltered.sif文件中的YPL248C。具体是,在画布右上方有个搜索按钮,输入名字,当都输入的时候一个下来菜单会显示匹配node ID的列表。这里可以搜索任何属性(不仅仅是ID)通过点击“configure search options)按钮
13.cytoscape储存用户数据为session file,
包含所有网络,layout,属性数据等。通过save current session as来进行储存。一旦被储存,就可以在file下被加载。
14.注释属性和表达数据
可选择的步骤:额外的node和edge属性文件可以被整合进你的网络。(box3)
15表达数据文件有一定的格式。
具体看galExpData.pvals,这种格式文件可以用text编辑器查看详细内容。如果表达文件的第一列与cytoscape网络中nodes精确匹配(case-sensitive大小写之分),那就直接看下面的16.)15适合样本文件。否则,依然遵从box3也就是14.下面详细说下这种属性文件的建立方法
为了输入属性文件和表达数据文件到cytoscape,基因或蛋白的标识符必须和cytoscape中的nodeID(或其他cytoscape属性已经被加载的)精确匹配。如果没有匹配标识,可以通过加载另外的标识文件作为新的node属性。现在介绍一种方法来建立和加载标识匹配属性。我们将使用一个教Synergizer的外部ID匹配服务,这是有哈佛大学Roth实验室提供的。也可以见http://baderlab.org/IdentifierMapping/
1)在cytoscape画布,选择所有节点,快捷键是CTRL-A
2)在node属性浏览窗口,选择所有nodes的ID column内容,有个快捷键是选中第一个nodeID,然后摁CTRL-SHIFT-END
3)转到synergize identifier翻译服务http://baderlab.org/IdentifierMapping/
4)转到box(IDS to translate)粘贴标识列表
5)选择物种,Set Select FROM namespace: to
ANY. In the field labeled Select TO namespace:, select the
identifier used in your expression data file. For the sample data,
select systematic. Click the box labeled Output as Spreadsheet: and
click Submit.
6.)Theidentifiermappingwillbedownloadedtoyourcomputerinan
Excel file named ‘translate’. Open the file with a spreadsheet viewer
such as Excel. The first column contains the original identifier list.
The second columncontains the identifiers that these mapto inthe
chosen namespace. Note the label given to this column in the first
line.
7). Save this file in the comma-separated (CSV) format.
m CRITICAL STEP There are system-dependent issues in producing
and reading EXCEL files. This step guards against such issues.
8). Import these data into Cytoscape.
a.GototheFilemenu,thenImportandthenAttributefromTable
(Text/MS Excel). This will bring up the Import Annotation File
window.
b. Click the Select File button and specify your ‘translated’ CSV
file.
c. Select Show Text File Import Options and select Transfer first
line as attribute names.
d. Click on the Import button.
e. Verify that the new attribute is loaded byre turning to the Node
Attribute Browser and click on the Select Attribute button.
16加载表达数据。
File-table
17 检查下右下角data panel面板node attributes列表。
每个实验应该有两个特征,分别含有“xexp”和“xsig”,其中x代表实验水平的名字。表达数据是xexp,如果表达数据有p-values,那么有xsig的那列就是。否则,xsig会是空白。如果使用样本文件,会看到gal1RG,gal4RG,gal80R,表达FC值分别是,gal1RGexp,gal4RGexp,gal80Rexp..通过选择一个或几个实验的属性,可以在画布显示不一样。
设置可视化的node 特征
18.左边面板控制节点和边的可视化特征。
Network右边有个style,点开,node和edge和network属性选项卡,位于control panel的底部。
使用红色到绿色渐变改变nodes颜色
19 颜色
想根据FC值来定义node颜色的第一步是定位在“node color property”,初始是灰色,双击它
20.节点属性
node颜色挨着的是,是please select an attribute,点击这个信息,会产生一个节点属性列表。滚动滚轮到你的expression data value,例如例子中就是gal4Rexp
21.mapping type
node颜色下,是mapping type 入口,点击右列,就在表达数据下面,有个下拉菜单,选择continuous mapping
22.标签可视化
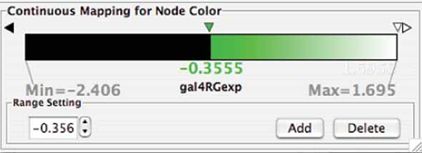
mapping type下面,可以看到标签可视化,graphical view,就是个空矩形。这会有一个窗口对node颜色进行梯度编辑,这会根据你的表达数据属性一直,两端显示最小和最大值。
23.add button
在这个节点颜色的梯度编辑器里,点击add button2次。会看到表达数据的属性分成两半,低的黑色,高的白色。
24.
双击白色的中间的下三角,会出现颜色选择窗口,选择绿色。现在看到range bar分为了两半,左边黑色,右边是梯度颜色,绿色到白色
25
滑动绿色的三角往左边去,留下一个端的黑色部分,像下图d。在cytoscape画布上,你会单独一些节点成黑色了(表达值低的),大多数节点都是黑色或淡绿色有一些是白色(表达值高的)
26
点击右端的下三角,也会产生一个颜色选择窗口,选择红色。这回产生一个梯度,是白色到红色的,看下图f。在画布上你会发现有些表达值高的节点现在是粉色或红色。更深的颜色代表更高的表达值。
28滑动红色的三角往左边挪动一小段距离,双击白色右三角(在右端),选择蓝色,见g
29
在cytoscape画布上,你会发现,节点颜色红色到绿色分布
(i)低表达值,绿色。高表达值红色,中间的白色
(ii)相对淡的颜色接近中间的那些,黑色表示range的末端
(iii)你会发现当你设置颜色的时候,画布颜色会即时改变
(iv)关闭梯度编辑窗口。
Figure 4 | The steps in creating a green-to-red node color gradient. (a) Specify a new attribute-based gradient; (b) add two range selector end points;
(c) change the color at the lower end point to green; (d) move this end point to the bottom of the range; (e) add another new range selector end point;
(f) change the color at the top of the range to red; (g) change the color at the top of the range to blue; and (h) a network with the nodes rendered with a
red–green color map
30
默认情况下,节点是粉色。相应的粉色的节点颜色暗含着表达值比range中间的值稍微高点。也可能意味着这个节点无表达。为了移除这种不确定性,改变默认节点颜色为灰色,步骤是
(i)还是在刚才的control panel,定位default panel,显示默认节点和边渲染
(ii)点击这个面板里的node,会显示一个默认node属性设置
(iii)在窗口中,点击node fill color按钮,选择灰色,click apply。