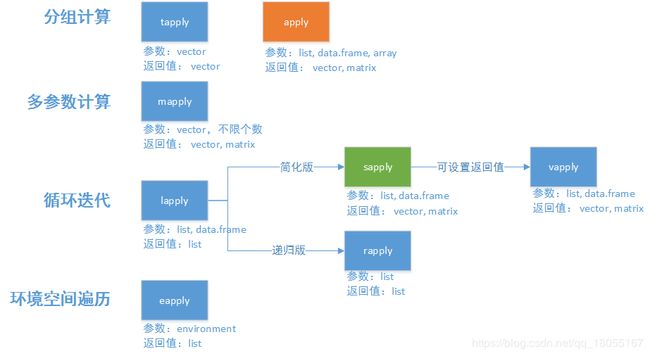
R语言中apply函数族的介绍
文章目录
- 综括
- ==apply函数==
- 利用apply函数实现
- 利用循环实现
- 定义一个结果的数据框
- 定义for循环
- 利用R的特性
- 三种方法消耗时间比较
- ==lapply函数==
- ==sapply函数==
- vapply函数
- ==mapply函数==
- ==tapply函数==
- rapply函数
- eapply函数
综括
apply函数
apply函数是最常用的代替for循环的函数。apply函数可以对矩阵、数据框、数组(二维、多维),按行或列进行循环计算,对子元素进行迭代,并把子元素以参数传递的形式给自定义的FUN函数中,并以返回计算结果。
函数定义:
apply(X, MARGIN, FUN, ...)
参数列表:
- X:数组、矩阵、数据框
- MARGIN: 按行计算或按按列计算,1表示按行,2表示按列
- FUN: 自定义的调用函数
- …: 更多参数,可选
比如,对一个矩阵的每一行求和,下面就要用到apply做循环了。
> x<-matrix(1:12,ncol=3)
> apply(x,1,sum)
[1] 15 18 21 24
下面计算一个稍微复杂点的例子,按行循环,让数据框的x1列加1,并计算出x1,x2列的均值。
利用apply函数实现
#生成data.frame
> x <- cbind(x1 = 3, x2 = c(4:1, 2:5)); x
x1 x2
[1,] 3 4
[2,] 3 3
[3,] 3 2
[4,] 3 1
[5,] 3 2
[6,] 3 3
[7,] 3 4
[8,] 3 5
#自定义函数myFUN,第一个参数x为数据
#第二、三个参数为自定义参数,可以通过apply的'...'进行传入。
myFUN<- function(x, c1, c2) {
c(sum(x[c1],1), mean(x[c2]))
}
#把数据框按行做循环,每行分别传递给myFUN函数,设置c1,c2对应myFUN的第二、三个参数
> apply(x,1,myFUN,c1='x1',c2=c('x1','x2'))
[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8]
[1,] 4.0 4.0 4.0 4 4.0 4 4.0 4
[2,] 3.5 3 2.5 2 2.5 3 3.5 4
通过这个上面的自定义函数myFUN就实现了,一个常用的循环计算。
利用循环实现
如果直接用for循环来实现,那么代码如下:
定义一个结果的数据框
df<-data.frame()
定义for循环
> for(i in 1:nrow(x)){
+ row<-x[i,] # 每行的值
+ df<-rbind(df,rbind(c(sum(row[1],1), mean(row)))) # 计算,并赋值到结果数据框
+ }
# 打印结果数据框
> df
V1 V2
1 4 3.5
2 4 3.0
3 4 2.5
4 4 2.0
5 4 2.5
6 4 3.0
7 4 3.5
8 4 4.0
通过for循环的方式,也可以很容易的实现上面计算过程,但是这里还有一些额外的操作需要自己处理,比如构建循环体、定义结果数据集、并合每次循环的结果到结果数据集。
利用R的特性
对于上面的需求,还有第三种实现方法,那就是完成利用了R的特性,通过向量化计算来完成的。
> data.frame(x1=x[,1]+1,x2=rowMeans(x))
x1 x2
1 4 3.5
2 4 3.0
3 4 2.5
4 4 2.0
5 4 2.5
6 4 3.0
7 4 3.5
8 4 4.0
那么,一行就可以完成整个计算过程了。
三种方法消耗时间比较
接下来,我们需要再比较一下3种操作上面性能上的消耗。
# 清空环境变量
> rm(list=ls())
# 封装fun1
> fun1<-function(x){
+ myFUN<- function(x, c1, c2) {
+ c(sum(x[c1],1), mean(x[c2]))
+ }
+ apply(x,1,myFUN,c1='x1',c2=c('x1','x2'))
+ }
# 封装fun2
> fun2<-function(x){
+ df<-data.frame()
+ for(i in 1:nrow(x)){
+ row<-x[i,]
+ df<-rbind(df,rbind(c(sum(row[1],1), mean(row))))
+ }
+ }
# 封装fun3
> fun3<-function(x){
+ data.frame(x1=x[,1]+1,x2=rowMeans(x))
+ }
# 生成数据集
> x <- cbind(x1=3, x2 = c(400:1, 2:500))
# 分别统计3种方法的CPU耗时。
> system.time(fun1(x))
用户 系统 流逝
0.01 0.00 0.02
> system.time(fun2(x))
用户 系统 流逝
0.19 0.00 0.18
> system.time(fun3(x))
用户 系统 流逝
0 0 0
从CPU的耗时来看,用for循环实现的计算是耗时最长的,apply实现的循环耗时很短,而直接使用R语言内置的向量计算的操作几乎不耗时间。通过上面的测试,对同一个计算来说,优先考虑R语言内置的向量计算,必须要用到循环时则使用apply函数,应该尽量避免显示的使用for,while等操作方法。
lapply函数
lapply函数是一个最基础循环操作函数之一,用来对list、data.frame数据集进行循环,并返回和X长度同样的list结构作为结果集,通过lapply的开头的第一个字母’l’就可以判断返回结果集的类型。
函数定义:
lapply(X, FUN, ...)
参数列表:
- X:list、data.frame数据
- FUN: 自定义的调用函数
- …: 更多参数,可选
比如,计算list中的每个KEY对应该的数据的分位数。
# 构建一个list数据集x,分别包括a,b,c 三个KEY值。
> x <- list(a = 1:10, b = rnorm(6,10,5), c = c(TRUE,FALSE,FALSE,TRUE));x
$a
[1] 1 2 3 4 5 6 7 8 9 10
$b
[1] 0.7585424 14.3662366 13.3772979 11.6658990 9.7011387 21.5321427
$c
[1] TRUE FALSE FALSE TRUE
# 分别计算每个KEY对应该的数据的分位数。
> lapply(x,fivenum)
$a
[1] 1.0 3.0 5.5 8.0 10.0
$b
[1] 0.7585424 9.7011387 12.5215985 14.3662366 21.5321427
$c
[1] 0.0 0.0 0.5 1.0 1.0
lapply就可以很方便地把list数据集进行循环操作了,还可以用data.frame数据集按列进行循环,但如果传入的数据集是一个向量或矩阵对象,那么直接使用lapply就不能达到想要的效果了。
比如,对矩阵的列求和。
# 生成一个矩阵
> x <- cbind(x1=3, x2=c(2:1,4:5))
> x; class(x)
x1 x2
[1,] 3 2
[2,] 3 1
[3,] 3 4
[4,] 3 5
[1] "matrix"
# 求和
> lapply(x, sum)
[[1]]
[1] 3
[[2]]
[1] 3
[[3]]
[1] 3
[[4]]
[1] 3
[[5]]
[1] 2
[[6]]
[1] 1
[[7]]
[1] 4
[[8]]
[1] 5
sapply函数
sapply函数是一个简化版的lapply,sapply增加了2个参数simplify和USE.NAMES,主要就是让输出看起来更友好,返回值为向量,而不是list对象。
函数定义:
sapply(X, FUN, ..., simplify=TRUE, USE.NAMES = TRUE)
参数列表:
- X:数组、矩阵、数据框
- FUN: 自定义的调用函数
- …: 更多参数,可选 simplify: 是否数组化,当值array时,输出结果按数组进行分组
- USE.NAMES: 如果X为字符串,TRUE设置字符串为数据名,FALSE不设置
我们还用上面lapply的计算需求进行说明。
> x <- cbind(x1=3, x2=c(2:1,4:5))
# 对矩阵计算,计算过程同lapply函数
> sapply(x, sum)
[1] 3 3 3 3 2 1 4 5
# 对数据框计算
> sapply(data.frame(x), sum)
x1 x2
12 12
# 检查结果类型,sapply返回类型为向量,而lapply的返回类型为list
> class(lapply(x, sum))
[1] "list"
> class(sapply(x, sum))
[1] "numeric"
如果simplify=FALSE和USE.NAMES=FALSE,那么完全sapply函数就等于lapply函数了。
> lapply(data.frame(x), sum)
$x1
[1] 12
$x2
[1] 12
> sapply(data.frame(x), sum, simplify=FALSE, USE.NAMES=FALSE)
$x1
[1] 12
$x2
[1] 12
对于simplify为array时,我们可以参考下面的例子,构建一个三维数组,其中二个维度为方阵。
详细内容可以参考:
http://blog.fens.me/r-apply/
> a<-1:2
# 按数组分组
> sapply(a,function(x) matrix(x,2,2), simplify='array')
, , 1
[,1] [,2]
[1,] 1 1
[2,] 1 1
, , 2
[,1] [,2]
[1,] 2 2
[2,] 2 2
# 默认情况,则自动合并分组
> sapply(a,function(x) matrix(x,2,2))
[,1] [,2]
[1,] 1 2
[2,] 1 2
[3,] 1 2
[4,] 1 2
对于字符串的向量,还可以自动生成数据名。
> val<-head(letters)
# 默认设置数据名
> sapply(val,paste,USE.NAMES=TRUE)
a b c d e f
"a" "b" "c" "d" "e" "f"
# USE.NAMES=FALSE,则不设置数据名
> sapply(val,paste,USE.NAMES=FALSE)
[1] "a" "b" "c" "d" "e" "f"
vapply函数
vapply类似于sapply,提供了FUN.VALUE参数,用来控制返回值的行名,这样可以让程序更健壮。
函数定义:
vapply(X, FUN, FUN.VALUE, ..., USE.NAMES = TRUE)
参数列表:
- X:数组、矩阵、数据框
- FUN: 自定义的调用函数
- FUN.VALUE: 定义返回值的行名row.names
- …: 更多参数,可选
- USE.NAMES: 如果X为字符串,TRUE设置字符串为数据名,FALSE不设置
- 比如,对数据框的数据进行累计求和,并对每一行设置行名row.names
# 生成数据集
> x <- data.frame(cbind(x1=3, x2=c(2:1,4:5)))
# 设置行名,4行分别为a,b,c,d
> vapply(x,cumsum,FUN.VALUE=c('a'=0,'b'=0,'c'=0,'d'=0))
x1 x2
a 3 2
b 6 3
c 9 7
d 12 12
# 当不设置时,为默认的索引值
> a<-sapply(x,cumsum);a
x1 x2
[1,] 3 2
[2,] 6 3
[3,] 9 7
[4,] 12 12
# 手动的方式设置行名
> row.names(a)<-c('a','b','c','d')
> a
x1 x2
a 3 2
b 6 3
c 9 7
d 12 12
通过使用vapply可以直接设置返回值的行名,这样子做其实可以节省一行的代码,让代码看起来更顺畅,当然如果不愿意多记一个函数,那么也可以直接忽略它,只用sapply就够了。
mapply函数
mapply也是sapply的变形函数,类似多变量的sapply,但是参数定义有些变化。第一参数为自定义的FUN函数,第二个参数’…’可以接收多个数据,作为FUN函数的参数调用。
函数定义:
mapply(FUN, ..., MoreArgs = NULL, SIMPLIFY = TRUE,USE.NAMES = TRUE)
参数列表:
- FUN: 自定义的调用函数
- …: 接收多个数据
- MoreArgs: 参数列表
- SIMPLIFY: 是否数组化,当值array时,输出结果按数组进行分组
- USE.NAMES: 如果X为字符串,TRUE设置字符串为数据名,FALSE不设置
比如,比较3个向量大小,按索引顺序取较大的值。
> set.seed(1)
# 定义3个向量
> x<-1:10
> y<-5:-4
> z<-round(runif(10,-5,5))
# 按索引顺序取较大的值。
> mapply(max,x,y,z)
[1] 5 4 3 4 5 6 7 8 9 10
再看一个例子,生成4个符合正态分布的数据集,分别对应的均值和方差为c(1,10,100,1000)。
> set.seed(1)
# 长度为4
> n<-rep(4,4)
# m为均值,v为方差
> m<-v<-c(1,10,100,1000)
# 生成4组数据,按列分组
> mapply(rnorm,n,m,v)
[,1] [,2] [,3] [,4]
[1,] 0.3735462 13.295078 157.57814 378.7594
[2,] 1.1836433 1.795316 69.46116 -1214.6999
[3,] 0.1643714 14.874291 251.17812 2124.9309
[4,] 2.5952808 17.383247 138.98432 955.0664
由于mapply是可以接收多个参数的,所以我们在做数据操作的时候,就不需要把数据先合并为data.frame了,直接一次操作就能计算出结果了。
tapply函数
tapply用于分组的循环计算,通过INDEX参数可以把数据集X进行分组,相当于group by的操作。
函数定义:
tapply(X, INDEX, FUN = NULL, ..., simplify = TRUE)
参数列表:
- X: 向量
- INDEX: 用于分组的索引
- FUN: 自定义的调用函数
- …: 接收多个数据
- simplify : 是否数组化,当值array时,输出结果按数组进行分组
比如,计算不同品种的鸢尾花的花瓣(iris)长度的均值。
# 通过iris$Species品种进行分组
> tapply(iris$Petal.Length,iris$Species,mean)
setosa versicolor virginica
1.462 4.260 5.552
对向量x和y进行计算,并以向量t为索引进行分组,求和。
> set.seed(1)
# 定义x,y向量
> x<-y<-1:10;x;y
[1] 1 2 3 4 5 6 7 8 9 10
[1] 1 2 3 4 5 6 7 8 9 10
# 设置分组索引t
> t<-round(runif(10,1,100)%%2);t
[1] 1 2 2 1 1 2 1 0 1 1
# 对x进行分组求和
> tapply(x,t,sum)
0 1 2
8 36 11
由于tapply只接收一个向量参考,通过’…’可以把再传给你FUN其他的参数,那么我们想去y向量也进行求和,把y作为tapply的第4个参数进行计算。
> tapply(x,t,sum,y)
0 1 2
63 91 66
得到的结果并不符合我们的预期,结果不是把x和y对应的t分组后求和,而是得到了其他的结果。第4个参数y传入sum时,并不是按照循环一个一个传进去的,而是每次传了完整的向量数据,那么再执行sum时sum(y)=55,所以对于t=0时,x=8 再加上y=55,最后计算结果为63。那么,我们在使用’…’去传入其他的参数的时候,一定要看清楚传递过程的描述,才不会出现的算法上的错误。
rapply函数
rapply是一个递归版本的lapply,它只处理list类型数据,对list的每个元素进行递归遍历,如果list包括子元素则继续遍历。
函数定义:
rapply(object, f, classes = "ANY", deflt = NULL, how = c("unlist", "replace", "list"), ...)
参数列表:
- object:list数据
- f: 自定义的调用函数
- classes : 匹配类型, ANY为所有类型
- deflt: 非匹配类型的默认值
- how: 3种操作方式,当为replace时,则用调用f后的结果替换原list中原来的元素;当为list时,新建一个list,类型匹配调用f函数,不匹配赋值为deflt;当为unlist时,会执行一次unlist(recursive = TRUE)的操作
- …: 更多参数,可选
比如,对一个list的数据进行过滤,把所有数字型numeric的数据进行从小到大的排序。
> x=list(a=12,b=1:4,c=c('b','a'))
> y=pi
> z=data.frame(a=rnorm(10),b=1:10)
> a <- list(x=x,y=y,z=z)
# 进行排序,并替换原list的值
> rapply(a,sort, classes='numeric',how='replace')
$x
$x$a
[1] 12
$x$b
[1] 4 3 2 1
$x$c
[1] "b" "a"
$y
[1] 3.141593
$z
$z$a
[1] -0.8356286 -0.8204684 -0.6264538 -0.3053884 0.1836433 0.3295078
[7] 0.4874291 0.5757814 0.7383247 1.5952808
$z$b
[1] 10 9 8 7 6 5 4 3 2 1
> class(a$z$b)
[1] "integer"
从结果发现,只有 z z za的数据进行了排序,检查 z z zb的类型,发现是integer,是不等于numeric的,所以没有进行排序。
接下来,对字符串类型的数据进行操作,把所有的字符串型加一个字符串’++++’,非字符串类型数据设置为NA。
> rapply(a,function(x) paste(x,'++++'),classes="character",deflt=NA, how = "list")
$x
$x$a
[1] NA
$x$b
[1] NA
$x$c
[1] "b ++++" "a ++++"
$y
[1] NA
$z
$z$a
[1] NA
$z$b
[1] NA
只有 x x xc为字符串向量,都合并了一个新字符串。那么,有了rapply就可以对list类型的数据进行方便的数据过滤了。
eapply函数
对一个环境空间中的所有变量进行遍历。如果我们有好的习惯,把自定义的变量都按一定的规则存储到自定义的环境空间中,那么这个函数将会让你的操作变得非常方便。当然,可能很多人都不熟悉空间的操作,那么请参考文章 揭开R语言中环境空间的神秘面纱,解密R语言函数的环境空间。
函数定义:
eapply(env, FUN, ..., all.names = FALSE, USE.NAMES = TRUE)
参数列表:
- env: 环境空间
- FUN: 自定义的调用函数
- …: 更多参数,可选 all.names: 匹配类型, ANY为所有类型
- USE.NAMES: 如果X为字符串,TRUE设置字符串为数据名,FALSE不设置
下面我们定义一个环境空间,然后对环境空间的变量进行循环处理。
# 定义一个环境空间
> env
# 向这个环境空间中存入3个变量
> env$a <- 1:10
> env$beta <- exp(-3:3)
> env$logic <- c(TRUE, FALSE, FALSE, TRUE)
> env
# 查看env空间中的变量
> ls(env)
[1] "a" "beta" "logic"
# 查看env空间中的变量字符串结构
> ls.str(env)
a : int [1:10] 1 2 3 4 5 6 7 8 9 10
beta : num [1:7] 0.0498 0.1353 0.3679 1 2.7183 ...
logic : logi [1:4] TRUE FALSE FALSE TRUE
计算env环境空间中所有变量的均值。
> eapply(env, mean)
$logic
[1] 0.5
$beta
[1] 4.535125
$a
[1] 5.5
再计算中当前环境空间中的所有变量的占用内存大小。
# 查看当前环境空间中的变量
> ls()
[1] "a" "df" "env" "x" "y" "z" "X"
# 查看所有变量的占用内存大小
> eapply(environment(), object.size)
$a
2056 bytes
$df
1576 bytes
$x
656 bytes
$y
48 bytes
$z
952 bytes
$X
1088 bytes
$env
56 bytes
eapply函数平时很难被用到,但对于R包开发来说,环境空间的使用是必须要掌握的。特别是当R要做为工业化的工具时,对变量的精确控制和管理是非常必要的。