【机器学习系列之七】模型调优与模型融合(代码应用篇)
这是本人对模型的融合的代码合集,环境是python3,只要复制过去就可以用了,非常方便。
- 目录
- 1.交叉验证
1.1 原理
1.2 GridSearchCV - 2.绘制学习曲线
- 3.stacking
3.1 stacking原理
3.2 代码实现不同版本的stacking
3.2.1.官网给的例子(简单粗暴)
3.2.2 用概率作为第二层模型的特征
3.2.3 特征多样性
3.2.4.参数详解
1.交叉验证
1.1 原理
基本思想就是把训练数据分成几份,分别为训练集和测试集,用训练集和验证集作为模型选择。最典型的是k折交叉验证: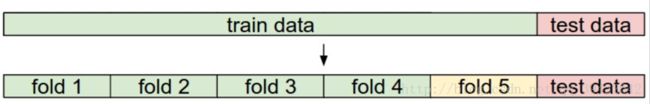
K折交叉验证,初始采样分割成K个子样本,一个单独的子样本被保留作为验证模型的数据,其他K-1个样本用来训练。交叉验证重复K次,每个子样本验证一次,平均K次的结果或者使用其它结合方式,最终得到一个单一估测。这个方法的优势在于,同时重复运用随机产生的子样本进行训练和验证,每次的结果验证一次,10次交叉验证是最常用的。
如图是5折,因此可以做五次验证,然后对结果进行平均。
1.2 GridSearchCV
一般使用GridSearchCV来做格点搜索寻找最优参数,代码如下:
import numpy as np
import pandas as pd
from pandas import DataFrame
from patsy import dmatrices
import string
from operator import itemgetter
import json
from sklearn.ensemble import RandomForestClassifier,GradientBoostingClassifier
from sklearn.cross_validation import cross_val_score
from sklearn.pipeline import Pipeline
from sklearn.grid_search import GridSearchCV
from sklearn.cross_validation import train_test_split,StratifiedShuffleSplit,StratifiedKFold
from sklearn import preprocessing
from sklearn.metrics import classification_report
from sklearn.externals import joblib
seed=0
clf=GradientBoostingClassifier(n_estimators=500)
###grid search找到最好的参数
param_grid = dict( )
##创建分类pipeline
pipeline=Pipeline([ ('clf',clf) ])
grid_search = GridSearchCV(pipeline, param_grid=param_grid, verbose=3,scoring='accuracy',\
cv=StratifiedShuffleSplit(y_train, n_iter=10, test_size=0.2,random_state=seed)).fit(x_train, y_train)
# 对结果打分
print("Best score: %0.3f" % grid_search.best_score_)
print(grid_search.best_estimator_)
print('-----grid search end------------')
print ('on all train set')
scores = cross_val_score(grid_search.best_estimator_, x_train, y_train,cv=3,scoring='accuracy')
print (scores.mean(),scores)
print ('on test set')
scores = cross_val_score(grid_search.best_estimator_, x_test, y_test,cv=3,scoring='accuracy')
print(scores.mean(),scores)
2.绘制学习曲线
要是模型太复杂,导致过拟合,对测试集的预测就不会太好,该怎么判断模型是否过拟合呢?
对的,就是学习曲线
def plot_learning_curve(estimator, title, X, y, ylim=None, cv=None,
n_jobs=1, train_sizes=np.linspace(.1, 1.0, 5)):
plt.figure()
plt.title(title)
if ylim is not None:
plt.ylim(*ylim)
plt.xlabel("Training examples")
plt.ylabel("Score")
train_sizes, train_scores, test_scores = learning_curve(
estimator, X, y, cv=cv, n_jobs=n_jobs, train_sizes=train_sizes)
train_scores_mean = np.mean(train_scores, axis=1)
train_scores_std = np.std(train_scores, axis=1)
test_scores_mean = np.mean(test_scores, axis=1)
test_scores_std = np.std(test_scores, axis=1)
plt.grid()
plt.fill_between(train_sizes, train_scores_mean - train_scores_std,
train_scores_mean + train_scores_std, alpha=0.1,
color="r")
plt.fill_between(train_sizes, test_scores_mean - test_scores_std,
test_scores_mean + test_scores_std, alpha=0.1, color="g")
plt.plot(train_sizes, train_scores_mean, 'o-', color="r",
label="Training score")
plt.plot(train_sizes, test_scores_mean, 'o-', color="g",
label="Cross-validation score")
plt.legend(loc="best")
return plt
title = "Learning Curves (Random Forest, n_estimators = 100)"
cv = cross_validation.ShuffleSplit(df_train_data.shape[0], n_iter=10,test_size=0.2, random_state=0)
estimator = RandomForestRegressor(n_estimators = 100)
plot_learning_curve(estimator, title, X, y, (0.0, 1.01), cv=cv, n_jobs=4)
plt.show()
一看就是欠拟合了,因为数据,特征都太少,模型学习的非常容易,所以在训练集上做的非常好,但是在测试集上做的特别差。其实主要看他们的差距,差距特别大,多半是不正常。
发现他过拟合之后该怎么办了呢?
对于过拟合:
1.找更多的数据来学习
2.增大正则化系数
3. 减少特征个数
对于欠拟合:
1. 找更多的特征
2. 减少正则化系数
权重分析
对于线性的模型,比如逻辑回归,线性回归,或者线性的SVM,可以把权重绝对值高/低的特征拿出来,做特征组合,或者特征细分。
3.stacking
3.1 stacking原理
原理也很简单,就是把很多基分类器,比如逻辑回归,随机森林,GBDT等分类器,预测的结果,作为特征,再来用分类器预测一波最终的结果。
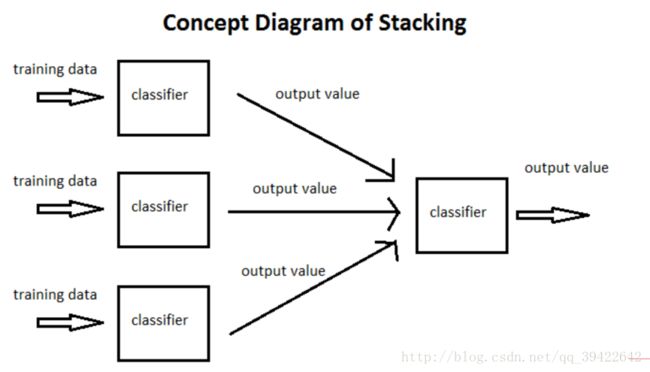
说详细点吧。
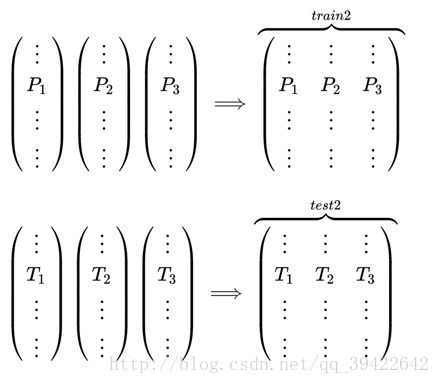
假设现在有三个分类器,分别是M1,M2,M3.
1.用M1对训练集训练,然后预测训练集和测试集(看清楚哦)的标签列。分别是 P1,T1
对M2,M3重复上述操作,得到 P2,T2,P3,T3
但是这样会造成过拟合,因为我们用训练集训练,然后又去预测了训练集,所以很多时候stacking的效果也不见得会很好,这个需要根据具体情况来看。
一般优化的思路是用交叉验证:
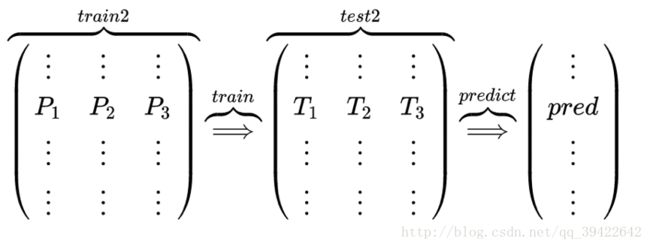
以2折交叉验证为例:假设数据集为D1,D2,先用D1训练模型,然后预测D2,得到 P12 。然后再用D2 训练模型,预测D1,得到 P11 ,将 P11,P12 组合起来,就得到了我们先前说的P1。
对于T1,有两种方法可以得到:
A.对D1训练的时候,预测了D2,同时也可以对整个测试集进行预测得到 T11 ,训练D2之后,也可以对整个测试集进行预测得到 T12 ,对 T11,T12 求平均,得到 T1 .
B. 第二种就是直接把 T11 当成 T1 使用。

该图是五折交叉验证,得到P1,T1的过程,通过对训练集的五次预测,组合得到P1的过程,这个得到的只是一个第二层的输入中的一个特征。
3.2 代码实现不同版本的stacking
重要的还是看一下怎么使用,在python中专门集成了用于stacking的库mlxtend,它能很快的完成模型的stacking。
这个的使用也有几种方法:
3.2.1.官网给的例子
import numpy as np
import matplotlib.pyplot as plt
import matplotlib.gridspec as gridspec
import itertools
from sklearn.linear_model import LogisticRegression
from sklearn.svm import SVC
from sklearn.ensemble import RandomForestClassifier
from mlxtend.classifier import EnsembleVoteClassifier
from mlxtend.data import iris_data
from mlxtend.plotting import plot_decision_regions
# Initializing Classifiers
clf1 = LogisticRegression(random_state=0)
clf2 = RandomForestClassifier(random_state=0)
clf3 = SVC(random_state=0, probability=True)
eclf = EnsembleVoteClassifier(clfs=[clf1, clf2, clf3], weights=[2, 1, 1], voting='soft')
# Loading some example data
X, y = iris_data()
X = X[:,[0, 2]]
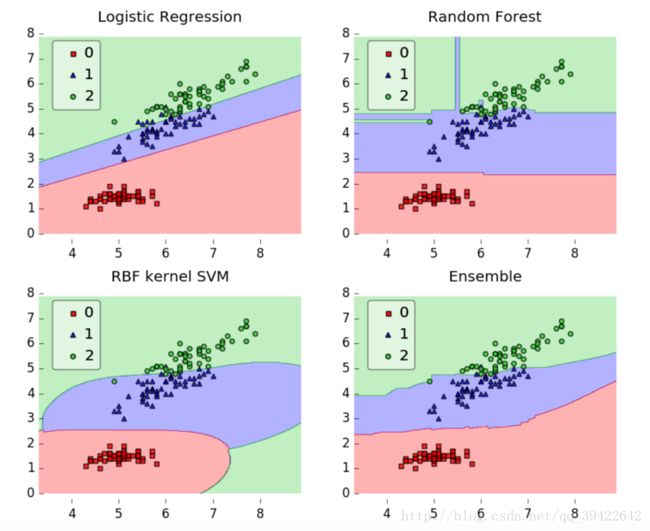
# Plotting Decision Regions
gs = gridspec.GridSpec(2, 2)
fig = plt.figure(figsize=(10, 8))
for clf, lab, grd in zip([clf1, clf2, clf3, eclf],
['Logistic Regression', 'Random Forest', 'RBF kernel SVM', 'Ensemble'],
itertools.product([0, 1], repeat=2)):
clf.fit(X, y)
ax = plt.subplot(gs[grd[0], grd[1]])
fig = plot_decision_regions(X=X, y=y, clf=clf, legend=2)
plt.title(lab)
plt.show()3.2.2 用概率作为第二层模型的特征
用概率值作为最终模型的输入,这个只要加一个参数,use_probas=True,average_probas=False。这里的average_probas要设置为False,如果设置为True,概率会被平均,比如:
classifier 1: [0.2, 0.5, 0.3]
classifier 2: [0.3, 0.4, 0.4]
1) average = True :
产生的meta-feature 为:[0.25, 0.45, 0.35]
2) average = False:
产生的meta-feature为:[0.2, 0.5, 0.3, 0.3, 0.4, 0.4]
clf1 = KNeighborsClassifier(n_neighbors=1)
clf2 = RandomForestClassifier(random_state=1)
clf3 = GaussianNB()
lr = LogisticRegression()
sclf = StackingClassifier(classifiers=[clf1, clf2, clf3],
use_probas=True,
average_probas=False,
meta_classifier=lr)
print('3-fold cross validation:\n')
for clf, label in zip([clf1, clf2, clf3, sclf],
['KNN',
'Random Forest',
'Naive Bayes',
'StackingClassifier']):
scores = model_selection.cross_val_score(clf, X, y,
cv=3, scoring='accuracy')
print("Accuracy: %0.2f (+/- %0.2f) [%s]"
% (scores.mean(), scores.std(), label)) 3.2.3 特征多样性
还有一种操作是给每个基分类器分不同的特征进行训练,比如有两个分类器M1,M2,M1可以训练前半部分特征,M2可以训练后半部分特征。这个可以通过sklearn中的pipelines实现。
from sklearn.datasets import load_iris
from mlxtend.classifier import StackingClassifier
from mlxtend.feature_selection import ColumnSelector
from sklearn.pipeline import make_pipeline
from sklearn.linear_model import LogisticRegression
iris = load_iris()
X = iris.data
y = iris.target
pipe1 = make_pipeline(ColumnSelector(cols=(0, 2)),
LogisticRegression())
pipe2 = make_pipeline(ColumnSelector(cols=(1, 2, 3)),
LogisticRegression())
sclf = StackingClassifier(classifiers=[pipe1, pipe2],
meta_classifier=LogisticRegression())
sclf.fit(X, y) 3.2.4.参数详解
StackingClassifier 使用API及参数解析:
StackingClassifier(classifiers, meta_classifier, use_probas=False, average_probas=False, verbose=0, use_features_in_secondary=False)
参数:
classifiers : 基分类器,数组形式,[cl1, cl2, cl3]. 每个基分类器的属性被存储在类属性 self.clfs_.
meta_classifier : 目标分类器,即将前面分类器合起来的分类器
use_probas : bool (default: False) ,如果设置为True, 那么目标分类器的输入就是前面分类输出的类别概率值而不是类别标签
average_probas : bool (default: False),用来设置上一个参数当使用概率值输出的时候是否使用平均值。
verbose : int, optional (default=0)。用来控制使用过程中的日志输出,当 verbose = 0时,什么也不输出, verbose = 1,输出回归器的序号和名字。verbose = 2,输出详细的参数信息。verbose > 2, 自动将verbose设置为小于2的,verbose -2.
use_features_in_secondary : bool (default: False). 如果设置为True,那么最终的目标分类器就被基分类器产生的数据和最初的数据集同时训练。如果设置为False,最终的分类器只会使用基分类器产生的数据训练。
方法属性:
clfs_ : 每个基分类器的属性,list, shape 为 [n_classifiers]。
meta_clf_ : 最终目标分类器的属性
方法:
fit(X, y)
fit_transform(X, y=None, fit_params)
get_params(deep=True),如果是使用sklearn的GridSearch方法,那么返回分类器的各项参数。
predict(X)
predict_proba(X)
score(X, y, sample_weight=None), 对于给定数据集和给定label,返回评价accuracy
set_params(params),设置分类器的参数,params的设置方法和sklearn的格式一样
参考
stacking代码详解
stacking方法总结
模型融合方法概述
mlxtend官网
我的竞赛总结