决策树之CART(分类回归树)详解
决策树之CART(分类回归树)详解
- 主要内容
- CART分类回归树简介
- CART分类回归树分裂属性的选择
- CART分类回归树的剪枝
1、CART分类回归树简介
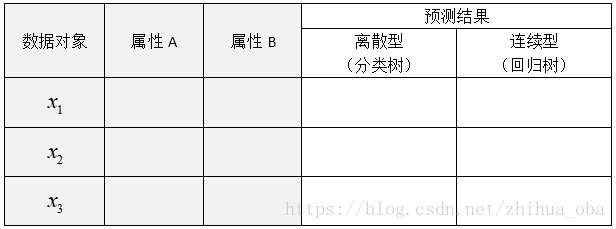
CART分类回归树是一种典型的二叉决策树,可以做分类或者回归。如果待预测结果是离散型数据,则CART生成分类决策树;如果待预测结果是连续型数据,则CART生成回归决策树。数据对象的属性特征为离散型或连续型,并不是区别分类树与回归树的标准,例如表1中,数据对象 xi x i 的属性A、B为离散型或连续型,并是不区别分类树与回归树的标准。作为分类决策树时,待预测样本落至某一叶子节点,则输出该叶子节点中所有样本所属类别最多的那一类(即叶子节点中的样本可能不是属于同一个类别,则多数为主);作为回归决策树时,待预测样本落至某一叶子节点,则输出该叶子节点中所有样本的均值。
2、CART分类回归树分裂属性的选择
2.1 CART分类树——待预测结果为离散型数据
选择具有最小 Gain_GINI G a i n _ G I N I 的属性及其属性值,作为最优分裂属性以及最优分裂属性值。 Gain_GINI G a i n _ G I N I 值越小,说明二分之后的子样本的“纯净度”越高,即说明选择该属性(值)作为分裂属性(值)的效果越好。
对于样本集 S S , GINI G I N I 计算如下:
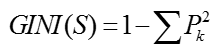
对于含有 N N 个样本的样本集 S S ,根据属性 A A 的第 i i 个属性值,将数据集 S S 划分成两部分,则划分成两部分之后, Gain_GINI G a i n _ G I N I 计算如下:
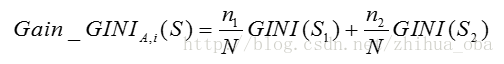
对于属性 A A ,分别计算任意属性值将数据集划分成两部分之后的 Gain_GINI G a i n _ G I N I ,选取其中的最小值,作为属性 A A 得到的最优二分方案:
对于样本集 S S ,计算所有属性的最优二分方案,选取其中的最小值,作为样本集 S S 的最优二分方案:
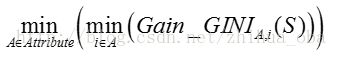
2.2 CART回归树——待预测结果为连续型数据
区别于分类树,回归树的待预测结果为连续型数据。同时,区别于分类树选取 Gain_GINI G a i n _ G I N I 为评价分裂属性的指标,回归树选取 Gain_σ G a i n _ σ 为评价分裂属性的指标。选择具有最小 Gain_σ G a i n _ σ 的属性及其属性值,作为最优分裂属性以及最优分裂属性值。 Gain_σ G a i n _ σ 值越小,说明二分之后的子样本的“差异性”越小,说明选择该属性(值)作为分裂属性(值)的效果越好。
针对含有连续型预测结果的样本集 S S ,总方差计算如下:
σ(S)=∑(yk−μ)2−−−−−−−−−−√ σ ( S ) = ∑ ( y k − μ ) 2
其中,
μ μ 表示样本集
S S 中预测结果的均值,
yk y k 表示第
k k 个样本预测结果。
对于含有 N N 个样本的样本集 S S ,根据属性 A A 的第 i i 个属性值,将数据集 S S 划分成两部分,则划分成两部分之后, Gain_σ G a i n _ σ 计算如下:
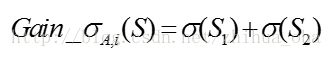
对于属性 A A ,分别计算任意属性值将数据集划分成两部分之后的 Gain_σ G a i n _ σ ,选取其中的最小值,作为属性 A A 得到的最优二分方案:

对于样本集 S S ,计算所有属性的最优二分方案,选取其中的最小值,作为样本集 S S 的最优二分方案:

3、CART分类回归树的剪枝
由于决策树的建立完全是依赖于训练样本,因此该决策树对训练样本能够产生完美的拟合效果。但这样的决策树对于测试样本来说过于庞大而复杂,可能产生较高的分类错误率。这种现象就称为过拟合。因此需要将复杂的决策树进行简化,即去掉一些节点解决过拟合问题,这个过程称为剪枝。
剪枝方法分为预剪枝和后剪枝两大类。预剪枝是在构建决策树的过程中,提前终止决策树的生长,从而避免过多的节点产生。预剪枝方法虽然简单但实用性不强,因为很难精确的判断何时终止树的生长。后剪枝是在决策树构建完成之后,对那些置信度不达标的节点子树用叶子结点代替,该叶子结点的类标号用该节点子树中频率最高的类标记。后剪枝方法又分为两种,一类是把训练数据集分成树的生长集和剪枝集;另一类算法则是使用同一数据集进行决策树生长和剪枝。常见的后剪枝方法有CCP(Cost Complexity Pruning)、REP(Reduced Error Pruning)、PEP(Pessimistic Error Pruning)、MEP(Minimum Error Pruning)。其中,悲观错误剪枝法PEP(Pessimistic Error Pruning)在 “决策树之C4.5算法详解”中有详细介绍,感兴趣的小童鞋可以了解学习。这里我们详细介绍CART分类回归树中应用最广泛的剪枝算法——代价复杂性剪枝法CCP(Cost Complexity Pruning)。
代价复杂性剪枝法CCP(Cost Complexity Pruning)主要包含两个步骤:(1)从原始决策树 T0 T 0 开始生成一个子树序列 {T0,T1,...,Tn} { T 0 , T 1 , . . . , T n } ,其中, Ti+1 T i + 1 从 Ti T i 产生, Tn T n 为根节点。(2)从第1步产生的子树序列中,根据树的真实误差估计选择最佳决策树。
CCP剪枝法步骤(1)
生成子树序列 {T0,T1,...,Tn} { T 0 , T 1 , . . . , T n } 的基本思想是从 T0 T 0 开始,裁剪 Ti T i 中关于训练数据集误差增加最小的分枝来得到 Ti+1 T i + 1 。实际上,当1棵树 T T 在节点 t t 处剪枝时,它的误差增加直观上认为是 R(t)−R(Tt) R ( t ) − R ( T t ) ,其中, R(t) R ( t ) 为在节点 t t 的子树被裁剪后节点 t t 的误差, R(Tt) R ( T t ) 为在节点 t t 的子树没被裁剪时子树 Tt T t 的误差。然而,剪枝后, T T 的叶子数减少了 L(Tt)−1 L ( T t ) − 1 ,其中, L(Tt) L ( T t ) 为子树 Tt T t 的叶子数,也就是说, T T 的复杂性减少了。因此,考虑树的复杂性因素,树分枝被裁剪后误差增加率由下式决定:
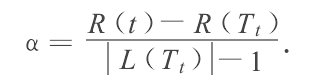
Ti+1 T i + 1 就是选择 Ti T i 中具有最小 α α 值所对应的剪枝树。
例如:图1中 ti t i 表示决策树中第 i i 个节点,A、B表示训练集中的两个类别,A、B之后的数据表示落入该节点分别属于A类、B类的样本个数。
图1,决策树中训练样本总个数为80。对于节点 t4 t 4 ,其中,A类样本46个,B类样本4个,根据大多数原则,则节点 t4 t 4 中样本为A类,故节点 t4 t 4 的子树( t8 t 8 、 t9 t 9 )被裁剪之后 t4 t 4 的误差为: 450∗5080=480 4 50 ∗ 50 80 = 4 80 。节点 t4 t 4 的子树( t8 t 8 、 t9 t 9 )被裁剪之前 t4 t 4 的误差为: 145∗4580+25∗580=380 1 45 ∗ 45 80 + 2 5 ∗ 5 80 = 3 80 。故 α(t4)=480−3802−1=0.0125 α ( t 4 ) = 4 80 − 3 80 2 − 1 = 0.0125 。类似过程,依次得到所有节点的误差增加率,如表2:
从表2可以看出,在原始树 T0 T 0 行,4个非叶节点中 t4 t 4 的 α α 值最小,因此,裁剪 T0 T 0 的 t4 t 4 节点的分枝得到 T1 T 1 ;在 T1 T 1 行,虽然 t2 t 2 和 t3 t 3 的 α α 值相同,但裁剪 t2 t 2 的分枝可以得到更小的决策树,因此, T2 T 2 是裁剪 T1 T 1 中的 t2 t 2 分枝得到的。
CCP剪枝法步骤(2)
如何根据第1步产生的子树序列 {T0,T1,...,Tn} { T 0 , T 1 , . . . , T n } ,选择出1棵最佳决策树是CCP剪枝法步骤(2)的关键。通常采用的方法有两种,一种是V番交叉验证(V-fold cross-validation),另一种是基于独立剪枝数据集。此处不在过分赘述,感兴趣的小童鞋,可以阅读参考文献[1][2][3]等。
参考文献
[1] 魏红宁. 决策树剪枝方法的比较[J]. 西南交通大学学报, 2005, 40(1):44-48.
[2] 张宇. 决策树分类及剪枝算法研究[D]. 哈尔滨理工大学, 2009.
[3] Breiman L, Friedman J H, Olshen R, et al. Classification and Regression Trees[J]. Biometrics, 1984, 40(3):358.