R语言(六)——横截面数据分类:机器学习(决策树、SVM、KNN、BP)
目录
一、决策树分类
1.普通树
2.adaboost分类
3.bagging分类
4.随机森林
三、支持向量机(SVM)分类
四、最近邻方法(KNN)分类
五、神经网络分类
1.拟合
2.不同参数的效果分析
六、总结
一、数据
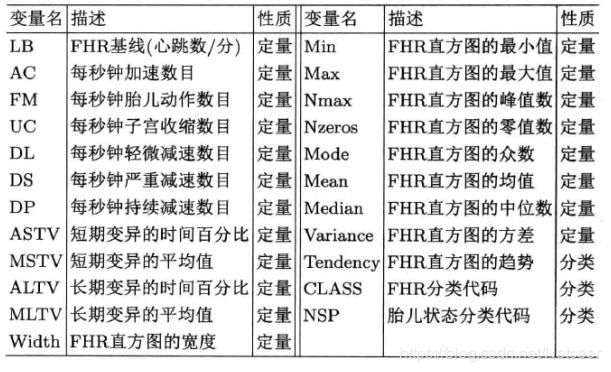
胎心宫缩监护(CTG.xls)数据有2129个观测值及23个变量,包含了致命心律的各种度量以及基于监护记录的由专家分类的公所特征。数据可以从http://archive.ics.uci.edu/ml/machine-learning-databases/00193/获得。最后三个分类变量的水平为:
| 变量 | 水平 | 含义 |
| Tendency(FHR直方图趋势) | -1 | 左不对称 |
| 0 | 对称 | |
| 1 | 右不对称 | |
| CLASS(FHR分类代码) | 1-10 | 平静-可疑的十种情况 |
| 1NSP(胎儿状态分类代码) | 1 | 正常 |
| 2 | 疑似 | |
| 3 | 病态 |
我们尝试用前面的头22个变量作为自变量预测作为因变量的NSP类别。查看数据后,删去3个有缺失值的观测值,形成新的CTG.NAOMIT.csv数据,可以直接使用我上传的经过处理的数据。
接下来使用决策树、分类树、adaboost、bagging、随机森林、SVM、最近邻等方法。其中一些已经在R语言(四)——十折交叉验证/机器学习回归【决策树(随机森林)、组合方法、SVR】中实验过,所以后面只需要进行分类测试。
使用Fold()函数把观测值随机分为10份。读入数据,并把用哑元表示的定性变量因子化:
Fold=function(Z=10,w,D,seed=7777){
n=nrow(w);d=1:n;dd=list()
e=levels(w[,D])
t=length(e)
set.seed(seed)
for(i in 1:t){
d0=d[w[,D]==e[i]]
j=length(d0)
ZT=rep(1:Z,ceiling(j/Z))[1:j]
id=cbind(sample(ZT,length(ZT)),d0)
dd[[i]]=id
}
#每个dd[[i]]是随机1:Z及i类的下标集组成的矩阵
mm=list()
for(i in 1:Z){
u=NULL
for(j in 1:t)u=c(u,dd[[j]][dd[[j]][,1]==i,2])
mm[[i]]=u #mm[[i]]为第i个下标集
}
return(mm) #输出Z个下标集
}
w=read.csv("CTG.NAOMIT.csv")
F=21:23 #因变量
for(i in F) w[,i]=factor(w[,i]) #因子化
D=23;Z=10;n=nrow(w);mm=Fold(Z,w,D,8888) #D是因变量位置,Z是折数一、决策树分类
1.普通树
分类树和回归树基本相同,首先把全部数据作为训练集来估计决策树:
#决策树
library(rpart.plot)
a=rpart(NSP~.,w) #用全部数据拟合
rpart.plot(a,type=2,extra=4) #画决策树产生了以下的打印结果和决策树
在决策树中出现两次分叉,都用了变量CLASS。当CLASS=1,2,3,4,6,7时,走向左边分支,以此类推,最终得到各分支终点处三种类别的数量比例。为了得到训练集的分类误差,我们可以查看混淆矩阵并计算误差率
wp=predict(a,w,type="class")
z=table(w[,D],wp)
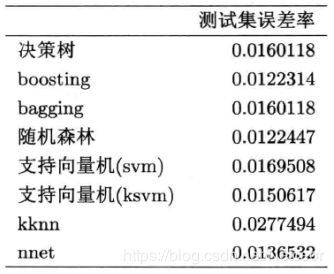
sum(w[,D]==wp)/nrow(w)共34个观测值被分错,误判率0.016。
接下来进行交叉验证:
#交叉验证
E=rep(0,Z)
for(i in 1:Z){
m=mm[[i]]
n1=length(m)
a=rpart(NSP~.,w[-m,])
E[i]=sum(w[m,D]!=predict(a,w[m,],type="class"))/n1
}
mean(E)得到误差率0.016
2.adaboost分类
adaboost是boosting的一种,为一种迭代式的组合方法,可以译为“自适应助推法”。所用的基础分类器(这里为决策树)随着迭代的进行,不断通过自助法(bootstrap)加权再抽样,根据新的样本改进分类器。每一次迭代都针对前一个分类器的某些观测值的误判加以修正,通常是采用增加(误判)或减少(正确分类)权重的办法。这样在新的样本中就可能有更多前一次分错的观测值,再形成一个新的分类器进入下一轮迭代,而且每轮迭代都对这一轮产生的分类器给出误判率,最终结果由各个阶段的分类器按照误判率加权投票产生,即为“自适应”。
它的缺点式对奇异点或离群点比较敏感,但其优点是对过拟合不那么敏感。这里使用的程序包是adabag,包含boosting()函数以及之后会使用的bagging()函数。boosting函数的默认迭代次数是100次,即产生100棵决策树。
同样先利用全部数据拟合,可用a$importance画出变量重要性图
library(adabag)
set.seed(4410)
a=boosting(NSP~.,w)
wp=predict(a,w)$class
z=table(w[,D],wp)
sum(w[,D]!=wp)/nrow(w)
barplot(a$importance,cex.name=.6) #变量重要性得到误判率0.00047。如果使用a$trees命令,会打印出boosting过程决策树产生的所有树,按照默认值会打印出100棵树.....如果要打印个别的树可用如a$trees[[1]]、rpart.plot(a$trees[[100]],type=2,extra=4)画出其中一棵。
交叉验证:
#交叉验证
set.seed(1010)
E=rep(0,Z)
for(i in 1:Z){
m=mm[[i]]
n1=length(m)
a=boosting(NSP~.,w[-m,])
E[i]=sum(as.character(w[m,D])!=predict(a,w[m,])$class)/n1
}
mean(E)得到平均误判率0.0122,看起来结果不如全部数据的好,但这个才更接近模型的真实预测误差。
3.bagging分类
bagging可以译为“自助整合法”。它对训练样本做许多次(比如k次)放回抽样,每次抽取样本量相同的观测值,由于是放回抽样,因此有了k个不同的样本。对每个样本生成一棵决策树,每棵树都对一个新的观测值产生一个预测。如果目的是分类,那么由这些树的分类结果来“投票”产生分类结果。与boosting相同,其默认迭代次数也是100。
#bagging分类
#拟合
set.seed(1010)
D=23
a=bagging(NSP~.,w)
wp=predict(a,w)$class
z=table(w[,D],wp)
sum(w[,D]!=wp)/nrow(w)
barplot(a$importance,cex.names = 0.8) #变量重要性
#交叉验证
w=read.csv("CTG.NAOMIT.csv")
F=21:23;D=23;Z=10;n=nrow(w)
mm=Fold(Z,w,d,8888)
for(i in F)w[,i]=factor(w[,i])
set.seed(1010)
E=rep(0,Z)
for(i in 1:Z){
m=mm[[i]]
n1=length(m)
a=bagging(NSP~.,w[-m,])
E[i]=sum(as.character(w[m,D])!=predict(a,w[m,])$class)/n1
}
mean(E)计算结果显示,平均误判率为0.016
4.随机森林
随机森林和使用决策树作为基本分类器的bagging有些类似,也是进行许多次自助放回抽样,多得到的样本数目及由此建立的决策树数量要远远多于bagging的样本数目。随机森林与bagging的关键区别在于,在生成每棵树的时候,每个节点的变量都仅仅在随机选出的少数变量中产生。因此不但样本是随机的,每个节点变量的产生都有很大的随机性。
随机森林让每棵树尽可能生长,而且不进行修剪。随机森林不惧怕很大的维数,即使有数千个变量也不必删除,还会给出分类中各个变量的重要性。这里使用randomForest程序包中的randomForest()函数,其建立棵树的默认值是500。
#随机森林分类
library(randomForest)
set.seed(1010)
a=randomForest(NSP~.,w,importance=TRUE,proximity=TRUE)
wp=predict(a,w)
z=table(w[,D],wp)
E0=(sum(z)-sum(diag(z)))/sum(z)
#绘制重要性图
#a$importance
par(mfrow=c(3,1))
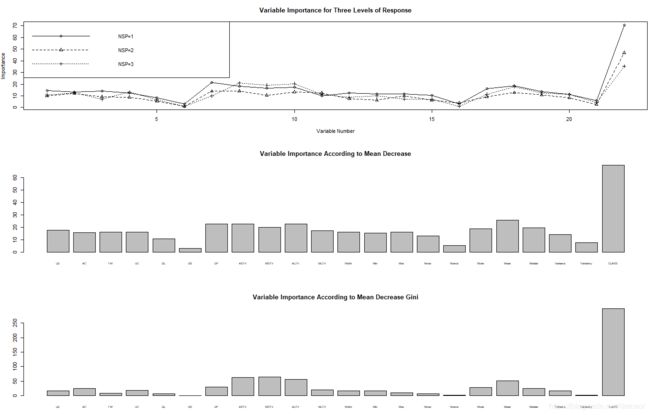
matplot(importance(a)[,1:3],type="o",pch=1:3,lty=1:3,
col=1,xlab="Variable Number",ylab="Importance")
title("Variable Importance for Three Levels of Response")
legend("topleft",legend=paste("NSP=",1:3,sep=""),
pch=1:3,lty=1:3,col=1)
barplot(importance(a)[,4],cex.names = 0.6)
title("Variable Importance According to Mean Decrease")
barplot(importance(a)[,5],cex.names = 0.6)
title("Variable Importance According to Mean Decrease Gini")
par(mfrow=c(1,1))
#交叉验证
set.seed(1010)
E=rep(0,Z)
for(i in 1:Z){
m=mm[[i]]
n1=length(m)
a=randomForest(NSP~.,data=w[-m,])
E[i]=sum(w[m,D]!=predict(a,w[m,]))/n1
}
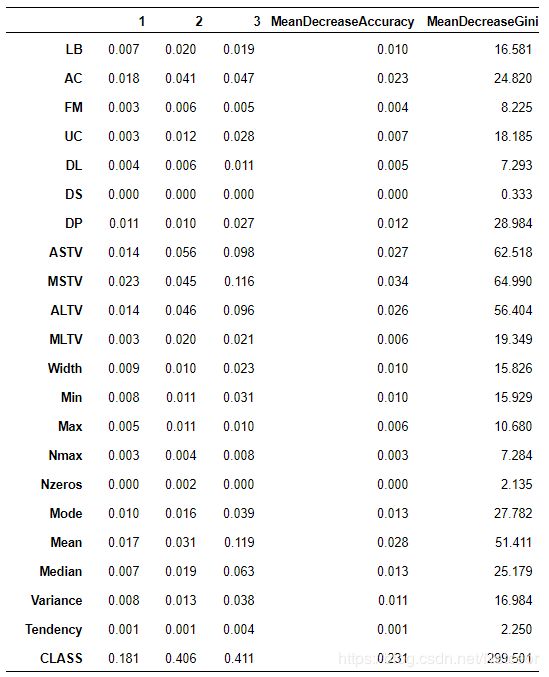
mean(E)各个变量的相对重要性可用a$importance打印出来,前三列显示了各个变量对NSP三个水平的相对影响,后两列分别展示了扰动这些变量对精确度及Gini指数的影响,相对影响大的被认为是重要变量。
也可以绘图显示:
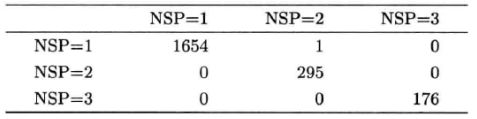
由于每棵树都由自助法抽样得到,抽样是放回抽样,每次都有30%-40%的数据没有被抽到,使得它们成为天然的测试集,这些观测值数据被成为OOB(out of bag)数据。用print(a)可以得到一个误差率的OOB估计及基于OOB的分类矩阵。打印结果如下:
这里给出OOB测试集得到的误判率是1.27%。
进行交叉验证,得到平均误判率0.0122,与基于OOB的误判率差距不大,说明其是一种非常聪明的误差验证指标。
三、支持向量机(SVM)分类
R语言中包含多种可以调用SVM的程序包,此处使用e1071中的svm()和kernlab中的ksvm()函数来做分类。
由于代码相似度极大,因此用注释来区分两种方式。
#支持向量机分类
library(e1071)
#library(kernlab)
a=svm(NSP~.,data=w,kernal="sigmoid")
#a=ksvm(NSP~.,data=w)
wp=predict(a,w)
z=table(w[,D],wp)
E=(sum(z)-sum(diag(z)))/sum(z)
#交叉验证
E=rep(0,Z)
for(i in 1:Z){
m=mm[[i]]
n1=length(m)
a=svm(NSP~.,data=w[-m,],kernal="sigmoid")
#a=ksvm(NSP~.,data=w[-m,])
E[i]=sum(w[m,D]!=predict(a,w[m,]))/n1
}
mean(E)e1071包运行全部数据的误判率为0.0108,kernlab包运行的全部数据的误判率为0.0085
交叉验证的结果:e1071平均误判率0.01695,kernlab平均误判率0.01506。
四、最近邻方法(KNN)分类
最近邻方法可能是所有算法建模中最简单的方法。每个回归或分类问题都有一些自变量,它们组成一个多为空间。首先在空间中假定一个距离。比如,在连续型自变量的情况下,可用欧氏距离、平方欧氏距离、绝对距离等。在分类问题中,测试集的一个点应该被判为训练集中离该点最近的k个点中多数点所属的类别。这种“多数投票方法”一般都加有权重,离得越近的点权重越大。
这里利用kknn程序包中的kknn()函数。
#最近邻方法分类
#全部数据拟合
library(kknn)
a=kknn(NSP~.,k=6,train=w,test=w) #k是交叉验证得到的
z=table(w[,D],a$fit)
E=(sum(z)-sum(diag(z)))/sum(z)
#交叉验证
E=rep(0,Z)
for(i in 1:Z){
m=mm[[i]]
n1=length(m)
a=kknn(NSP~.,k=6,train=w[-m,],test=w[m,])
E[i]=sum(w[m,D]!=a$fit)/n1
}
mean(E)得到平均误判率0.0277,调整k值后误判率也会相应改变。经试验,对于此数据而言,k=6时有较低的平均误判率
五、神经网络分类
1.拟合
#神经网络分类
library(nnet)
a=nnet(NSP~.,data=w,subset=1:n,size=2,rang=0.01,
decay=5e-4,maxit=200)
wp=predict(a,w[m,],type="class")
sum(w[m,D]!=wp)/length(m)
#交叉验证
E=rep(0,Z)
for(i in 1:Z){
m=mm[[i]]
mc=setdiff(1:n,m)
a=nnet(NSP~.,data=w,subset=mc,size=2,rang=0.1,
decay=0.01,maxit=200)
E[i]=sum(w[m,D]!=predict(a,w[m,],type="class"))/length(m)
}
mean(E)得到误判率0.01365
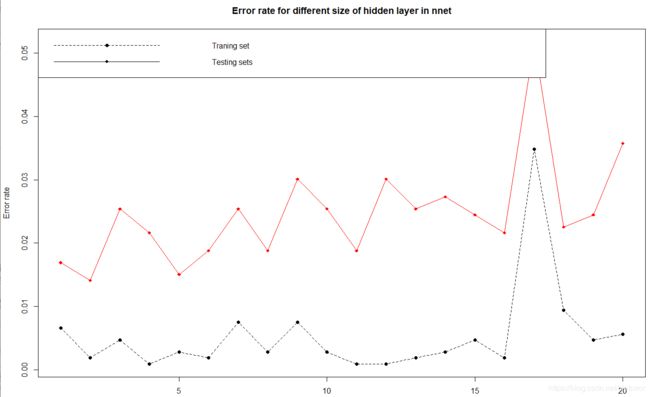
2.不同参数的效果分析
隐藏层节点个数是神经网络的重要参数之一,我们考虑其对训练集和测试集的误判率的影响,固定参数delay=0.01,从1-20变化size(即隐藏层节点个数),绘制训练集和测试集的误差变化图。这里把数据随机分成两组测试:
#不同参数拟合效果
set.seed(1010)
d1=sample(1:n,n/2)
d2=setdiff(1:n,d1)
ww=matrix(0,20,2)
for(s in 1:20){
a=nnet(NSP~.,data=w,subset=d1,size=s,rang=0.1,
decay=0.01,maxit=200)
ww[s,1]=sum(w[d1,D]!=predict(a,w[d1,],type="class"))/length(d1)
ww[s,2]=sum(w[d2,D]!=predict(a,w[d2,],type="class"))/length(d2)
matplot(1:20,ww,type="o",lty=2:1,pch=c(16,18),
ylab="Error rate",xlab="Size")
legend("topleft",c("Traning set","Testing sets"),
pch=c(16,18),lty=2:1)
title("Error rate for different size of hidden layer in nnet")
}六、总结
对于每种方法,都利用Fold()函数产生数据的10个子集,并进行交叉验证,绘制图标查看不同模型的预测效果。