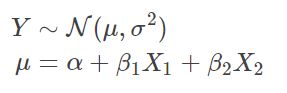贝叶斯学习笔记
概率编程允许在用户自定义的概率模型上进行自动贝叶斯推断。
本文主要基于概率编程的一个常用框架——PyMC3进行操作。PyMC3使用Theano通过变分推理进行梯度计算,并使用了C实现加速运算。PyMC3具有先进的下一代MCMC采样算法如No-U-Turn Sampler (NUTS; Hoffman, 2014)和Hamiltonian Monte Carlo自整定变体(HMC; Duane, 1987)。这类采样算法在高维和复杂的后验分布上具有良好的效果,允许对复杂模型进行拟合而不需要对拟合算法有特殊的了解。NUTS和HMC算法从似然函数中获得梯度信息,因此其收敛速度比传统采样方法快很多,特别是针对大模型。
预备知识
1、各种分布的基本特点:指数分布、Beta分布、Gamma分布等常见分布。其中Beta分布具有处于0~1之间,适合表征概率参数的概率分布(如抛硬币正面朝上的概率p的概率分布)。
2、离散分布的PMF、连续分布的PDF,以及CDF。
推荐另一个值得一读的博客:浅谈贝叶斯和MCMC
案例1:贝叶斯线性回归
本案例来自:PyMC3 - 简介和入门、Getting started with PyMC3
对于线性回归,频率学派采用最小二乘法等方法求解系数,那么贝叶斯学派怎么思考这个问题呢?
考虑一个简单的贝叶斯线性回归模型,其参数具有正态分布(Normal)先验。我们预测的具有正态分布的观测值 Y ,其期望 μ 是两个预测变量(X1、X2)的线性组合:
其中 α 是截距, βi 是变量 Xi 的系数; σ 代表观察误差。
建立贝叶斯模型,模型中的未知变量需要赋予先验分布,这里选择零均值的正态先验。其中,系数 α 、 βi 的方差为100,代表参数的弱信息。选择半正态分布作为观测误差 σ 的先验分布(半正态分布以0为边界的、形态为正态分布的一半)。
代码:
import numpy as np
import matplotlib.pyplot as plt
import pymc3 as pm
# 模拟观察数据
np.random.seed(123)
alpha=1
sigma=1
beta =[1, 2.5]
N=100
X1=np.random.randn(N)
X2=np.random.randn(N)
Y=alpha + beta[0]*X1 + beta[1]*X2 + np.random.randn(N)*sigma
# 建立贝叶斯模型
basic_model = pm.Model()
with basic_model:
# 设置各参数的先验信息,使用随机变量(stochastic)
alpha=pm.Normal('alpha',mu=0,sd=10)
beta=pm.Normal('beta',mu=0,sd=10,shape=2)
sigma=pm.HalfNormal('sigma',sd=1)
# 构建线性回归关系,使用确定性变量(deterministic)
mu=alpha+beta[0]*X1+beta[1]*X2
# 设置Y的先验,并加入Y的观察数据
Y_obs=pm.Normal('Y_obs',mu=mu,sd=sigma,observed=Y)
## 采样推断模型参数
# 用MAP获得初始点
start = pm.find_MAP(fmin=optimize.fmin_powell)
# 实例化采样器
step = pm.Slice(vars=[sigma])
# 对后验分布进行5000次采样
trace = pm.sample(5000, step=step,start=start)说明:
为了使用MCMC采样以获得后验分布的样本,PyMC3需要指定和特定MCMC算法相关的步进方法(采样方法),如Metropolis, Slice sampling, or the No-U-Turn Sampler (NUTS)。PyMC3中的 step_methods 子模块提供了如下采样器: NUTS, Metropolis, Slice, HamiltonianMC, and BinaryMetropolis。可以由PyMC3自动指定也可以手动指定。自动指定是根据模型中的变量类型决定的:
* 二值变量:指定 BinaryMetropolis
* 离散变量:指定 Metropolis
* 连续变量:指定 NUTS
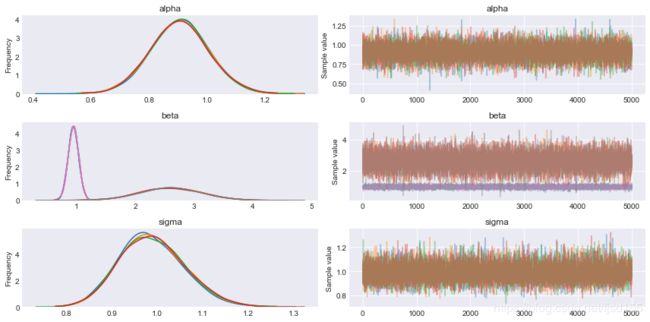
PyMC3提供了 traceplot 函数来绘制后验采样的趋势图。
pm.traceplot(trace)
案例2:贝叶斯A/B测试
摘自:Bayesian Methods for Hackers: Probabilistic Programming and Bayesian Inference
有一批关于A药和B药治疗某种疾病的数据(是否治愈),我们想知道两个药的治愈率哪个更高。假定药物治愈该疾病服从伯努利分布。频率学派常采用 t 检验等方法进行测试,那么贝叶斯学派又是如何测试呢?
代码:
import pymc3 as pm
figsize(12, 4)
# 真实值,但现实情况中我们并不知道
true_p_A = 0.05
true_p_B = 0.04
# notice the unequal sample sizes -- no problem in Bayesian analysis.
N_A = 1500
N_B = 750
# 模拟观察数据
observations_A = stats.bernoulli.rvs(true_p_A, size=N_A)
observations_B = stats.bernoulli.rvs(true_p_B, size=N_B)
# 贝叶斯建模推断
with pm.Model() as model:
# 设置先验
p_A = pm.Uniform("p_A", 0, 1)
p_B = pm.Uniform("p_B", 0, 1)
# Define the deterministic delta function. This is our unknown of interest.
delta = pm.Deterministic("delta", p_A - p_B)
# Set of observations, in this case we have two observation datasets.
obs_A = pm.Bernoulli("obs_A", p_A, observed=observations_A)
obs_B = pm.Bernoulli("obs_B", p_B, observed=observations_B)
# 设置step方法,采样后丢弃前1000样本(burn策略)
step = pm.Metropolis()
trace = pm.sample(20000, step=step)
burned_trace=trace[1000:]
p_A_samples = burned_trace["p_A"]
p_B_samples = burned_trace["p_B"]
delta_samples = burned_trace["delta"]
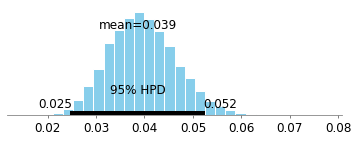
pm.plots.plot_posterior(trace=p_A_samples)
pm.plots.plot_posterior(trace=p_B_samples)
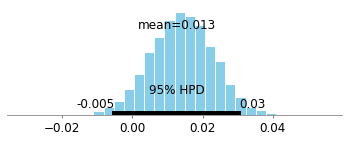
pm.plots.plot_posterior(trace=delta_samples)
print("Probability site A is BETTER than site B: %.3f" % \
np.mean(delta_samples > 0))Probability site A is BETTER than site B: 0.918
案例3:抛硬币与共轭分布
这是一个极其简单的例子,此处举例主要是为了引出“共轭分布”的概念。
我们都知道抛硬币正面朝上的概率为0.5(硬币没被做手脚的情况下,并且本案例也默认真实概率为0.5),频率学派直接用频率推断概率。那么贝叶斯学派是怎么推断这个概率的呢?贝叶斯学派先要假定一个先验概率,我们假定先验知识为:正面朝上的概率服从0~1之间的均匀分布(我们一无所知),随着抛硬币实验结果一个个出现,我们用实验结果逐步更新后验概率。代码如下
%matplotlib inline
from IPython.core.pylabtools import figsize
import numpy as np
from matplotlib import pyplot as plt
figsize(11, 9)
import scipy.stats as stats
dist = stats.beta
n_trials = [0, 1, 2, 3, 4, 5, 8, 15, 50, 500]
data = stats.bernoulli.rvs(0.5, size=n_trials[-1])
x = np.linspace(0, 1, 100)
# For the already prepared, I'm using Binomial's conj. prior.
for k, N in enumerate(n_trials):
sx = plt.subplot(len(n_trials)/2, 2, k+1)
plt.xlabel("$p$, probability of heads") \
if k in [0, len(n_trials)-1] else None
plt.setp(sx.get_yticklabels(), visible=False)
heads = data[:N].sum()
y = dist.pdf(x, 1 + heads, 1 + N - heads)
plt.plot(x, y, label="observe %d tosses,\n %d heads" % (N, heads))
plt.fill_between(x, 0, y, color="#348ABD", alpha=0.4)
plt.vlines(0.5, 0, 4, color="k", linestyles="--", lw=1)
leg = plt.legend()
leg.get_frame().set_alpha(0.4)
plt.autoscale(tight=True)
plt.suptitle("Bayesian updating of posterior probabilities",
y=1.02,
fontsize=14)
plt.tight_layout()注意到,我们直接使用公式更新了概率分布,而不是每次都进行MCMC采样推断。这个公式利用了一个重要的原理:
一个Beta先验分布连同二项式生成的观测数据将形成一个Beta后验分布。即:
若 p~Beta(α, β),且 X~Binomial(N, p),则 p|X ~ Beta(α+X, β+N-X) 。
注意:均匀分布等价于Beta(1, 1),即服从Beta分布 。
我们称Beta分布是二项分布的共轭分布。更多的共轭分布,可以查看维基百科的共轭先验表。比如Dirichlet 分布是多项式分布的共轭分布(Beta分布和二项式分布的多维推广)。对于简单的一维问题,可以尝试这种方法。但是,对于复杂的问题,往往很难利用共轭分布这个技巧,MCMC采样推断仍然是最重要的。
需要强调的是,对于一个基本贝叶斯推断问题,至少存在2个分布类型需要设置:所需估计的参数的先验分布,观察数据的分布。先验分布根据经验进行设定,可以存在一定的主观性,先验的分布类型选错了也不会导致特别大的问题(一般情况下);观察数据的分布,是真正的贝叶斯模型,是观察数据产生的框架,分布类型如果没选好,模型也就难以拟合。贝叶斯推断,是基于这个假定的模型框架,根据观察数据去推断这个模型框架的各个参数值。在贝叶斯推断里,复杂的变换可以通过建立逻辑关系映射到最终的因变量,然后为因变量选定分布类型,使得观察数据的产生服从这个分布类型。
案例4:概率质量函数推断似然函数
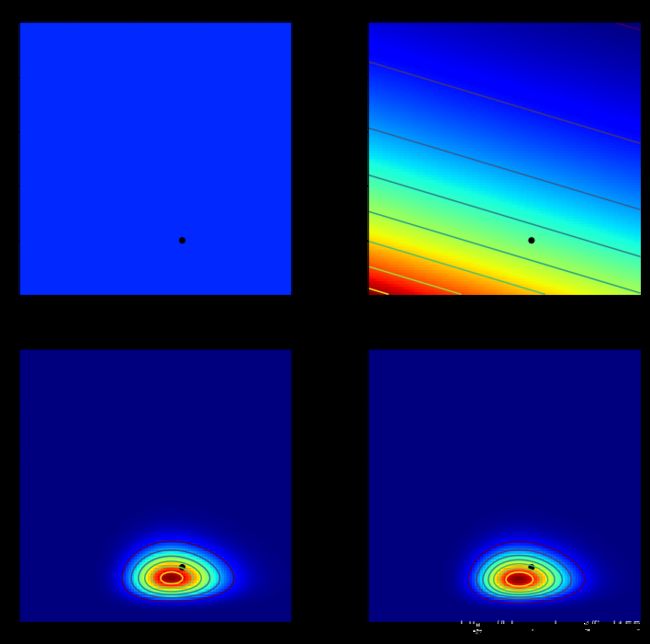
Poisson分布的概率质量函数在参数 lambda 处取得最大值(这一点不容忽视)。假设当前假定的lambda值为b(所有可能的lambda值通过枚举产生),利用观察数据a和模拟的 lambda值b 可以求出观察数据a处的概率密度值;若有多个观察数据,则可将多个概率密度值累乘,从而估算出参数b为真实lambda值的可能性。将这个可能性与先验分布相乘,则可得到后验分布。
%matplotlib inline
import scipy.stats as stats
from IPython.core.pylabtools import figsize
import numpy as np
import matplotlib.pyplot as plt
jet = plt.cm.jet
# sample size of data we observe, trying varying this (keep it less than 100 ;)
N = 10
# the true parameters, but of course we do not see these values...
lambda_1_true = 1
lambda_2_true = 3
#...we see the data generated, dependent on the above two values.
data = np.concatenate([
stats.poisson.rvs(lambda_1_true, size=(N, 1)),
stats.poisson.rvs(lambda_2_true, size=(N, 1))
], axis=1)
print("observed (2-dimensional,sample size = %d):" % N, data)
# plotting details.
x = y = np.linspace(.01, 5, 100)
likelihood_x = np.array([stats.poisson.pmf(data[:, 0], _x)
for _x in x]).prod(axis=1)
likelihood_y = np.array([stats.poisson.pmf(data[:, 1], _y)
for _y in y]).prod(axis=1)
L = np.dot(likelihood_x[:, None], likelihood_y[None, :])
figsize(12.5, 12)
# matplotlib heavy lifting below, beware!
plt.subplot(221)
uni_x = stats.uniform.pdf(x, loc=0, scale=5)
uni_y = stats.uniform.pdf(x, loc=0, scale=5)
M = np.dot(uni_x[:, None], uni_y[None, :])
im = plt.imshow(M, interpolation='none', origin='lower',
cmap=jet, vmax=1, vmin=-.15, extent=(0, 5, 0, 5))
plt.scatter(lambda_2_true, lambda_1_true, c="k", s=50, edgecolor="none")
plt.xlim(0, 5)
plt.ylim(0, 5)
plt.title("Landscape formed by Uniform priors on $p_1, p_2$.")
plt.subplot(223)
plt.contour(x, y, M * L)
im = plt.imshow(M * L, interpolation='none', origin='lower',
cmap=jet, extent=(0, 5, 0, 5))
plt.title("Landscape warped by %d data observation;\n Uniform priors on $p_1, p_2$." % N)
plt.scatter(lambda_2_true, lambda_1_true, c="k", s=50, edgecolor="none")
plt.xlim(0, 5)
plt.ylim(0, 5)
plt.subplot(222)
exp_x = stats.expon.pdf(x, loc=0, scale=3)
exp_y = stats.expon.pdf(x, loc=0, scale=10)
M = np.dot(exp_x[:, None], exp_y[None, :])
plt.contour(x, y, M)
im = plt.imshow(M, interpolation='none', origin='lower',
cmap=jet, extent=(0, 5, 0, 5))
plt.scatter(lambda_2_true, lambda_1_true, c="k", s=50, edgecolor="none")
plt.xlim(0, 5)
plt.ylim(0, 5)
plt.title("Landscape formed by Exponential priors on $p_1, p_2$.")
plt.subplot(224)
# This is the likelihood times prior, that results in the posterior.
plt.contour(x, y, M * L)
im = plt.imshow(M * L, interpolation='none', origin='lower',
cmap=jet, extent=(0, 5, 0, 5))
plt.scatter(lambda_2_true, lambda_1_true, c="k", s=50, edgecolor="none")
plt.title("Landscape warped by %d data observation;\n Exponential priors on \
$p_1, p_2$." % N)
plt.xlim(0, 5)
plt.ylim(0, 5)
补充知识
理性使用MCMC采样点
1、MCMC采样点的初期阶段,采样不够成熟,可考虑舍弃初期预热的采样点,即采用burn策略;此外,可以利用MAP方法(包括fmin、Powell等方法)设定MCMC的起点来缩短预热期。
2、MCMC采样具有自相关性(不容忽视的概念),为了保证采样的独立性,可考虑进行稀释(每隔K个点纳入最终分析)。
贝叶斯建模的基本思路
我们拿到一堆观察数据,思考这些数据是如何产生的是进行贝叶斯建模的不错的开始。
1、什么样的随机变量最可能产生这些观察数据?也就是说,我们需要选定一个分布类型。
2、所选定的分布类型,需要哪些参数?这些参数又是怎么产生的?比如某些参数服从某种分布,比如某些参数是其他参数推导来的。如果需要的参数服从某种分布,那么那种分布又需要哪些参数来构建?
3、重复2的思考,直到可以放心地设置某个参数的先验分布。
总结一下:贝叶斯建模的关键在于,理解观察数据产生的随机规律(分布类型)(对应于概率模型),感知参数产生的规律(对应于先验分布),归纳参数之间的逻辑规律(对应于推导变换)。
概率模型的诊断
1、利用当前推断的参数(得到的模型)生存新的观测数据,比较新数据与旧数据的相似性。
2、贝叶斯p值。
3、分离图(separation plots)。
更多案例
其他典型案例见 Bayesian Methods for Hackers: Probabilistic Programming and Bayesian Inference
如:用户短信行为变化、学生作弊、挑战者号事故、无监督聚类等等。
用户短信行为和无监督聚类的例子,本质上是类似的。前者在数据上有两个参数不同的Poisson分布(lambda不同),后者在数据上有两个参数不同的正态分布(均值和方差不同)。都可以采用共同的随机变量,推断不同的参数。注意构建共同变量时采用的技巧。
学生作弊问题采用了两种贝叶斯推断模型,能对比两种思路可知,灵活的数学推导思路直接影响了贝叶斯建模。理解背后的数学规律、观察数据如何产生,这些都很重要。
挑战者号事故,本质上是在拟合Logistic回归,可与“贝叶斯线性回归”案例对比。
贝叶斯推断不仅可以解决频率学派能解决的统计学问题,贝叶斯还能解决一些频率学派不太好解决的情景。之后有时间将对贝叶斯的优势进行简单举例和总结。本文的案例都比较简单,对于复杂的问题,需要挑选合适的先验分布,这其实并不简单;如何为不同参数选择合适的MCMC具体算法(如 M-H、NUTS、HMC等)也不简单。
参考资料
Bayesian Methods for Hackers: Probabilistic Programming and Bayesian Inference
贝叶斯方法:概率编程与贝叶斯推断
PyMC3 - 简介和入门
Getting started with PyMC3