机器学习-学习笔记 聚类
聚类
聚类任务
背景
在无监督学习(密度估计、异常检测等)中,训练样本的标记信息是未知的(即不人为指定),旨在发现数据之间的内在联系和规律,为进一步的数据分析提供基础。
此类学习任务中研究最多、应用最广的是聚类。
定义
聚类将数据集中的样本划分为若干个通常是不相交(有的时候会相交)的子集(簇,也叫类),即将样本分类,但是分类后得到的簇的标记是未指定的,这里需要使用者自行定义。
作用
聚类既能作为一个单独过程,用于寻找数据内在的分布结构,可以作为分类等其他学习任务的前驱过程。
主要概括起来,就是寻找数据内在的规律和联系,进行整合,分类,得到的一些数据集(类)。
性能度量(有效性指标)
类似于监督性学习的性能度量。
聚类理想情况就是,簇内相似度高,簇间相似度低。
聚类的性能度量大致有两类,分为外部指标(具有参考模型)和内部指标(直接考察聚类结果)。
外部指标举例的话,比如给你一群人的特征,一种理想情况就是聚类为男|女。
外部指标
书上根据上面的数据给出了如下四个式子
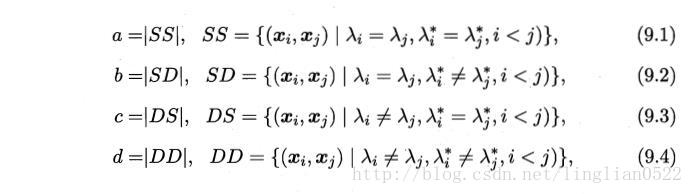
其中 第一个S 表示C中属于相同簇, D 表示C中不属于相同簇, 第二个S 表示 C∗ 中属于相同簇,D表示 C∗ 中不属于相同簇。
很明显,只有这四种情况,所有得出 a + b + c + d = m (m - 1) / 2,其中m (m - 1) / 2 为所有数据两两相对的情况数。
基于上面的四个式子可以导出下面这些常用的聚类性能度量外部指标:
- Jaccard系数(JC)
JC=aa+b+c - FM指数(FMI)
FMI=aa+b⋅aa+c−−−−−−−−−−√ - Rand指数(RI)
RI=2(a+d)m(m−1)
显然,上述的三个性能度量的结果值均在[0, 1]区间,值越大越好。
内部指标
接着,又能得出下面这四个式子(基于样本之间的距离dist来得出: avg簇内平均,diam簇内最远,min簇间最近,cen簇间中心点间的距离)

基于上面四个式子,我们可以导出下面这些常用的聚类性能度量的内部指标
- DB指数(DBI)
DBI=1k∑i=1kmaxj≠i(avg(Ci)+avg(Cj)dcen(ui,uj))
分析可知,上式求得的就是簇之间两两比较,计算出二个簇的平均距离之和除以二个簇之间的中心距离,取最大值,并求和,最后得出的结果代表着簇间相似度,为什么呢?
这里就要说一下,dist,是一个函数,能够计算二个样本之间的距离,那么,这个得出的距离是什么呢?就是二者的相似性,距离越大,代表相似性越低,差异性越高,那么,cen就代表着二个簇之间的相似性,越小越相似,即上述式子就越大。
但是为什么是簇间相似度而不是簇内相似度,他计算的是cen簇内中间距离,得出的是二个簇之间的相似度。
- Dunn指数(DI)
DI=min1⩽i⩽k{minj≠i(dmin(Ci,Cj)max1⩽l⩽kdiam(Cl))}
类似于DBI的分析,也可以得出DI代表的是簇内相似度,比较的是diam簇内最远。
综上所述,DBI越小越号,DI越大越好(不懂分子那里为什么)。
距离计算
这里主要说的就是上面说的dist函数,若他是一个距离度量,则需满足一些基本性质
- 非负性(即算出的差异性不能为负数)
dist(xi,xj)⩽0 - 同一性(即自己跟自己距离要为0)
dist(xi,xj)=0当且仅当xi=xj - 对称性(即二点之间距离从这走和从对面走是一样的)
dist(xi,xj)=dist(xj,xi) - 直递性(三角不等式)
dist(xi,xj)⩽dist(xi,xk)+dist(xk,xj)
闵可夫斯基距离(MinkovDM)
预备知识:范数
上面这个式子就是范数 ||xi−xj||p
当p=2时,计算的就是欧式距离。
当p=1时,计算的就是曼哈顿距离(街区距离)。
属性分为连续属性(数值属性)和离散属性(列名属性),连续属性一般为有序属性,离散属性一般为无序属性,闵可夫斯基距离可用于有序属性的计算, 如果需要对无序属性进行计算,则需要使用VDM。
VDM(不同值的标量)

看到这里,感觉就是在计算二个值(a,b)在不同簇被引用的比例。
接着,我们就可以使用以下这些小连招- -,处理混合属性的时候

有的时候,距离度量可能不满足直递性,这样的距离成为非度量距离。
原型聚类(基于原型的聚类)
此类算法假设聚类结构能通过一组原型刻画,在现实聚类任务中极为常用。
原理
先对原型进行初始化,然后对原型进行迭代更新求解,采用不同的原型表示、不同的求解方式,将产生不同的算法。
k均值算法(kmeans)
![]()
算法流程
![]()
kmeans(X,k)
idx = kmeans(X,k)
idx = kmeans(X,k,Name,Value)
[idx,C] = kmeans(_)
[idx,C,sumd] = kmeans(_)
[idx,C,sumd,D] = kmeans(_)
X :N*P的数据矩阵
K: 表示将X划分为几类,为整数
Idx :N*1的向量,存储的是每个点的聚类标号
C: K*P的矩阵,存储的是K个聚类质心位置
sumd 1*K的和向量,存储的是类间所有点与该类质心点距离之和
D N*K的矩阵,存储的是每个点与所有质心的距离
[…]=Kmeans(…,’Param1’,Val1,’Param2’,Val2,…)
这其中的参数Param1、Param2等,主要可以设置为如下:
- ‘Distance’(距离度量)
‘sqEuclidean’ 欧式距离(默认时,采用此距离方式)
‘cityblock’ 绝度误差和,又称:L1
‘cosine’ 针对向量
‘correlation’ 针对有时序关系的值
‘Hamming’ 只针对二进制数据 - ‘Start’(初始质心位置选择方法)
‘sample’ 从X中随机选取K个质心点
‘uniform’ 根据X的分布范围均匀的随机生成K个质心
‘cluster’ 初始聚类阶段随机选择10%的X的子样本(此方法初始使用’sample’方法)
matrix 提供一K*P的矩阵,作为初始质心位置集合 - ‘Replicates’(聚类重复次数)
整数
%第一类数据
mu1=[0 0 0]; %均值
S1=[0.3 0 0;0 0.35 0;0 0 0.3]; %协方差
data1=mvnrnd(mu1,S1,100); %产生高斯分布数据
%%第二类数据
mu2=[1.25 1.25 1.25];
S2=[0.3 0 0;0 0.35 0;0 0 0.3];
data2=mvnrnd(mu2,S2,100);
%第三个类数据
mu3=[-1.25 1.25 -1.25];
S3=[0.3 0 0;0 0.35 0;0 0 0.3];
data3=mvnrnd(mu3,S3,100);
%显示数据
figure, hold on;
plot3(data1(:,1),data1(:,2),data1(:,3),'b+');
hold on;
plot3(data2(:,1),data2(:,2),data2(:,3),'r+');
plot3(data3(:,1),data3(:,2),data3(:,3),'g+');
grid on;
%三类数据合成一个不带标号的数据类
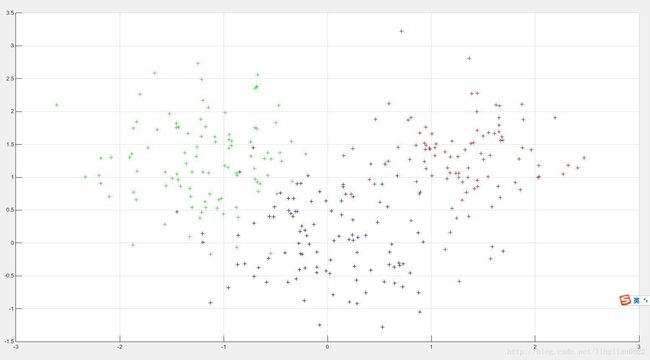
data=[data1;data2;data3]; %这里的data是不带标号的,即没有分好类
k = 3;
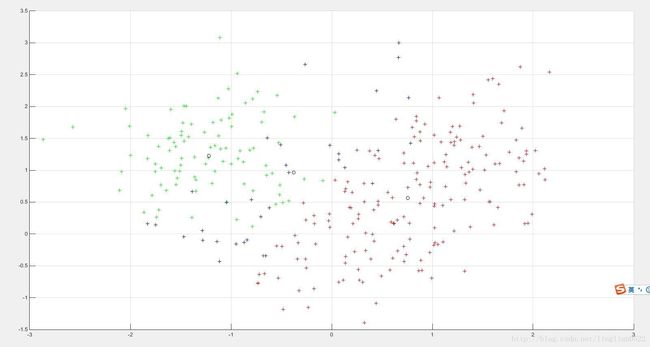
[idx, c] = kmeans(data, k); %分成3份
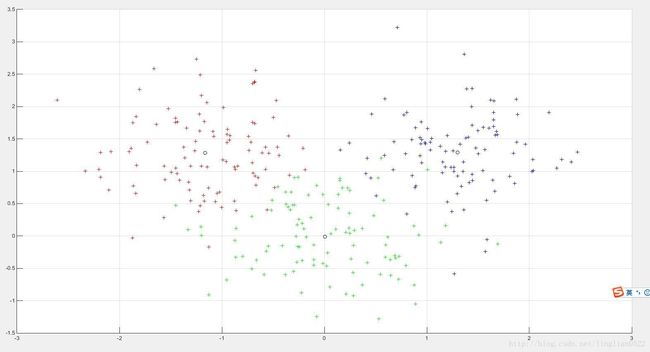
figure, hold on;
for i=1:300
if idx(i) == 1
plot3(data(i,1),data(i,2),data(i,3), 'b+');
elseif idx(i) == 2
plot3(data(i,1),data(i,2),data(i,3), 'r+');
else
plot3(data(i,1),data(i,2),data(i,3), 'g+');
end
end
for i=1:k
plot3(c(i, 1), c(i, 2), c(i, 3), 'ko'); % 画出中心点
end
grid on;图中原点就是簇的中心。
学习向量量化(LVQ)
与k均值算法类似,LVQ也是基于原型的聚类,但与一般的不同,LVQ假设数据样本带有类别标记,学习过程利用样本的这些监督信息来辅助聚类。
这个是用来建立神经网络的- -。
算法描述
![]()
newlvq(P,S1,PC,LR,LF) 建立LVQ向量化神经网络
P为一个Rx2维的网络输入向量取值范围的矩阵[Pmin Pmax];
Sl表示隐含层神经元的数目;
PC表示在第二层的权值中列所属类别的百分比;
LR表示学习速率,默认值为0.01;
Lf表示学习函数,默认值为learnlv1。
P = [-3 -2 -2 0 0 0 0 +2 +2 +3; ...
0 +1 -1 +2 +1 -1 -2 +1 -1 0];
Tc = [1 1 1 2 2 2 2 1 1 1];
T = ind2vec(Tc);
net = newlvq(P,4,[.6 .4]);
net = train(net,P,T);高斯混合聚类(EM算法)
利用概率模型来表达聚类原型。
这个算法会算法每个样本可能在每个簇的概率。
%第一类数据
mu1=[0 0 0]; %均值
S1=[0.3 0 0;0 0.35 0;0 0 0.3]; %协方差
data1=mvnrnd(mu1,S1,100); %产生高斯分布数据
%%第二类数据
mu2=[1.25 1.25 1.25];
S2=[0.3 0 0;0 0.35 0;0 0 0.3];
data2=mvnrnd(mu2,S2,100);
%第三个类数据
mu3=[-1.25 1.25 -1.25];
S3=[0.3 0 0;0 0.35 0;0 0 0.3];
data3=mvnrnd(mu3,S3,100);
%显示数据
figure, hold on;
plot3(data1(:,1),data1(:,2),data1(:,3),'b+');
hold on;
plot3(data2(:,1),data2(:,2),data2(:,3),'r+');
plot3(data3(:,1),data3(:,2),data3(:,3),'g+');
grid on;
%三类数据合成一个不带标号的数据类
data=[data1;data2;data3]; %这里的data是不带标号的,即没有分好类
k = 3;
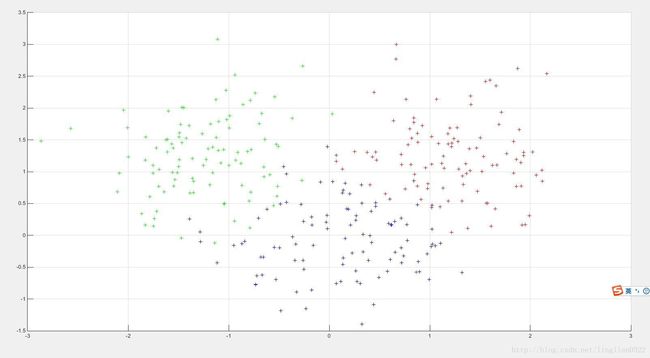
[px, model] = gmm(data, k); %分成3份
figure, hold on;
for i=1:300
[m, idx] = max(px(i, :));
if idx == 1
plot3(data(i,1),data(i,2),data(i,3), 'b+');
elseif idx == 2
plot3(data(i,1),data(i,2),data(i,3), 'r+');
else
plot3(data(i,1),data(i,2),data(i,3), 'g+');
end
end
c = model.Miu;
for i=1:k
plot3(c(i, 1), c(i, 2), c(i, 3), 'ko'); % 画出中心点
end
grid on;密度聚类(基于密度的聚类)
此类算法假设聚类结构能通过样本分布的紧密程度确定。
原理
从样本密度的角度来考察样本之间的可连接性,并基于可连接样本不断扩展聚类簇以获得最终的聚类结果。
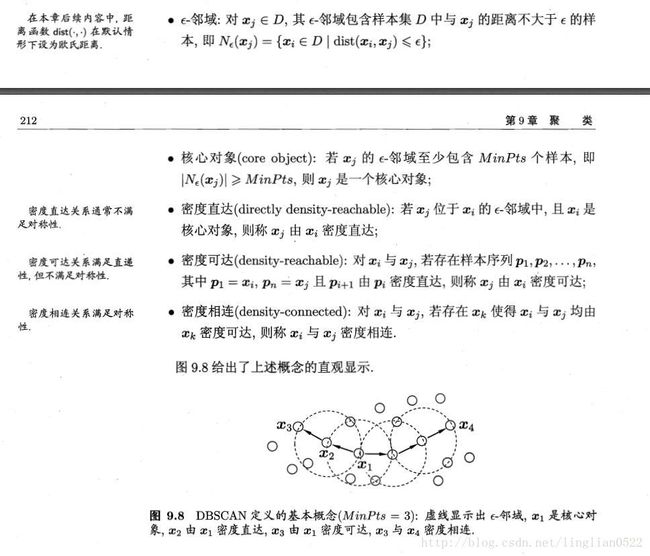
DBSCAN
层次聚类
层次聚类视图在不同层次对数据集进行划分,从而形成属性的聚类结构。
数据集的划分可以采用自底向上的聚合策略,也可采用自顶向下的分拆策略。
AGNES
clusterdata(X, cutoff)
X = [gallery('uniformdata',[10 3],12);...
gallery('uniformdata',[10 3],13)+1.2;...
gallery('uniformdata',[10 3],14)+2.5];
subplot(1, 2, 1), plot3(X(:, 1), X(:, 2), X(:, 3), '*');
grid on;
T = clusterdata(X,'maxclust',3);
subplot(1, 2, 2), scatter3(X(:,1),X(:,2),X(:,3),100,T,'filled');