笔记︱多种常见聚类模型以及分群质量评估(聚类注意事项、使用技巧)
聚类分析在客户细分中极为重要。有三类比较常见的聚类模型,K-mean聚类、层次(系统)聚类、最大期望EM算法。在聚类模型建立过程中,一个比较关键的问题是如何评价聚类结果如何,会用一些指标来评价。
在后面我补充以下两个应用:
应用一:如果Kmeans出现超级大群,分群数据两极分化的时候,如何解决?
应用二:R语言利用caret包进行KNN聚类
本篇笔记来源于CDA-DSC,L2-R语言课程,感谢老师上课的辛勤。
—————————————————————————————————————————————
一、聚类分析的距离问题
聚类分析的目的就是让类群内观测的距离最近,同时不同群体之间的距离最大。
1、样本聚类距离
一般来说,聚类分析的数据都会进行标准化,标准化是因为聚类数据会受数据的量纲影响。
在以上的几个距离明氏距离受量纲影响较大。马氏距离受量纲影响较小,还有cos(余弦相似性)余弦值的范围在[-1,1]之间,值越趋近于1,代表两个向量的方向越趋近于0,他们的方向更加一致。相应的相似度也越高(cos距离可以用在文本挖掘,文本词向量距离之上)。
几种标准化的方法,有规范化、标准化(R语言︱数据规范化、归一化)
2、群体聚类距离
前面是样本之间的距离,如果是一个点集,群落,如何定义群体距离。一般有以下几种距离。
![]()
—————————————————————————————————————————————
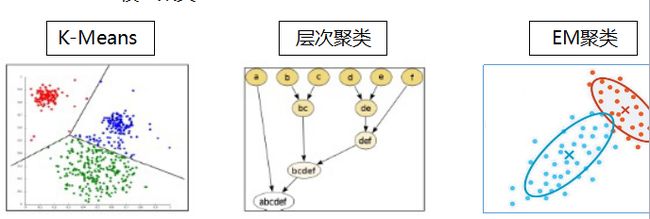
二、三个常见的聚类模型
有三类比较常见的聚类模型,K-mean聚类、层次(系统)聚类、最大期望EM算法。
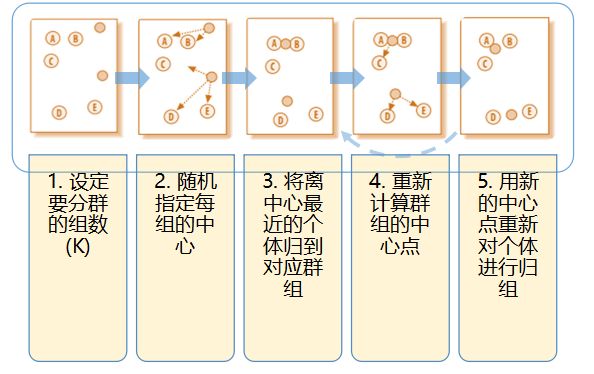
1、K-mean聚类
K-Means 聚类(MacQueen, 1967)是以样本间距离为基础,将所有的观测之间划分到K个群体,使得群体和群体之间的距离尽量大,同时群体内部的观测之间的“距离和”最小。
K均值是期望最大化算法的特殊情况,K均值是在每次迭代中只计算聚类分布的质心。
R语言中kmeans函数,输出结果的指标都是:
"cluster"是一个整数向量,用于表示记录所属的聚类
"centers"是一个矩阵,表示每聚类中各个变量的中心点
"totss"表示所生成聚类的总体距离平方和
"withinss"表示各个聚类组内的距离平方和
"tot.withinss"表示聚类组内的距离平方和总量
"betweenss"表示聚类组间的聚类平方和总量
"size"表示每个聚类组中成员的数量
(1)这四种条件都可能成为K均值聚类的终止条件:
这个条件限制了聚类算法的运行时间,但是在一些情况下,由于迭代次数不足,聚类的质量会很差。
在局部最小值不是特别差的情况下,会产生良好的聚类,但是运行时间可能相当长。
这种条件要确保算法已经收敛在最小值以内。
在 RRS 下降到阈值以下时终止,可以确保之后聚类的质量。实际上,这是一个很好的做法,在结合迭代次数的同时保证了K均值的终止。
(2)K-均值最害怕什么?
K均值聚类算法对离群值最敏感,因为它使用集群数据点的平均值来查找集群的中心。
在数据包含异常值、数据点在数据空间上的密度扩展具有差异、数据点为非凹形状的情况下,K均值聚类算法的运行结果不佳。
(3)多次kmeans会不会有更好的结果?
K均值聚类算法通常会对局部最小值进行转换,个别时候这个局部最小值也是全局最小值,但这种情况比较少。因此,更建议在绘制集群的推断之前,多次运行K均值算法。
然而,每次运行K均值时设置相同的种子值是有可能得出相同的聚类结果的,但是这样做只是通过对每次的运行设置相同的随机值来进行简单的算法选择。
K均值对簇中心初始化非常敏感。而且,初始化不良会降低收敛的速度差并会使得整体聚集效果不佳。
用于K均值初始化的方法是 Forgy 和随机分区。Forgy 方法从数据集中随机选择k个观测值,并将其作为初始值。随机分区方法是先随机为每个观测值分配一个簇,随后进行更新,簇的随机分配点的质心就是计算后得到的初始平均值。
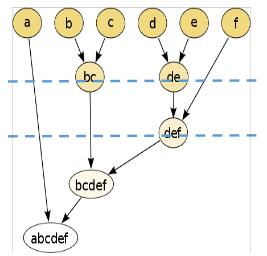
2、层次聚类——系统聚类
层次聚类也称系统聚类法,是根据个体间距离将个体向上两两聚合,再将聚合的小群体两两聚合一直到聚为一个整体。计算所有个体之间的距离,最相近距离的个体合体,不断合体。
对于层次聚类的的群平均值,两个簇的接近度指的是不同集群中的每一对点对的近似值的平均值。这是最大值和最小值方法之间的中间方法。
(1)Ward 方法的接近函数
Ward 方法是一种质心算法。质心方法通过计算集群的质心之间的距离来计算两个簇的接近度。对于 Ward 方法来说,两个簇的接近度指的是当两个簇合并时产生的平方误差的增量。在6%的样本数据集中,使用 Ward 方法产生的结果和使用最大值、最小值、组平均值的聚类结果会有所不同。
3、EM最大期望模型
最大期望(EM)算法是在概率模型中寻找参数最大似然估计或者最大后验估计的算法,其中概率模型依赖于无法观测的隐藏变量(Latent Variable)。K-means可以看作是EM模型聚类的一个特例。
A、设定要分成的类的数目K以及每一个类的初始概率分布
B、E步:对每一个个体计算其属于每一类的后验概率
C、M步:基于极大似然估计更新每个类的概率分布函数
D、迭代执行以上E步和M步直至似然函数收敛
E、基于最大概率值确定每个个体所属的类
在聚类中使用期望最大化算法,本质是将数据点按照所选数量的簇进行分类,这个数量和预期生成的不同分布的数量是相同的,而且分布也必须是相同的类型。
K均值是期望最大化算法的特殊情况,K均值是在每次迭代中只计算聚类分布的质心。
4、常见聚类模型的比较
|
K-means(kmeans) |
层次聚类(kmeans) |
EM模型聚类(mclust包) |
优点 |
属于快速聚类,计算效率高 |
1、能够展现数据层次结构,易于理解 2、可以基于层次事后再选择类的个数(根据数据选择类,但是数据量大,速度慢) |
相比其他方法能够拟合多种形状的类 |
缺点 |
1、需要实现指定类的个数(需要指定类) 2、有时会不稳定,陷入局部收敛 |
1、计算量比较大,不适合样本量大的情形 2、较多用于宏观综合评价 |
需要事先指定类的个数和初始分布 |
—————————————————————————————————————————————
三、实际案例中注意点
1、聚类指标的选取
用于聚类的指标对于聚类结果的影响很大。
2、指标的预处理
量纲不一致会导致距离计算的偏差,聚类数据对量纲要求比较高,选择不同的距离一般需要对数据先进行标准化。然可以根据实际情况,重要的变量对比不需要做量纲。
特征缩放保证了在聚类分析中每一个特征都有同样的权重。想象这样一个例子,对体重范围在55-100(kg)和身高在5.6到6.4(英寸)的人进行聚类分析。因为体重的范围远远高于身高的范围,如果不进行缩放,产生的簇会对结果产生误导。因此,使它们具有相同的级别就显得很有必要了,只有这样才能保证聚类结果权重相同。
3、聚类分群的数量如何确定?分群效果如何?
没有固定标准,一般会3-10分群。或者用一些指标评价,然后交叉验证不同群的分群指标。
一般的指标:轮廓系数silhouette(-1,1之间,值越大,聚类效果越好)(fpc包),兰德指数rand;R语言中有一个包用30种方法来评价不同类的方法(NbClust),但是速度较慢
商业上的指标:分群结果的覆盖率;分群结果的稳定性;分群结果是否从商业上易于理解和执行
轮廓系数旨在将某个对象与自己的簇的相似程度和与其他簇的相似程度进行比较。轮廓系数最高的簇的数量表示簇的数量的最佳选择。
![]()
一般来说,平均轮廓系数越高,聚类的质量也相对较好。在这,对于研究区域的网格单元,最优聚类数应该是2,这时平均轮廓系数的值最高。但是,聚类结果(k=2)的 SSE 值太大了。当 k=6 时,SEE 的值会低很多,但此时平均轮廓系数的值非常高,仅仅比 k=2 时的值低一点。因此,k=6 是最佳的选择。
4、聚类算法如何进行特征提取?
将集群的 id 设置为虚拟变量和将集群的质心设置为连续变量,这两项可能不会为多维数据的回归模型提供更多的相关信息。但是当在一个维度上进行聚类分析时,上面给出的所有方法都有望为多维数据的回归模型提供有意义的信息。
举个例子,根据头发的长度将人们分成两组,将聚类类别存储为虚拟变量,将聚类质心存储为连续变量,这样一来,多维数据的回归模型将会得到有用的信息。
5、聚类会受异方差、多重共线性的影响吗?
聚类分析不会受到异方差性的负面影响,但是聚类中使用的特征/变量多重共线性会对结果有负面的影响,因为相关的特征/变量会在距离计算中占据很高的权重。
——————————————————————————————————————————
应用一:kmeans时候出现的超级大群现象,如何解决?
kmeans做聚类的时候,往往会出现一个超级大群,一类样本数据很多很多,其他类别数量很少。两极分化很严重。在实际使用的时候会出现以下这几个问题:
1、有序变量的聚类问题
有序变量,譬如天气类型:阴天=0,晴天=1,下雨=2等,这样类型的变量,不适合取均值,因为均值是没有意义的。同时kmeans是根据空间关系来定义的,所以0-1与0-2,很显然是0-1距离近,这一特性会引起最终分类时候出现歧义。
但是一些数值很大的指标,就需要通过标准化来消除量纲。
参考AlgorithmDog 公众号的一些内容:在归一化处理后,只取值0/1的特征就会变成强特征,对聚类有很大的影响。一个直观的理解,假设只有两维特征,其中一个取值0/1。那么归一化处理后,样本的分布就会位于两条线段上,对这些数据进行聚类的话,如果初始点分布在两侧,那么两条线段的数据会被分开,只会在两条线段上分别进行聚类。位于不同线段上的两个点,它们之间的距离大于等于1,大于线段内两两点之间的距离。
同理在三维中,如果有个特征只取值0/1,那么数据是分布在两个正方形面上的,聚类也很可能在每个面内单独进行。所以如果采用one-hot编码,那些0/1特征在很大程度上就决定了聚类结果,其他特征的重要性变很小了。但有时这种枚举型数据又很重要,可以很好地用来刻画实例,那么我们的做法是在聚完类后,将聚类结果和枚举数据做交叉分析。看在每类中,枚举值的分布情况。
2、超级大群/长尾特征
参考AlgorithmDog 公众号的内容:
80%的数据分布在1%的空间内,而剩下的20%的数据分布在99%的空间内。聚类时,分布在1%空间内的大部分数据会被聚为一类,剩下的聚为一类。当不断增加K值时,模型一般是对99%空间内的数据不断进行细分,因为这些数据之间的空间距离比较大。
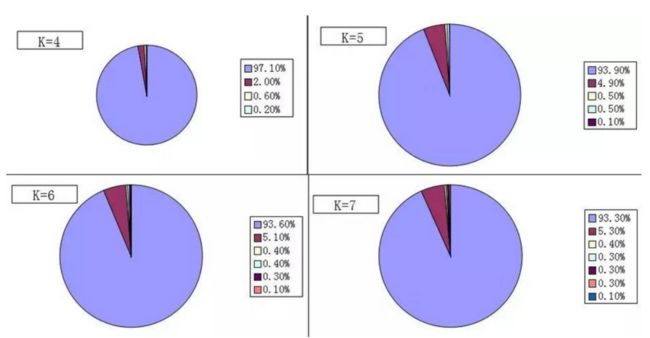
而对分布在1%空间内的数据则很难进一步细分,或者即使细分了,也只是剥离出了外侧少量数据。下图是我们在某个项目中的聚类结果,可以看到有一类用户占了90%以上,而且随着K的增加,这类用户里只有很小一部分数据会被划分出来。
解决办法:那么为了解决这个问题,一种可行的方法是是对特征取LOG,减轻长尾问题。经过这两种方法处理后,都能较好的对玩家进行分类。下图是上图中的数据点取LOG后得到的分布图。
缺点:取LOG的方法的缺点在于,会使数据变得不直观,不好理解。
————————————————————————————————————
应用二:R语言使用caret包进行KNN聚类
本应用来自于经管之家论坛的一位坛友:http://bbs.pinggu.org/thread-4938780-1-1.html
# Classifying using the K-Nearest Neighbors (KNN) approach
#----------------------------------------------------------------
library(class)
library(caret)
vac <- read.csv("vacation-trip-classification.csv")
vac$Income.z <- scale(vac$Income)
vac$Family_size.z <- scale(vac$Family_size)
set.seed(1000)
train.idx <- createDataPartition(vac$Result, p = 0.5, list = FALSE)
train <- vac[train.idx, ]
temp <- vac[-train.idx, ]
val.idx <- createDataPartition(temp$Result, p = 0.5, list = FALSE)
val <- temp[val.idx, ]
test <- temp[-val.idx, ]
pred1 <- knn(train[,4:5], val[,4:5], train[,3], 1)
errmat1 = table(val$Result, pred1, dnn = c("Actual", "Predicted"))
pred.test <- knn(train[,4:5], test[,4:5], train[,3], 1)
errmat.test = table(test$Result, pred.test, dnn = c("Actual", "Predicted"))
knn.automate <- function (trg_predictors, val_predictors, trg_target, val_target, start_k, end_k)
{
for (k in start_k:end_k) {
pred <- knn(trg_predictors, val_predictors,
trg_target, k)
tab <- table(val_target, pred, dnn = c("Actual", "Predicted"))
cat(paste("Error matrix for k=", k,"\n"))
cat("==========================\n")
print(tab)
cat("--------------------------\n\n\n")
}
}
knn.automate(train[,4:5], val[,4:5], train[,3], val[,3], 1,7)
pred5 <- knn(train[4:5], val[,4:5], train[,3], 5, prob=TRUE)
pred5应用三:R语言动态聚类DBSCAN
动态聚类往往聚出来的类有点圆形或者椭圆形。基于密度扫描的算法能够解决这个问题。思路就是定一个距离半径,定最少有多少个点,然后把可以到达的点都连起来,判定为同类。在r中的实现
dbscan(data, eps, MinPts, scale, method, seeds, showplot, countmode)其中eps是距离的半径,minpts是最少多少个点。 scale是否标准化(我猜) ,method 有三个值raw,dist,hybird,分别表示,数据是原始数据避免计算距离矩阵,数据就是距离矩阵,数据是原始数据但计算部分距离矩阵。showplot画不画图,0不画,1和2都画。countmode,可以填个向量,用来显示计算进度。用鸢尾花试一试
> install.packages("fpc", dependencies=T)
> library(fpc)
> newiris <- iris[1:4]
> model <- dbscan(newiris,1.5,5,scale=T,showplot=T,method="raw")# 画出来明显不对 把距离调小了一点
> model <- dbscan(newiris,0.5,5,scale=T,showplot=T,method="raw")
> model #还是不太理想……
dbscan Pts=150 MinPts=5 eps=0.5
0 1 2
border 34 5 18
seed 0 40 53
total 34 45 71从实践的效果来看,层次聚类一运行,大数据集因为内存问题就爆,动态聚类倒是可以运行,但是呢!!
消耗CPU较大,运行地起来,就是慢...