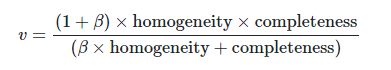聚类:KMeans、DBSCAN、层次聚类AgglomerativeClustering及聚类评价指标
聚类(无监督学习)
目标
簇内相似度高,簇间相似度低
聚类性能度量
- 外部指标:将聚类结果与某个“参考模型”进行比较。
- 内部指标:直接考察聚类结果,不利用任何参考模型。
聚类方法
一、KMeans
给定k个聚类的数量固定,将观察值分配给这些聚类,以使各个聚类(对于所有变量)的均值尽可能彼此不同。
KMeans(n_clusters = 8,*,init ='k-means ++',n_init = 10,max_iter = 300,tol = 0.0001,precompute_distances ='deprecated',verbose = 0,random_state = None,copy_x = True,n_jobs ='deprecated ',algorithm ='auto' )参数解释
二、层次聚类
AgglomerativeClustering(n_clusters=2, *, affinity='euclidean', memory=None, connectivity=None, compute_full_tree='auto', linkage='ward', distance_threshold=None)参数解释
n_clusters——聚类的个数
linkage——连接方法。ward(方差最小化)complete(两组中所有观测值间的最大距离)average(平均距离)single(最小距离)
affinity——用于计算linkage的度量。“euclidean”:欧几里得距离(如果linkage 是 “ward”, 则affinity只可选为此选项) ,“l1”,“l2”
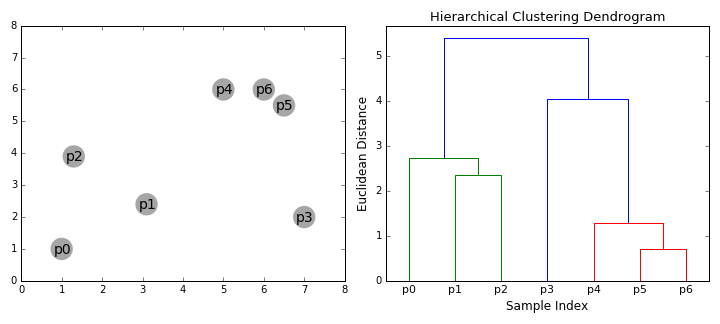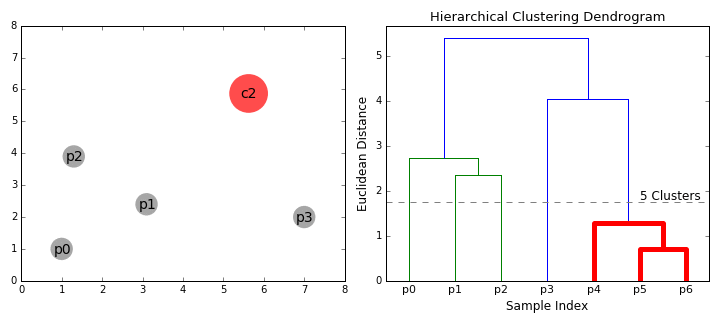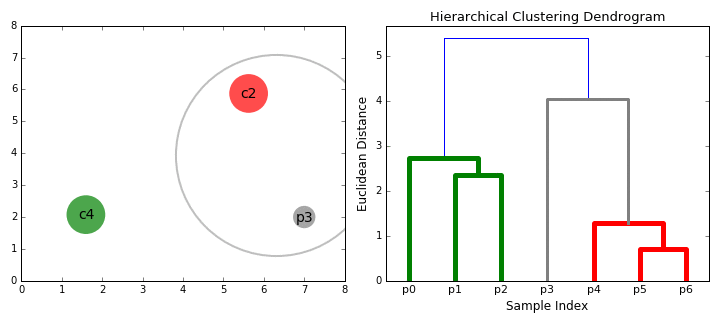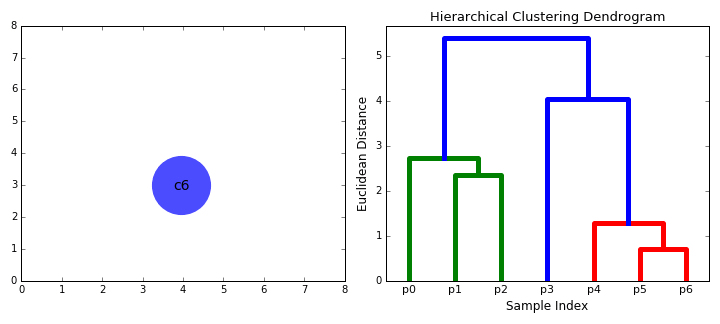
1)先计算样本之间的距离。每次将距离最近的点合并到同一个类。然后,再计算类与类之间的距离,将距离最近的类合并为一个大类。不停的合并,直到合成了一个类。其中类与类的距离的计算方法有:最短距离法,最长距离法,中间距离法,类平均法等。比如最短距离法,将类与类的距离定义为类与类之间样本的最短距离。
2)层次聚类算法根据层次分解的顺序分为:自下底向上和自上向下,即凝聚的层次聚类算法和分裂的层次聚类算法(agglomerative和divisive),也可以理解为自下而上法(bottom-up)和自上而下法(top-down)。自下而上法就是一开始每个个体(object)都是一个类,然后根据linkage寻找同类,最后形成一个“类”。自上而下法就是反过来,一开始所有个体都属于一个“类”,然后根据linkage排除异己,最后每个个体都成为一个“类”。这两种路方法没有孰优孰劣之分,只是在实际应用的时候要根据数据特点以及你想要的“类”的个数,来考虑是自上而下更快还是自下而上更快。至于根据Linkage判断“类”的方法就是最短距离法、最长距离法、中间距离法、类平均法等等(其中类平均法往往被认为是最常用也最好用的方法,一方面因为其良好的单调性,另一方面因为其空间扩张/浓缩的程度适中)。为弥补分解与合并的不足,层次合并经常要与其它聚类方法相结合,如循环定位。
AGNES是一种采用自底向上聚合策略的层次聚类算法。AGNES算法也被称为“单链接”(single)、“全链接”(complete)、“均链接”(average)算法。
三、DBSCAN
根据半径eps和半径内的最小样本数min_samples,以半径内的每一个样本为原点,不停“生长”,当该样本半径内的最小样本数小于min_samples时,停止“生长”。
DBSCAN(eps=0.5, *, min_samples=5, metric='euclidean', metric_params=None, algorithm='auto', leaf_size=30, p=None, n_jobs=None)~参数解释
eps——半径
min_samples——半径内的最小样本数

聚类评价指标(有真实标签)
调整后的rand指数——adjusted Rand index
有界范围[-1,1]:负值为分类效果不好(独立标签),相似的聚类具有正的ARI,1.0是完美匹配分数;对于均匀分布的数据,ARI接近于0。
from sklearn import metrics
labels_true = [0, 0, 0, 1, 1, 1]
labels_pred = [0, 0, 1, 1, 2, 2]
metrics.adjusted_rand_score(labels_true, labels_pred)
0.24...adjusted_rand_score是对称的:交换参数不会更改分数
在预测的标签中置换0和1,将2重命名为3,可以获得相同的分数:
labels_pred = [1, 1, 0, 0, 3, 3]
metrics.adjusted_rand_score(labels_true, labels_pred)
0.24...~调整后的互信息(AMI)——Adjusted Mutual Information
互信息用来衡量两个分布一致性,并且忽略顺序。直观上,互信息度量 X 和 Y 共享的信息:它度量知道这两个变量其中一个,对另一个不确定度减少的程度。例如,如果 X 和 Y 相互独立,则知道 X 不对 Y 提供任何信息,反之亦然,所以它们的互信息为零。
随机的预测AMI会接近于0;最大上界为1,说明预测完全正确。
from sklearn import metrics
labels_true = [0, 0, 0, 1, 1, 1]
labels_pred = [0, 0, 1, 1, 2, 2]
metrics.adjusted_mutual_info_score(labels_true, labels_pred)
0.22504...~规范化互信息(NMI)——Normalized Mutual Information
mutual_info_score,adjusted_mutual_info_score和 normalized_mutual_info_score是对称的:交换参数不改变分数
homogeneity_score、completeness_score
同质性 homogeneity:每个聚类仅包含单个类的成员。
完整性 completeness:给定类的所有成员都分配给同一集群。
V-度量是以上两者的调和平均数。
评分范围:0—1(越高越好)
from sklearn import metrics
labels_true = [0, 0, 0, 1, 1, 1]
labels_pred = [0, 0, 1, 1, 2, 2]
metrics.homogeneity_score(labels_true, labels_pred)
0.66...
metrics.completeness_score(labels_true, labels_pred)
0.42...~它们的谐波均值称为V-measure由v_measure_score以下公式计算
metrics.v_measure_score(labels_true, labels_pred)
0.51...~metrics.v_measure_score(labels_true, labels_pred, beta=0.6)
0.54...~重量更大将归因于同质性,并且使用大于1的值:
metrics.v_measure_score(labels_true, labels_pred, beta=1.8)
0.48...~可以用同质性和完整性 来定性分析具有不良V度量的聚类, 以更好地感觉到分配所造成的“错误”类型。
V度量实际上等效于上面讨论的互信息(NMI),聚合函数是算术平均值。
齐次性,完整性和V度量可使用homogeneity_completeness_v_measure计算
metrics.homogeneity_completeness_v_measure(labels_true, labels_pred)
(0.66..., 0.42..., 0.51...)~优点:0代表差,1代表好;不需要对簇结构进行假设,可以对不同聚类算法进行比较。
缺点:当簇数目比较大时,这三者的值都不会趋近于0;当样本数多于1000或者簇数目少于10时可以避免这个问题,其他情况可以选择适用ARI;另外这三个指标需要知道样本的真实分类。
Fowlkes-Mallows scores
Fowlkes-Mallows分数FMI定义为成对精度和召回率的几何平均值:

其中TP,“正阳性”的数量(即在真标签和预测标签中属于同一聚类的点对的数量)
FP是“ 假阳性”的数目(即属于“ 正标签”和“预测标签” 的点对的数量)在真正的标签,而不是在所预测的标签相同的簇)
和FN是的数量假阴性(即是,在所预测的标签和未在真标记)相同的簇所属的一对点的数目。
分数范围是0到1。较高的值表示两个聚类之间的相似性良好。
优点:随机预测值趋近于0;上限为1,表示预测完全正确;不需要对簇类型进行假设。
缺点:需要知道样本类别。
聚类评价指标(无真实标签)
轮廓系数—Silhouette Coefficient
每个样本都有各自的轮廓系数,该系数由两个分数组成:
a:样本与同一类别中所有其他点之间的平均距离。
b:样本与下一个最近的簇中所有其他点之间的平均距离。
单个样本的Silhouette系数s为:

注意:仅当标签数为2 <= n_labels <= n_samples-1时,才定义轮廓系数。
即聚类的类别大于1,且小于样本数。
此函数返回所有样本的平均轮廓系数。
最佳值为1,最差值为-1。接近0的值表示重叠的群集。负值通常表示样本已分配给错误的聚类,因为不同的聚类更为相似。
from sklearn import metrics
from sklearn.cluster import KMeans
kmeans_model = KMeans(n_clusters=3, random_state=1).fit(X)
labels = kmeans_model.labels_
metrics.silhouette_score(X, labels, metric='euclidean')
0.55...优点:不需要知道样本标签;-1表示存在错误聚类,0表示有交叉的簇,1表示高密度聚类。
缺点:凸形簇的轮廓系数通常高于其他类型的簇。
Calinski-Harabasz Index
该指数是所有集群的集群间离散度和集群间离散度之和的比率(其中,离散度定义为距离平方的总和),也称为方差比标准。
Calinski-Harabaz的分数S被定义为组间离散与组内离散的比率,该分值越大说明聚类效果越好。

BK是组间离散矩阵
WK是组内离散矩阵

n是样本点数,cq 是在聚类q中的样本点,Cq 是聚类q的中心点,nq 是聚类q中的样本点数量,c是E的中心。
计算Calinski和Harabasz得分。也称为差异比率标准。得分定义为簇内分散体和簇间分散体之间的比率。
from sklearn import metrics
from sklearn.cluster import KMeans
kmeans_model = KMeans(n_clusters=3, random_state=1).fit(X)
labels = kmeans_model.labels_
metrics.calinski_harabasz_score(X, labels)
561.62...优点:易计算(比轮廓系数快很多);当类聚合地很好,类间差距很大时,该值增大,反映了簇的标准概念。
缺点:对于凸形簇,该值会很大,但是对于其他类型簇,如基于密度型,该值无可奈何。
Davies-Bouldin Index
分数定义为每个群集与其最相似群集的平均相似性度量,其中相似度是群集内距离与群集间距离的比率。因此,距离更远且分散程度较低的群集将获得更好的分数。
from sklearn.cluster import KMeans
from sklearn.metrics import davies_bouldin_score
kmeans = KMeans(n_clusters=3, random_state=1).fit(X)
labels = kmeans.labels_
davies_bouldin_score(X, labels)
0.6619...缺点:对于凸性簇,该值会高一点;质心距离限制距离度量欧氏空间;良好的值并不意味着最好的分类结果。
Contingency Matrix—权变矩阵
报告每个真实/预测的聚类对的相交基数。权变矩阵为所有聚类指标提供了足够的统计数据,在这些度量中样本是独立且分布均匀的,不需要考虑某些未聚类的实例。
from sklearn.metrics.cluster import contingency_matrix
>>> x = ["a", "a", "a", "b", "b", "b"]
>>> y = [0, 0, 1, 1, 2, 2]
>>> contingency_matrix(x, y)
array([[2, 1, 0],
[0, 1, 2]])输出数组的第一行指示存在三个样本,其真实簇为“ a”。其中两个在预测聚类0中,一个在1中,一个都不在2中。第二行表示存在三个样本,其真实聚类为“ b”。其中,没有一个在预测聚类0中,一个在1中,两个在2中。
缺点:1.列联矩阵对于少数群集很容易解释,但对于大量群集则很难解释。
2.它没有给出一个单一的指标来用作集群优化的目标。