“Super-enhancer神器“ROSE安装及教程
好像又好久好久没更新了。这周师兄给我的任务就是找super enhancer。那最出名的工具就是Richard A.Young实验室开发的ROSE:RANK ORDERING OF SUPER ENHANCERS了。我花了2天不到的时间大概摸清了这个工具如何使用。鉴于当时google的资料太少,所以决定结合大神的资料自己也写一下。
一、安装
ROSE依赖软件有:Python 2.7.3, R 2.15.3, 和 SAMtools 0.1.18,因此在安装ROSE前,首先确保服务器上安装了这三个工具
wget https://bitbucket.org/young_computation/rose/get/1a9bb86b5464.zip
unzip young_computation-rose-1a9bb86b5464.zip解压后文件见下图,可以直接调用python *.py工具
二、使用
其中最重要的一个就是ROSE_main.py,用于寻找增强子。
python ROSE_main.py -g GENOME_BUILD -i INPUT_CONSTITUENT_GFF -r RANKING_BAM -o OUTPUT_DIRECTORY
[optional: -s STITCHING_DISTANCE -t TSS_EXCLUSION_ZONE_SIZE -c CONTROL_BAM]
# 参数解释
-g hg18、hg19、mm8、mm9或mm10,用于构建UCSC参考基因组
-i 输入gff文件,一般为使用MACS鉴定得到的富集区域
-r 排序后的bam文件,同时需为bam添加index
-o 输出文件目录
#可选参数
-s STITCHING_DISTANCE,合并两个region的最大距离,默认值为12.5kb
-t TSS_EXCLUSION_ZONE_SIZE,排除TSS区域大小,排除与TSS前后某距离内的区域,以排除启动子偏差(默认值:0;推荐值:2500)。如果设置该值为0,将不会查找基因。
-c CONTROL_BAM,control样本的bam文件
#输入文件格式要求:
bam文件格式要求:需要排序和构建index(samtools可以操作),bam文件的染色体id需要以chr开头。
gff文件格式很重要,因为这个吃了亏,半天时间花在了找bug上,甚至去改源代码找bug(论学好python的重要性)。
gff文件格式要求:gff文件必须有以下列
1.染色体位置(chr#)
2.每个增强子区域的特定id
4.区域起始位置
5.区域终止位置
7.正负链信息(+, -, .)
ROSE也有转换bam为gff的工具,在运行ROSE_mian.py 时,会调用ROSE_bamToGFF.py。不过我没用,直接通过bed文件自己做的gff文件。
下面这个是重点重点,敲黑板!如果报错一定要仔细看这个文件夹!!!
附上一个例子文件夹,有示例输入文件和运行代码以及输出文件。由于bam文件有点大,就不上传了。尤其是gff文件列与列之间的tab键个数一定要数清楚,不然会犯我出现的错误。简单说下,文件列为:chrom,id,[blank],start,end,[blank],[.],[blank],id,具体见例子。
链接:https://pan.baidu.com/s/1YI-ZmYBJlt9DXIH5wf0W7Q
提取码:t0us
复制这段内容后打开百度网盘手机App,操作更方便哦
#ROSE_main.py运行实例
python ROSE_main.py -g hg19 -i ./data/H3K27ac.new.gff -r ./data/ENCFF063OFD.sort.bam -c ./data/control.sort.bam -o example -s 12500 -t 2500
#输出文件如下:
1.**OUTPUT_DIRECTORY/gff/*.gtf 该文件为输入gtf文件的副本;
2.**OUTPUT_DIRECTORY/gff/*STITCHED*.gtf 该文件为通过在STITCHING_DISTANCE将INPUT_CONSTITUENT_GFF拼接在一起创建的gff文件;文件列数如下:
chrom, name, [blank], start, end, [blank], [blank], strand, [blank], [blank], name
其中 name 字段的命名方式为:拼接起来的区域数+最左端区域ID。
3.**OUTPUT_DIRECTORY/mappedGFF/*_MAPPED.gff 每个bam文件通过bamToGFF的输出文件,包含以下列:
(成分ID,测试区域,平均读取密度(单位为每百万位元每百万映射的单位读数密度))
4.**OUTPUT_DIRECTORY/mappedGFF/* _STITCHED * _MAPPED.gff 每个bam文件通过bamToGFF的输出文件,该文件中对增强子区域进行了拼接,包含以下列:
(拼接增强子ID,测试区域,平均读取密度(单位为百万映射每单位拼接增强子数))
5.**OUTPUT_DIRECTORY/STITCHED_ENHANCER_REGION_MAP.txt bamToGFF计算后得到的拼接增强子密度文件,包含以下列:
(拼接增强子ID,染色体,拼接增强子起始位置,拼接增强子末端位置,拼接数,BAM信号等级,BAM信号)
6..**OUTPUT_DIRECTORY/*_AllEnhancers.table.txt 增强子列表,包含每个增强子的排名和是否为超级增强子,包含以下列:
(增强子ID,染色体,拼接增强子起始位点,拼接增强子末端,拼接数,拼接成分大小,BAM的信号,BAM的等级,是否为超增强子:是(1)否(0))
7.**OUTPUT_DIRECTORY/* _SuperEnhancers.table.txt 超级增强子的排名,为*_AllEnhancers.table.txt 文件的子集。包含以下列:
(拼接增强剂ID,染色体,拼接增强子起始位点,拼接增强子末端,拼接数,缝合在一起的成分的大小,RANKING_BAM的信号,RANKING_BAM的等级,超增强子的二进制(1)与典型(0))
8.**OUTPUT_DIRECTORY/*_Enhancers_withSuper.bed 可以加载到UCSC浏览器中可视化的增强子bed文件
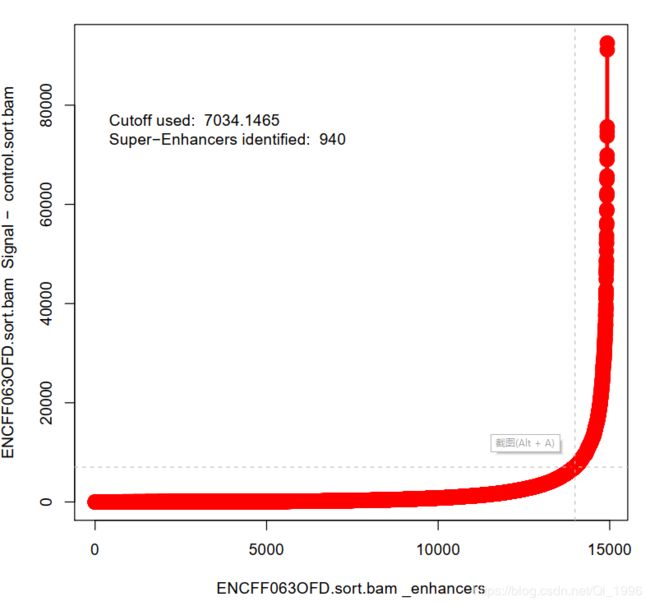
9.**OUTPUT_DIRECTORY/*_Plot_points.png 所有增强子散点图,如下图:
总而言之,对我来说有用的就是两个txt文件。还有一个ROSE_geneMapper.py 用法我没用过,所以复制粘贴另一个博主的内容:
Usage: ROSE_geneMapper.py [options] -g [GENOME] -i [INPUT_ENHANCER_FILE]
# 参数解释
-i INPUT 输入ROSE_mian.py生成的enhancer table文件
-g GENOME 输入genome信息(MM9,MM8,HG18,HG19等)
-o OUT 输出路径
# 可选参数
-l GENELIST 要过滤的基因列表
-w WINDOW 搜索基因距离,默认值为50,000bp
-f format 如果使用此参数,将保持原输入文件格式输出
ROSE_geneMapper.py 运行实例:
python $SOFT_PATH/ROSE_geneMapper.py -i $WORK_PATH/ROSE/TE7/TE7_peaks_AllEnhancers.table.txt \
-g HG38 -o $WORK_PATH/ROSE/TE7 2>$LOG_PATH/TE7_enhancer_anno.log
输出文件如下:
1.**OUTPUT_DIRECTORY/*ENHANCER_TO_GENE.txt enhancer重叠基因、附近基因以及最近的基因列表
2.**OUTPUT_DIRECTORY/*GENE_TO_ENHANCER.txt 以每个基因为列名的和其相关的增强子位置信息列表
得到这两个表格即可对基因进行筛选然后进行GO及KEGG分析等。
---------------------
作者:寂桐
来源:CSDN
原文:https://blog.csdn.net/oxygenjing/article/details/77862115
版权声明:本文为博主原创文章,转载请附上博文链接!
工具开发者网站:http://younglab.wi.mit.edu/super_enhancer_code.html
以上很多内容来自于上面那位大大,也多亏了实验室的指导说明。非常感谢他们辣!
下两个博客打算写两个这段时间一直用的工具GAT和SICER。