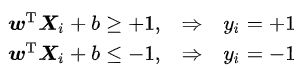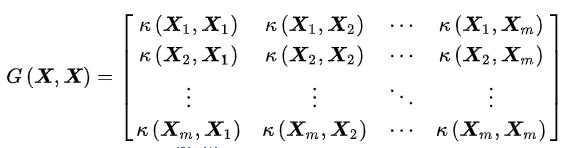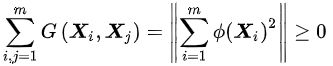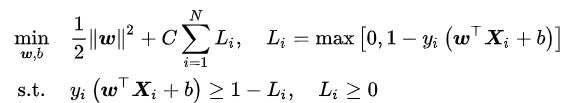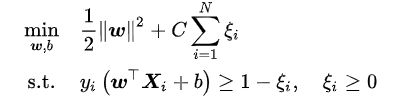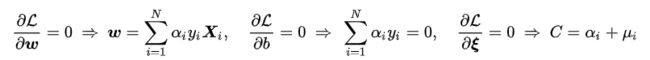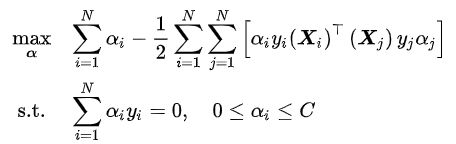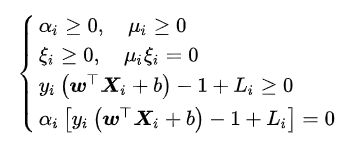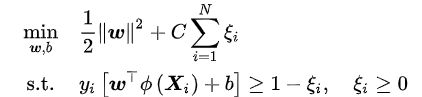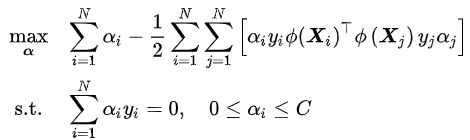支持向量机理论+简单代码实现
文章目录
- 一、支持向量机理论简介
- 1.线性分类
- 线性可分性
- 2.核方法
- Mercer定理
- 常见的核函数
- 3.算法
- 线性SVM
- (1)硬边距
- (2)软边距
- 非线性SVM
- 二、代码实现
- 模拟升维
- 超参数
- 三、参考文章
一、支持向量机理论简介
支持向量机是一类按监督学习方式对数据进行二元分类的广义线性分类器,其决策边界是对学习样本求解的最大边距超平面
1.线性分类
线性可分性
在分类问题中给定输入数据和学习目标:X={X1,X2,…,XN},y={y1,…,yN},其中输入数据的每个样本都包含多个特征并由此构成特征空间:X=[x1,x2,…,xn],而学习目标为二元变量
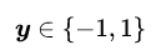
表示负类和正类
若输入数据所在的特征空间存在作为决策边界的超平面将学习目标按正类和负类分开,并使任意样本的点到平面距离大于等于1
则称该分类问题具有线性可分性,参数w,b分别为超平面的法向量和截距。
满足该条件的决策边界实际上构造了2个平行的超平面作为间隔边界以判别样本的分类:
所有在上间隔边界上方的样本属于正类,在下间隔边界下方的样本属于负类。两个间隔边界的距离d=2/||w||被定义为边距(margin),位于间隔边界上的正类和负类样本为支持向量。
2.核方法
一些线性不可分的问题可能是非线性可分的,即特征空间存在超曲面将正类和负类分开。使用非线性函数可以将非线性可分问题从原始的特征空间映射至更高维的希尔伯特空间,从而转化为线性可分问题,此时作为决策边界的超平面表示如下:
由于映射函数具有复杂的形式,难以计算其内积,因此可使用核方法,即定义映射函数的内积为核函数:
以回避内积的显式计算
Mercer定理
核函数的选择需要一定条件,函数
![]()
是核函数的充要条件是,对输入空间的任意向量:
![]()
其核矩阵,即如下形式的格拉姆矩阵
是半正定矩阵。上述结论被称为Mercer定理
作为充分条件:特征空间内两个函数的内积是一个二元函数,在其核矩阵为半正定矩阵时,该二元函数具有可再生性:
因此其内积空间是一个赋范向量空间,可以完备化得到希尔伯特空间 ,即再生核希尔伯特空间。作为必要条件,对核函数构造核矩阵后易知:
常见的核函数
3.算法
线性SVM
(1)硬边距
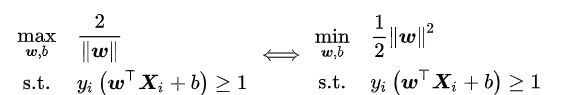
给定输入数据和学习目标:X={X1,X2,…,XN},y={y1,…,yN},硬边界SVM是在线性可分问题中求解最大边距超平面的算法,约束条件是样本点到决策边界的距离大于等于1。硬边界SVM可以转化为一个等价的二次凸优化问题进行求解:
我们注意到虽然超平面法向量w是唯一优化目标,但学习数据和超平面的截距通过约束条件影响了该优化问题的求解。硬边距SVM是正则化系数取0时的软边距SVM,下面我们来看一看软边距SVM。
(2)软边距
在线性不可分问题中使用硬边距SVM将产生分类误差,因此可在最大化边距的基础上引入损失函数构造新的优化问题。SVM使用铰链损失函数,沿用硬边界SVM的优化问题形式,软边距SVM的优化问题有如下表示:
上式表明可知,软边距SVM是一个L2正则化分类器,式中Li表示铰链损失函数。使用松弛变量:ξ≥0处理铰链损失函数的分段取值后,上式可化为:
求解上述软边距SVM通常利用其优化问题的对偶性
推导:
定义软边距SVM的优化问题为原问题,通过拉格朗日乘子:![]()
可得到其拉格朗日函数:
令拉格朗日函数对优化目标w,b,ξ的偏导数为0,可得到一系列包含拉格朗日乘子的表达式:
对偶问题的约束条件中包含不等关系,因此其存在局部最优的条件是拉格朗日乘子满足条件:
由上述KKT条件可知,对任意样本(Xi,yi),总有αi=0或yi(w^TXi+b)=1-ξi ,对前者,该样本不会对决策边界wTXi+b=0产生影响,对后者,该样本满足yi(wTXi+b)=1-ξi意味其处于间隔边界上(αi< C)、间隔内部(αi=C)或被错误分类(αi>C),即该样本是支持向量。由此可见,软边距SVM决策边界的确定仅与支持向量有关,使用铰链损失函数使得SVM具有稀疏性
非线性SVM
使用非线性函数将输入数据映射至高维空间后应用线性SVM可得到非线性SVM。非线性SVM有如下优化问题:
类比软边距SVM,非线性SVM有如下对偶问题:
注意到式中存在映射函数内积,因此可以使用核方法,即直接选取核函数:![]()
非线性SVM的对偶问题的KKT条件可同样类比软边距线性SVM。
二、代码实现
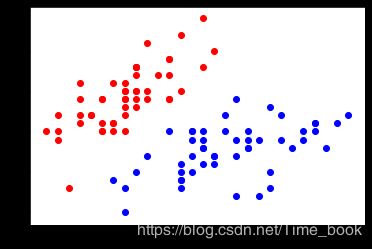
未经标准化的原始数据点分布
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.preprocessing import StandardScaler
from sklearn.svm import LinearSVC
iris = datasets.load_iris()
X = iris.data
y = iris.target
# X = X[y<2,:2] #只取y<2的类别,也就是0 1 并且只取前两个特征 y = y[y<2]
plt.scatter(X[y==0,0],X[y==0,1],color='red')
plt.scatter(X[y==1,0],X[y==1,1],color='blue')
plt.show()
# 标准化
standardScaler = StandardScaler()
standardScaler.fit(X) #计算训练数据的均值和方差
X_standard = standardScaler.transform(X) #再用scaler中的均值和方差来转换X,使X标准化
svc = LinearSVC(C=1e9) #线性SVM分类器
svc.fit(X_standard,y) # 训练svm
from sklearn.preprocessing import PolynomialFeatures,StandardScaler
from sklearn.svm import LinearSVC
from sklearn.pipeline import Pipeline
def PolynomialSVC(degree,C=1.0):
return Pipeline([ ("poly",PolynomialFeatures(degree=degree)),#生成多项式
("std_scaler",StandardScaler()),#标准化
("linearSVC",LinearSVC(C=C))#最后生成svm
])
from matplotlib.colors import ListedColormap
def plot_decision_boundary(model, axis):
x0, x1 = np.meshgrid( np.linspace(axis[0], axis[1], int((axis[1]-axis[0])*100)).reshape(-1,1), np.linspace(axis[2], axis[3], int((axis[3]-axis[2])*100)).reshape(-1,1) )
X_new = np.c_[x0.ravel(), x1.ravel()]
y_predict = model.predict(X_new)
zz = y_predict.reshape(x0.shape)
custom_cmap = ListedColormap(['#EF9A9A','#FFF59D','#90CAF9'])
plt.contourf(x0, x1, zz, linewidth=5, cmap=custom_cmap)
# 绘制决策边界
# plot_decision_boundary(svc,axis=[-3,3,-3,3])
# # x,y轴都在-3到3之间
# # 绘制原始数据
# plt.scatter(X_standard[y==0,0],X_standard[y==0,1],color='red')
# plt.scatter(X_standard[y==1,0],X_standard[y==1,1],color='blue')
# plt.show()
使用多项式特征和核函数
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
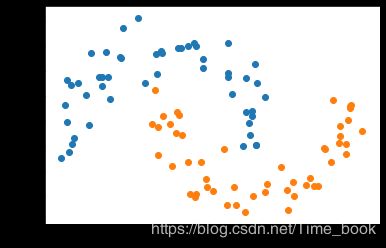
X, y = datasets.make_moons()
#使用生成的数据
print(X.shape)
# (100,2)
print(y.shape)
# (100,)
(100, 2)
(100,)
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
增加噪声点
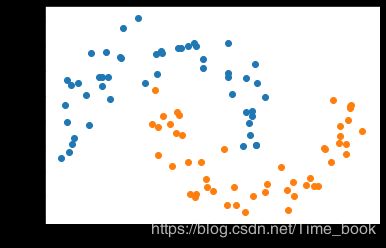
X, y = datasets.make_moons(noise=0.15,random_state=777)
#随机生成噪声点,random_state是随机种子,noise是方差
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
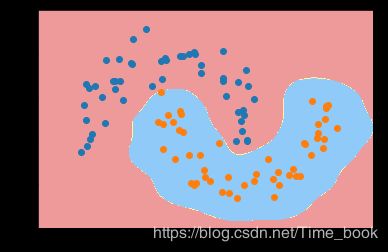
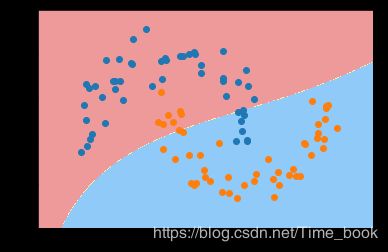
利用多项式特征的SVM来进行分类
from sklearn.linear_model import LogisticRegressionCV
poly_svc = PolynomialSVC(degree=3)
poly_svc.fit(X,y)
plot_decision_boundary(poly_svc,axis=[-1.5,2.5,-1.0,1.5])
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
利用核对数据进行处理,使其维度提升,使原本线性不可分的数据,在高维空间变成线性可分的。再用线性SVM来进行处理。
from sklearn.svm import SVC
def PolynomialKernelSVC(degree,C=1.0):
return Pipeline([ ("std_scaler",StandardScaler()), ("kernelSVC",SVC(kernel="poly")) # poly代表多项式特征
])
poly_kernel_svc = PolynomialKernelSVC(degree=3)
poly_kernel_svc.fit(X,y)
plot_decision_boundary(poly_kernel_svc,axis=[-1.5,2.5,-1.0,1.5])
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
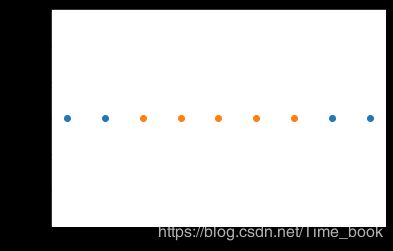
模拟升维
import numpy as np
import matplotlib.pyplot as plt
x = np.arange(-4,5,1)
#生成测试数据
y = np.array((x >= -2 ) & (x <= 2),dtype='int')
plt.scatter(x[y==0],[0]*len(x[y==0]))
# x取y=0的点, y取0,有多少个x,就有多少个y
plt.scatter(x[y==1],[0]*len(x[y==1]))
plt.show()
利用高斯核函数,进行数据映射
# 高斯核函数
def gaussian(x,l):
gamma = 1.0
return np.exp(-gamma * (x -l)**2)
l1,l2 = -1,1
X_new = np.empty((len(x),2))
#len(x) ,2
for i,data in enumerate(x):
X_new[i,0] = gaussian(data,l1)
X_new[i,1] = gaussian(data,l2)
plt.scatter(X_new[y==0,0],X_new[y==0,1])
plt.scatter(X_new[y==1,0],X_new[y==1,1])
plt.show()
超参数
生成数据
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
X,y = datasets.make_moons(noise=0.15,random_state=777)
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
定义一个RBF核的SVM
from sklearn.preprocessing import StandardScaler
from sklearn.svm import SVC
from sklearn.pipeline import Pipeline
def RBFKernelSVC(gamma=1.0):
return Pipeline([ ('std_scaler',StandardScaler()), ('svc',SVC(kernel='rbf',gamma=gamma)) ])
svc = RBFKernelSVC()
svc.fit(X,y)
plot_decision_boundary(svc,axis=[-1.5,2.5,-1.0,1.5])
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
改变svc = RBFKernelSVC() 中的参数为100
from sklearn.preprocessing import StandardScaler
from sklearn.svm import SVC
from sklearn.pipeline import Pipeline
def RBFKernelSVC(gamma=1.0):
return Pipeline([ ('std_scaler',StandardScaler()), ('svc',SVC(kernel='rbf',gamma=gamma)) ])
svc = RBFKernelSVC(100)
svc.fit(X,y)
plot_decision_boundary(svc,axis=[-1.5,2.5,-1.0,1.5])
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
改成10
from sklearn.preprocessing import StandardScaler
from sklearn.svm import SVC
from sklearn.pipeline import Pipeline
def RBFKernelSVC(gamma=1.0):
return Pipeline([ ('std_scaler',StandardScaler()), ('svc',SVC(kernel='rbf',gamma=gamma)) ])
svc = RBFKernelSVC(10)
svc.fit(X,y)
plot_decision_boundary(svc,axis=[-1.5,2.5,-1.0,1.5])
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
改成0.1
from sklearn.preprocessing import StandardScaler
from sklearn.svm import SVC
from sklearn.pipeline import Pipeline
def RBFKernelSVC(gamma=1.0):
return Pipeline([ ('std_scaler',StandardScaler()), ('svc',SVC(kernel='rbf',gamma=gamma)) ])
svc = RBFKernelSVC(0.1)
svc.fit(X,y)
plot_decision_boundary(svc,axis=[-1.5,2.5,-1.0,1.5])
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
boston = datasets.load_boston()
X = boston.data
y = boston.target
from sklearn.model_selection import train_test_split
X_train,X_test,y_train,y_test = train_test_split(X,y,random_state=777)
# 把数据集拆分成训练数据和测试数据
from sklearn.svm import LinearSVR
from sklearn.svm import SVR
from sklearn.preprocessing import StandardScaler
def StandardLinearSVR(epsilon=0.1):
return Pipeline([ ('std_scaler',StandardScaler()), ('linearSVR',LinearSVR(epsilon=epsilon)) ])
svr = StandardLinearSVR()
svr.fit(X_train,y_train)
svr.score(X_test,y_test)
0.6991720760289629
三、参考文章
http://blog.sina.com.cn/s/blog_6c3438600102yn9x.html
百度百科