PCA
PCA 数据降维
一、主成分的计算步骤
1- 对原始数据进行标准化处理,消除量纲
2- 计算标准化数据的相关系数矩阵
3- 计算标准化数据的相关系数矩阵的特征根及对应的特征向量
4- 选出最大的特征根,对应的特征向量等于第一主成分的系数;选出第二大的特征根,对应的特征向量等于 第二主成分的系数;以此类推
5- 计算累积贡献率,选择恰当的主成分个数
6- 解释主成分:写出前k个主成分的表达式
7- 确定各样本的主成分得分
8- 根据主成分得分的数据,做进一步的统计分析
二、R语言实现
- 载入需要的数据包 psych
apply(USArrests,2,mean)
## Murder Assault UrbanPop Rape
## 7.788 170.760 65.540 21.232
apply(USArrests,2,var)
## Murder Assault UrbanPop Rape
## 18.97047 6945.16571 209.51878 87.72916
##发现均值和方差的差异较大,因此必须进行标准化
## scale(USArrests,center = T,scale = T)
##然后进行PCA分析,函数包含了标准化操作
pr.out<-prcomp(USArrests,scale=T)
names(pr.out)
## [1] "sdev" "rotation" "center" "scale" "x"
##center和scale是标准化前的均值和标准差
##rotation包含了主成分载荷信息,列向量是主成分载荷向量
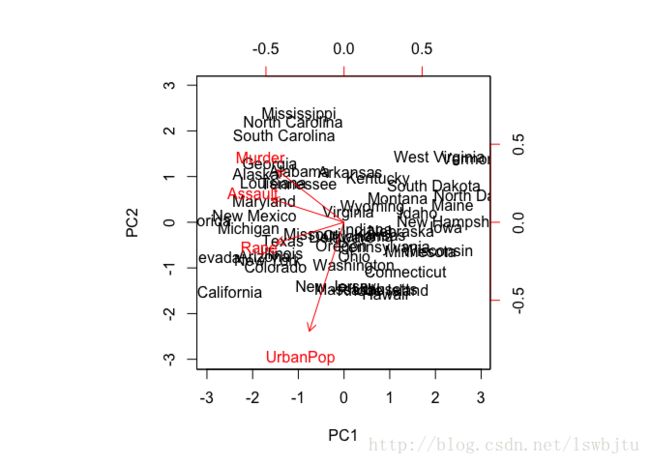
pr.out$rotation
## PC1 PC2 PC3 PC4
## Murder -0.5358995 0.4181809 -0.3412327 0.64922780
## Assault -0.5831836 0.1879856 -0.2681484 -0.74340748
## UrbanPop -0.2781909 -0.8728062 -0.3780158 0.13387773
## Rape -0.5434321 -0.1673186 0.8177779 0.08902432
biplot(pr.out,scale=0) ##前两个主成分的双标图##主成分的标准差
pr.out$sdev ##一般来说,第一主成分的方差>第二主成分的方差>.....
## [1] 1.5748783 0.9948694 0.5971291 0.4164494
pr.out$var<-pr.out$sdev^2
##计算每个主成分的方差解释比例
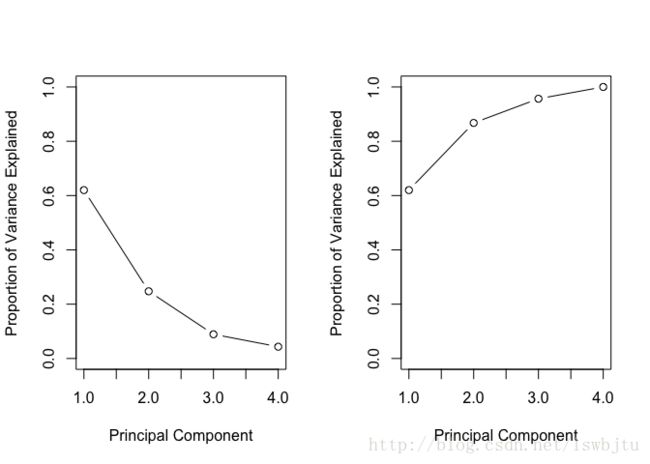
pve<-pr.out$var/sum(pr.out$var)
pve
## [1] 0.62006039 0.24744129 0.08914080 0.04335752
##绘制每个主成分的PVE和累积PVE图
par(mfrow=c(1,2))
plot(pve,xlab = "Principal Component",ylab="Proportion of Variance Explained",
ylim=c(0,1),type='b')
plot(cumsum(pve),xlab = "Principal Component",ylab="Proportion of Variance Explained",
ylim=c(0,1),type='b')PVE图和累积PVE图可以用于选择主成分,一般选取前几个能解释大部分数据方差的主成分。
三、判断主成分的个数
1.可根据上述提到的累积PVE图,解释大部分方差。
2.主成分对应于相关系数矩阵的特征值,第一主成分与最大特征值关联,第二主成分与第二大的特征值关联 - Kaiser-Harris准则:保留特征值大于1的主成分,特征值小于1的成分解释的方差更少。
Cattell碎石检验:绘制了特征值与主成分数的图形,在图形变化最大处之上的主成分保留
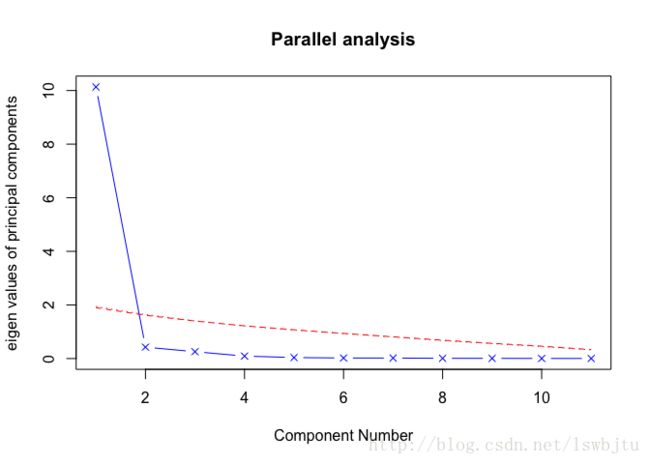
平行分析:用与初始矩阵大小相同的随机数矩阵,若基于真实数据的某个特征值大于一组随机数矩阵相应 的平均特征值,那么该主成分可以保留。
library(psych)
fa.parallel(USJudgeRatings[,-1],fa="pc",n.iter=100,show.legend=F,main = "Parallel analysis")## Parallel analysis suggests that the number of factors = NA and the number of components = 1
四、提取主成分
principal(USJudgeRatings[,-1],nfactors = 1,scores = T)
## Principal Components Analysis
## Call: principal(r = USJudgeRatings[, -1], nfactors = 1, scores = T)
## Standardized loadings (pattern matrix) based upon correlation matrix
## PC1 h2 u2 com
## INTG 0.92 0.84 0.1565 1
## DMNR 0.91 0.83 0.1663 1
## DILG 0.97 0.94 0.0613 1
## CFMG 0.96 0.93 0.0720 1
## DECI 0.96 0.92 0.0763 1
## PREP 0.98 0.97 0.0299 1
## FAMI 0.98 0.95 0.0469 1
## ORAL 1.00 0.99 0.0091 1
## WRIT 0.99 0.98 0.0196 1
## PHYS 0.89 0.80 0.2013 1
## RTEN 0.99 0.97 0.0275 1
##
## PC1
## SS loadings 10.13
## Proportion Var 0.92
##
## Mean item complexity = 1
## Test of the hypothesis that 1 component is sufficient.
##
## The root mean square of the residuals (RMSR) is 0.04
## with the empirical chi square 6.21 with prob < 1
##
## Fit based upon off diagonal values = 1
## nfactors:设定主成分个数
## scores:是否计算主成分得分计算结果中,h2指主成分对每个变量的解释比例,u2指无法被解释的比例,SS 表示主成分对应的特征值,Proportion 表示主成分解释比例
五、主成分旋转
- 正交旋转:选择的成分保持不相关
- 斜交旋转:选择的成分保持相关 正交旋转,方差极大旋转,对载荷矩阵的列进行去噪,使每个成分只由一组有限的变量来解释。
##varimax:方差极大旋转
data_pca<-principal(USJudgeRatings[,-1],nfactors = 2,rotate='varimax',scores=T)
data_pca
## Principal Components Analysis
## Call: principal(r = USJudgeRatings[, -1], nfactors = 2, rotate = "varimax",
## scores = T)
## Standardized loadings (pattern matrix) based upon correlation matrix
## RC1 RC2 h2 u2 com
## INTG 0.48 0.87 0.98 0.0164 1.6
## DMNR 0.47 0.87 0.97 0.0252 1.6
## DILG 0.81 0.54 0.95 0.0531 1.7
## CFMG 0.88 0.45 0.97 0.0312 1.5
## DECI 0.89 0.43 0.97 0.0282 1.5
## PREP 0.82 0.56 0.98 0.0232 1.8
## FAMI 0.81 0.55 0.96 0.0405 1.8
## ORAL 0.77 0.63 0.99 0.0091 1.9
## WRIT 0.77 0.62 0.98 0.0196 1.9
## PHYS 0.79 0.44 0.82 0.1779 1.6
## RTEN 0.70 0.70 0.98 0.0176 2.0
##
## RC1 RC2
## SS loadings 6.30 4.26
## Proportion Var 0.57 0.39
## Cumulative Var 0.57 0.96
## Proportion Explained 0.60 0.40
## Cumulative Proportion 0.60 1.00
##
## Mean item complexity = 1.7
## Test of the hypothesis that 2 components are sufficient.
##
## The root mean square of the residuals (RMSR) is 0.02
## with the empirical chi square 1.53 with prob < 1
##
## Fit based upon off diagonal values = 1获得主成分与各个变量的线性关系:
data_pca$weights
## RC1 RC2
## INTG -0.48346666 0.74273535
## DMNR -0.48569660 0.74465965
## DILG 0.20799349 -0.10532833
## CFMG 0.37290093 -0.31034542
## DECI 0.39824278 -0.34206511
## PREP 0.19639630 -0.08844609
## FAMI 0.19341796 -0.08612799
## ORAL 0.07139417 0.06794620
## WRIT 0.07630098 0.06104358
## PHYS 0.29502592 -0.22487622
## RTEN -0.07162767 0.24353657获得主成分的得分:
data_pca$scores
## RC1 RC2
## AARONSON,L.H. -0.43625484 0.244382210
## ALEXANDER,J.M. -0.10942078 1.324198203
## ARMENTANO,A.J. -0.13200175 0.275566691
## BERDON,R.I. 1.03630087 0.524734415
## BRACKEN,J.J. -0.84161349 -2.393324391
## BURNS,E.B. 0.07016044 1.133641671
## CALLAHAN,R.J. 0.80342055 0.955949303
## COHEN,S.S. -1.48721483 -2.158157424
## DALY,J.J. 0.78118367 0.862237453
## DANNEHY,J.F. 1.34837402 -1.144061632
## DEAN,H.H. -0.22300367 0.106829116
## DEVITA,H.J. -1.13307674 0.680899493
## DRISCOLL,P.J. -1.67954055 1.743477389
## GRILLO,A.E. -1.03431030 -0.333006989
## HADDEN,W.L.JR. 0.55568244 -0.013057617
## HAMILL,E.C. -0.24589775 0.081800124
## HEALEY.A.H. -1.11092595 -0.072503392
## HULL,T.C. 0.36760733 -0.813535607
## LEVINE,I. 0.26214560 -0.002514484
## LEVISTER,R.L. -0.42930902 -1.642625753
## MARTIN,L.F. -1.70399893 1.199710785
## MCGRATH,J.F. -0.93021019 -0.407508501
## MIGNONE,A.F. -3.02272761 0.563745360
## MISSAL,H.M. -0.71555075 0.842676970
## MULVEY,H.M. 0.96871191 0.476402631
## NARUK,H.J. 1.43954883 0.492593338
## O'BRIEN,F.J. 0.05462851 0.681736508
## O'SULLIVAN,T.J. 0.59487618 0.998368712
## PASKEY,L. 1.03774800 -0.338111014
## RUBINOW,J.E. 0.94982286 1.212624991
## SADEN.G.A. 1.76584671 -1.637857192
## SATANIELLO,A.G. 0.20423752 0.019872042
## SHEA,D.M. 1.07210787 -0.036757710
## SHEA,J.F.JR. 0.76998366 0.844587964
## SIDOR,W.J. -1.37809553 -1.724334530
## SPEZIALE,J.A. 0.78017665 0.048591613
## SPONZO,M.J. 0.26696254 0.271806601
## STAPLETON,J.F. 0.24752546 0.051941979
## TESTO,R.J. -0.32808603 -0.645394531
## TIERNEY,W.L.JR. 0.81387101 -0.330270574
## WALL,R.A. 0.38346323 -1.766162327
## WRIGHT,D.B. -0.13301770 0.895446778
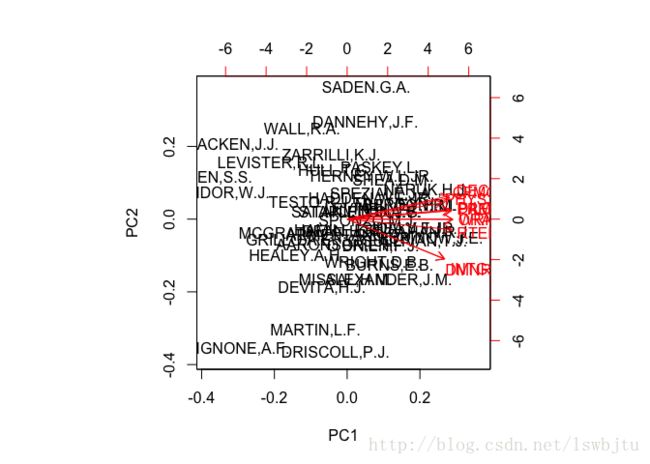
## ZARRILLI,K.J. 0.49987054 -1.074638671获得主成分分析图
biplot(prcomp(USJudgeRatings[,-1],scale=T))推广:在多重共线性存在时,线性回归的效果并不是很好,这时候可以考虑先用PCA降维,挑选出少量的主成分以后,将主成分作为自变量进行预测,此时得到的效果要更好。即避免了多重共线性,又有效的解释了自变量和因变量的关系。