2d和3d cnn 解决医疗影像分析问题
文章目录
- 项目介绍
- 预处理方法
- augmentation(flip, resize, crop)
- spacing
- class weight
- 交叉验证
- 模型
- 权值初始化方法
- Xavier 正态分布
- kaiming 正态分布
- 方法
- 2维的方法
- 3维的方法
- 方法1:
- 方法2:
- 结果
- 不同模型之间的比较
- resnet34
- 训练
- 测试
- densenet
- 训练
- 测试
- 调整参数后的比较
- 情况1
- 情况2
- 更改预处理中数据增广的组合
- 是否class weight的比较
- 设置了class weight
- 未设置class weight
- 为不同的层设置不同的学习率
- 采用不同的优化器
- 调整学习率
- 总结:
- 感想
项目介绍
本次项目的主题是对医学图像进行一个分类预测。训练数据是来自两个医院提供的实际病人的肠壁肿瘤MRI图像和一个带有标记的excel文件。图像具体选择的是T2weighted图像。训练的类别标签为“TRG分级”,取值有四种(0,1,2,3),为离散的值,即该问题为一个四分类问题。四种类别的数据量十分不均衡。如:提供的数据中,CC-ROI数据表中的TRG分级(0,1,2,3)类对应的数据为(176,166,307,4)总计653个样例。而6-ROI的数据表中的TRG分级(0,1,2,3)类对应的数据为(100,109,124,13)总计346个样例。另外,MRI图像的肿瘤区域大小不一。因此需要进行较多的数据预处理步骤
预处理方法
augmentation(flip, resize, crop)
由于目前拥有的实际数据中,CC-ROI有653个样例,6-ROI为346个样例,样例数不够,因此需要进行数据增广。通过使用数据增广,增加训练数据集,让数据集尽可能的多样化,使得训练的模型具有更强的泛化能力。
现有的各大深度学习框架都已经自带了数据增广,因此直接调用对应的函数即可。(本项目使用pytorch)
train_transform = transforms.Compose([
# transforms.CenterCrop(size=224),
# transforms.RandomRotation(degrees=[-10, 10]),
# transforms.CenterCrop(size=512)
# transforms.RandomCrop(size=224),
# transforms.RandomResizedCrop(size=224),
transforms.RandomHorizontalFlip(p=0.5),
transforms.RandomVerticalFlip(p=0.5),
# transforms.ColorJitter(brightness=0.1, contrast=0.1),
])
在深度学习中的数据增广一般会采用多种增广方式的组合,这里就会涉及到矩阵乘法运算,根据其运算的规则,可以知道不同的组合顺序结果是不一样的,即线性代数中的 A B ≠ B A AB \neq BA AB=BA ,当然特例除外。
- Flip代表的是翻转,即图像镜像,包括水平和垂直翻转
- resize即进行图像缩放
- crop是进行图像的切割
spacing
医学图像在xy方向是固定的512x512,在z轴分辨率不同会导致图像矩阵深度不一样。如果不对分辨率标准化可以,直接对图像进行缩放到满足神经网络标准化输入的要求。但是在3d图像处理过程中,常常需要对其进行分辨率标准化。实际应用中,有标准到1x1x1或者3x3x3。做这种的标准化并不是为了应用于神经网络,传统的医学模式识别也是要这么做的。ct图像中有几个非常重要的量,xy方向的spacing,大多数ct的spacing都是0.5左右,而z轴是slide thickness,也就是层厚。国内外甚至每个脏器的扫描都会有不同。比如luna数据集,肺结节很小,又是国外的数据,普遍层厚非常小,0.6mm到1.5mm左右。比如miccai肝脏数据集,基本是1.5mm。在我们的数据集T2序列中,层厚从0.8px/mm 到1.8px/mm不等,当面临的是数据源的差别很大,神经网络的泛化能力根本做不到一个模型处理所有情况的程度,由于3D医学影像在z轴分辨率是差别太大,如果不做分辨率标准化,会导致泛化能力下降,检测的时候假阳性会很多。
这次实验中使用的spacing处理步骤为:
- 获取每张图片的spacing,计算所有slice的spacing众数
- 根据该spacing对所有图片进行resize,使其spacing相同。此时图片xy方向尺寸不同(最大为615*615)
- 将所有图片resize到224 * 224
# 1. spacing resize
if self.is_spacing is True:
shape = self.dataset[index]["shape"]
shape_spc = self.dataset[index]["shape_spc"]
if shape[0] != shape_spc[0]:
img = img.resize(size=(shape_spc[0], shape_spc[1]), resample=Image.NEAREST) # spacing resize
# 2. 以肿瘤中心切割
h_min, h_max, w_min, w_max = self.dataset[index]["tumor_hw_min_max_spc"]
tumor_origin = ( (h_min + h_max) / 2, (w_min + w_max) / 2 ) # 肿瘤中心点坐标
if self.is_train is True:
crop_size = 224 # 切割后图片大小
else:
crop_size = 224 # 切割后图片大小
img = TF.crop(
img=img, # Image to be cropped.
i=int(round(tumor_origin[0] - crop_size / 2)), # Upper pixel coordinate.
j=int(round(tumor_origin[1] - crop_size / 2)), # Left pixel coordinate.
h=crop_size, # Height of the cropped image.
w=crop_size # Width of the cropped image.
)
return img
class weight
由于每一类的样本数目不均匀,因此进行class weight处理。使用样本数的倒数作为每一类的weight。
def get_class_weight(self):
class_num = [0 for x in range(settings.num_classes)] # 统计每类样本的数量
for data in self.dataset:
label = settings.class_specifier[data['label']]
class_num[label] += 1
class_weight = [1.0 / x for x in class_num]
"""
class_weight normalization, class_weihght: $$/frac{1/n_i}{1/n1 + 1/n2 + 1/n3}$$
after class_weight normalization, the sum of class_weight is 1
"""
sum_class_weight = sum(class_weight)
class_weight = [x / sum_class_weight for x in class_weight]
return class_weight
交叉验证
采用5折交叉验证。对于不同的病人类型,按照比例随机划分。
for data_choose in data_chooses:
log.logger.info("Initializing dataset {} ...".format(data_choose))
patients = _load_inf(data_choose) # 读取病人的编号及结局(标签){sub001:0, sub002:0}
dataset = {str(x): {} for x in range(K)} # 储存id和label
class_patients = [[] for x in range(settings.num_classes)]
# 划分训练集和测试集
for patient_id, patient_label in patients.items():
# print(patient_id, patient_label)
class_patients[patient_label].append(patient_id)
print(len(class_patients[0]),len(class_patients[1]),len(class_patients[2]), len(class_patients[3]))
# 随机选择k-fold,没折比例相同
for i in range(len(class_patients)):
rand_index = np.arange(len(class_patients[i])) # 对每一类病人,获取随机下标
np.random.shuffle(rand_index)
for j in range(len(rand_index)):
if j < 0.2 * len(rand_index): # 1/5
dataset["0"][class_patients[i][rand_index[j]]] = i # id:label
elif j >= 0.2 * len(rand_index) and j < 0.4 * len(rand_index):
dataset["1"][class_patients[i][rand_index[j]]] = i
elif j >= 0.4 * len(rand_index) and j < 0.6 * len(rand_index):
dataset["2"][class_patients[i][rand_index[j]]] = i
elif j >= 0.6 * len(rand_index) and j < 0.8 * len(rand_index):
dataset["3"][class_patients[i][rand_index[j]]] = i
else:
dataset["4"][class_patients[i][rand_index[j]]] = i
模型
- resnet34
- densenet
- vgg19
权值初始化方法
Xavier 正态分布
torch.nn.init.xavier_normal_(tensor, gain=1)
kaiming 正态分布
torch.nn.init.kaiming_uniform_(tensor, a=0, mode='fan_in', nonlinearity='leaky_relu')
方法
2维的方法
对于每个病人的每张slides,取其中含有肿瘤的部分,对图片预处理之后,进行单张图片的训练
3维的方法
方法1:
- 对病人的slides数进行统一
- 将整个脏器放入模型进行训练
方法2:
- 对病人的slides数进行统一
- 对每个病人取三张slides为一组,即进行切块,然后传入模型
结果
不同模型之间的比较
resnet34
训练
测试
-
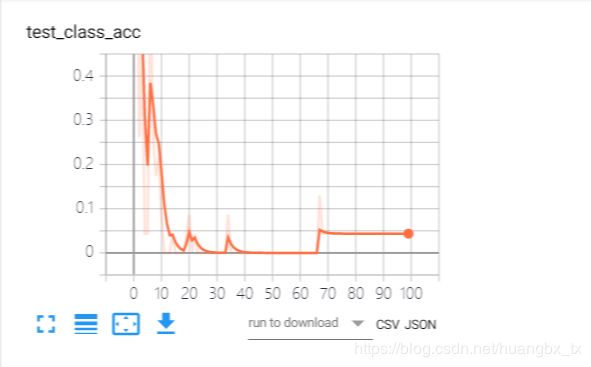
accuracy of class 0

类别0出现了较大波动,最后准确度也大致在20%左右。epoch为10时准确度较高。原因可能是采样不平均或者是样本数不够。 -
accuracy of class 2

类别2在第20个epoch的时候准确率达到60%,但后面下降到40%+,原因可能是过拟合了
- 准确率为:
- class 0: 28%
- class 1: 28%
- class 2: 41%
- class 3: 4%
- 召回率为:
- class 0: 29%
- class 1: 28%
- class 2: 37%
- class 3: 11%
并且从混淆矩阵中可以看出,该模型倾向于将结果预测成class 2,这与class 2的样本数占了较大比重有关。
densenet
训练
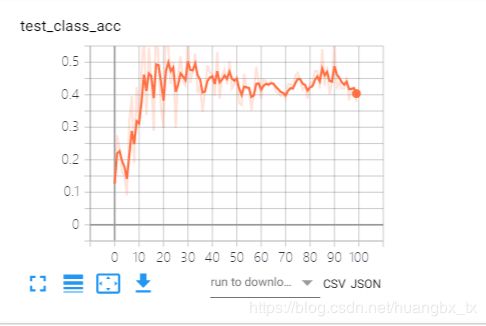
其他几个类型的曲线也和class 0 曲线类似。与resnet相比,densenet同样也是在大约第20个epoch的时候达到了100%
测试
-
混淆矩阵

从混淆矩阵可以看出,densenet将最后一类全部预测错误。并且同样有倾向于预测倾向于class 2的情况。但是在对class 0的预测中,尽管预测结果不好,但是最大值出现在class 1而不是class 2
调整参数后的比较
情况1
Hyper parameters
batch_size = 80
num_epochs = 100
lr = 1e-4
momentum = 0.9
weight_decay = 5e-4
情况1的结果即第一组resnet实验的结果
情况2
batch_size = 160
num_epochs = 100
lr = 1e-6
momentum = 0.9
weight_decay = 1e-6
batch_size增大一倍,相对于小批量处理,运行速度会更慢。但全数据集确定的方向能够更好地代表样本总体,从而能够更准确地朝着极值所在的方向。批次越小,梯度的估值就越不准确。
learning rate缩小:有上述实验可以看出,学习率设置的过大时,梯度可能会在最小值附近来回震荡,甚至可能无法收敛。因此,尝试将学习率缩小。
实验结果:
更改预处理中数据增广的组合
-
原始组合为:
transforms.RandomHorizontalFlip(p=0.5), transforms.RandomVerticalFlip(p=0.5),结果为上述resnet的结果
-
增加增广操作组合
transforms.CenterCrop(size=224), transforms.RandomRotation(degrees=[-10, 10]), transforms.RandomHorizontalFlip(p=0.5), transforms.RandomVerticalFlip(p=0.5),
结果为:
1. accuracy of class 0

2. accuracy of class 1

3. accuracy of class 2

3. accuracy of class 3

4. 混淆矩阵

class 2 的预测准确度有所提高,并且偏向于同一类的倾向不会很严重。
是否class weight的比较
设置了class weight
模型的训练结果即为上图展示的情况。
未设置class weight
-
混淆矩阵

可以看出,在没有class weight的情况下,预测结果几乎很严重的倾向于class 2,而class 3的预测数几乎全部趋近于0。很明显,模型的判断收到了样本数的很大影响。因此,设置class weight是正确的操作。
为不同的层设置不同的学习率
ignored_params = list(map(id, net.fc3.parameters())) # 返回的是 parameters 的 内存地址
base_params = filter(lambda p: id(p) not in ignored_params, net.parameters()) # 返回 base params 的 内存地址
optimizer = optim.SGD([
{'params': base_params},
{'params': net.fc3.parameters(), 'lr': 0.001*10}], 0.001, momentum=0.9, weight_decay=1e-4)
采用不同的优化器
-
SGD
optimizer = torch.optim.SGD(params=model.parameters(), lr=lr, momentum=momentum, weight_decay=weight_decay) -
Adam
optimizer = torch.optim.Adam(params=model.parameters(), weight_decay=weight_decay, lr=lr) -
LBFGS
class torch.optim.LBFGS(params, lr=1, max_iter=20, max_eval=None, tolerance_ grad=1e-05, tolerance_change=1e-09, history_size=100, line_search_fn=None)
结果显示不同的优化器在训练的结果上并没有什么差异,但是训练的效率不同。并且LBFGS更加节省内存。
调整学习率
-
stepLR
等间隔调整学习率,调整倍数为 gamma 倍,调整间隔为 step_size。间隔单位是step。需要注意的是,step 通常是指 epoch,不要弄成 iteration 了。torch.optim.lr_scheduler.StepLR(optimizer, step_size, gamma=0.1, las t_epoch=-1) -
MultiStepLR
按设定的间隔调整学习率。这个方法适合后期调试使用,观察 loss 曲线,为每个实验定制学习率调整时机torch.optim.lr_scheduler.MultiStepLR(optimizer, milestones, gamma =0.1, last_epoch=-1) -
CosineAnnealingLR
以余弦函数为周期,并在每个周期最大值时重新设置学习率 -
lr_scheduler.LambdaLR
为不同参数组设定不同学习率调整策略。调整规则为, l r = b a s e _ l r ∗ l a m b d a ( s e l f . l a s t _ e p o c h ) 。 lr = base\_lr * lambda(self.last\_epoch) 。 lr=base_lr∗lambda(self.last_epoch)。ignored_params = list(map(id, net.fc3.parameters())) base_params = filter(lambda p: id(p) not in ignored_params, net.parameters()) optimizer = optim.SGD([ {'params': base_params}, {'params': net.fc3.parameters(), 'lr': 0.001*100}], 0.001, momentum=0.9, weight_decay=1e-4) lambda1 = lambda epoch: epoch // 3 lambda2 = lambda epoch: 0.95 ** epoch scheduler = torch.optim.lr_scheduler.LambdaLR(optimizer, lr_lambda=[lambda1, lambda2]) for epoch in range(100): scheduler.step() print('epoch: ', i, 'lr: ', scheduler.get_lr()) train(...) validate(...)
总结:
PyTorch 提供了六种学习率调整方法,可分为三大类,分别是:
- 有序调整;
- 自适应调整;
- 自定义调整。
第一类,依一定规律有序进行调整,这一类是最常用的,分别是等间隔下降(Step),按需设定下降间隔(MultiStep),指数下降(Exponential)和 CosineAnnealing。这四种方法的调整时机都是人为可控的,也是训练时常用到的。
第二类,依训练状况伺机调整,这就是 ReduceLROnPlateau 方法。该法通过监测某一指标的变化情况,当该指标不再怎么变化的时候,就是调整学习率的时机,因而属于自适应的调整。
第三类,自定义调整,Lambda。Lambda 方法提供的调整策略十分灵活,我们可以为不同的层设定不同的学习率调整方法,这在 fine-tune 中十分有用,我们不仅可为不同的层设定不同的学习率,还可以为其设定不同的学习率调整策略。
结果表明,使用lambda方法模型能够更快地收敛。在本实验中,在大约18个epoch时就能收敛。
感想
此次项目在老师的带领下得以获得真实的案例进行学习,所有数据来源全部来自医院的真实数据,在保证保密性的要求下让我们学生能够进行学习体会,机会十分难得,并且在项目过程中也得以与医生和老师进行多次直接交流,因此对于医疗图像的基本知识也获得了许多额外的了解。并且由于数据来源的特殊性,战如何在进行数据预处理时候的一些操作也是一项较大的挑战。在做这个项目的过程中,我们花了大量的时间进行数据的预处理操作。除此之外,我们也学会了如何进行模型的评估,如通过召回率,准确度,F1 score进行判定。并且,也学会了如何利用多种不同的模型进行比较。从一开始的自己通过pytorch搭建简单的vgg,到后面的resnet,再到后面使用预训练模型。
起初本次项目是准备从3D-cnn入手,原因在于对于医学图像,都是三维的信息,因此采用3d-cnn能够保存z轴上面的信息,效果必然会比2维的好。于是我们尝试了使用降采样再分割的方法,但由于刚入门,因此并没有做好3D的处理。出现了爆显存的现象。老师也曾鼓励说3D的结果应该会更好,但是中间遇到了很多的问题没能解决,最后只能放弃,十分遗憾。