SVM算法编程练习
这里写自定义目录标题
- SVM算法编程练习
- 一、支持向量机SVM
- 1、超平面
- 2、算法
- ①、线性SVM(linear SVM)
- ②、非线性SVM
- 二、Soft Margin SVM
- 1、加载鸢尾花数据集并查看散点图分布
- 2、绘制决策边界
- 3、再次实例化SVC,重新传入一个较小的C
- 三、使用多项式与核函数
- 1、加载月亮数据集
- 2、绘制散点图
- 3、加入噪声点
- 4、通过多项式特征的SVM进行分类
- 5、使用核技巧来对数据进行处理
- 四、核函数
- 1、产生测试点以及绘制散点图
- 2、将数据升为二维
- 五、超参数γ
- 1、加载月亮数据集
- 2、定义一个RBF核的SVM
- 3、修改γ值
- ①、修改γ为100
- ②、修改γ为10
- ③、修改为0.1
- 六、回归问题
SVM算法编程练习
一、支持向量机SVM
Svm(support Vector Mac)又称为支持向量机,是一种二分类的模型。当然如果进行修改之后也是可以用于多类别问题的分类。支持向量机可以分为线性核非线性两大类。其主要思想为找到空间中的一个更够将所有数据样本划开的超平面,并且使得本本集中所有数据到这个超平面的距离最短。
1、超平面
我们知道,分类的目的是学会一个分类函数或分类模型(或者叫做分类器),该模型能把数据库中的数据项映射到给定类别中的某一个,从而可以用于预测未知类别。对于用于分类的支持向量机,它是个二分类的分类模型。也就是说,给定一个包含正例和反例(正样本点和负样本点)的样本集合,支持向量机的目的是寻找一个超平面来对样本进行分割,把样本中的正例和反例用超平面分开,但是不是简单地分看,其原则是使正例和反例之间的间隔最大。学习的目标是在特征空间中找到一个分类超平面wx+b=0,分类面由法向量w和截距b决定。分类超平面将特征空间划分两部分,一部分是正类,一部分是负类。法向量指向的一侧是正类,另一侧为负类。
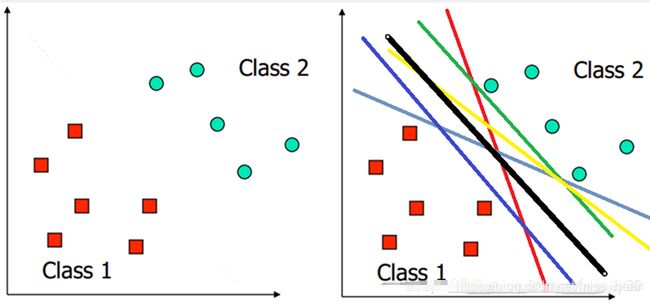
用一个二维空间里仅有两类样本的分类问题来举个小例子。假设我们给定了下图左图所示的两类点Class1和Class2。我们的任务是要找到一个线,把他们划分开。这样可以画出无数条直线将他们分开。但是哪条最好呢?这里的“好”,可以理解为对Class1和Class2都是公平的。因此下图中的黑色那条线,应该是比较符合的。
对于分类来说,我们需要确定一个分类的线,如果新的一个样本到来,如果落在线的左边,那么这个样本就归为class1类,如果落在线的右边,就归为class2这一类。那哪条线才是最好的呢?我们仍然认为是中间的那条,因为这样,对新的样本的划分结果我们才认为最可信,那这里的“好”就是可信了。另外,在二维空间,分类的就是线,如果是三维的,分类的就是面了,更高维,名字就叫超平面。因此,一般将任何维的分类边界都统称为超平面。
2、算法
①、线性SVM(linear SVM)
(1)、硬边距
给定输入数据和学习目标
![]()
,硬边界SVM是在线性可分问题中求解最大边距超平面的算法,约束条件是样本点到决策边界的距离大于等于1。硬边界SVM可以转化为一个等价的二次凸优化问题进行求解

由上式得到的决策边界可以对任意样本进行分类
![]()
注意到虽然超平面法向量 是唯一优化目标,但学习数据和超平面的截距通过约束条件影响了该优化问题的求解 。硬边距SVM是正则化系数取0时的软边距SVM,其对偶问题和求解参见软边距SVM
2. 软边距
在线性不可分问题中使用硬边距SVM将产生分类误差,因此可在最大化边距的基础上引入损失函数构造新的优化问题。SVM使用铰链损失函数,沿用硬边界SVM的优化问题形式,软边距SVM的优化问题有如下表示:
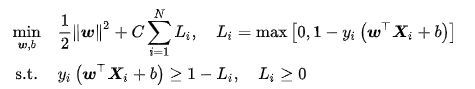
上式表明可知,软边距SVM是一个L2正则化分类器,式中 表示铰链损失函数。使用松弛变量![]() 处理铰链损失函数的分段取值后,上式可化为
处理铰链损失函数的分段取值后,上式可化为
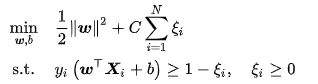
求解上述软边距SVM通常利用其优化问题的对偶性(duality)
②、非线性SVM
使用非线性函数将输入数据映射至高维空间后应用线性SVM可得到非线性SVM。非线性SVM有如下优化问题
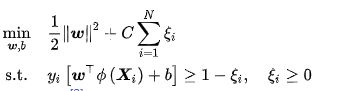
类比软边距SVM,非线性SVM有如下对偶问题
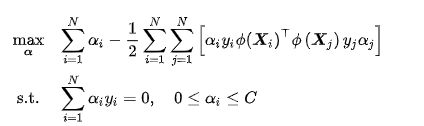
注意到式中存在映射函数内积,因此可以使用核方法,即直接选取核函数:
![]()
非线性SVM的对偶问题的KKT条件可同样类比软边距线性SVM
二、Soft Margin SVM
1、加载鸢尾花数据集并查看散点图分布
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.preprocessing import StandardScaler
from sklearn.svm import LinearSVC
iris = datasets.load_iris()
X = iris.data
y = iris.target
X = X [y<2,:2] #只取y<2的类别,也就是0 1 并且只取前两个特征
y = y[y<2] # 只取y<2的类别
# 分别画出类别0和1的点
plt.scatter(X[y==0,0],X[y==0,1],color='red')
plt.scatter(X[y==1,0],X[y==1,1],color='blue')
plt.show()
# 标准化
standardScaler = StandardScaler()
standardScaler.fit(X) #计算训练数据的均值和方差
X_standard = standardScaler.transform(X) #再用scaler中的均值和方差来转换X,使X标准化
svc = LinearSVC(C=1e9) #线性SVM分类器
svc.fit(X_standard,y) # 训练svm
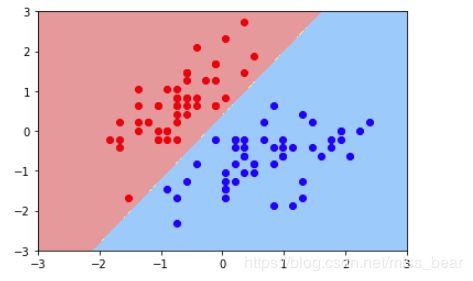
2、绘制决策边界
def plot_decision_boundary(model, axis):
x0, x1 = np.meshgrid(
np.linspace(axis[0], axis[1], int((axis[1]-axis[0])*100)).reshape(-1,1),
np.linspace(axis[2], axis[3], int((axis[3]-axis[2])*100)).reshape(-1,1)
)
X_new = np.c_[x0.ravel(), x1.ravel()]
y_predict = model.predict(X_new)
zz = y_predict.reshape(x0.shape)
from matplotlib.colors import ListedColormap
custom_cmap = ListedColormap(['#EF9A9A','#FFF59D','#90CAF9'])
plt.contourf(x0, x1, zz, linewidth=5, cmap=custom_cmap)
# 绘制决策边界
plot_decision_boundary(svc,axis=[-3,3,-3,3]) # x,y轴都在-3到3之间
# 绘制原始数据即散点图
plt.scatter(X_standard[y==0,0],X_standard[y==0,1],color='red')
plt.scatter(X_standard[y==1,0],X_standard[y==1,1],color='blue')
plt.show()
3、再次实例化SVC,重新传入一个较小的C
#C越小容错空间越大
svc2 = LinearSVC(C=0.01)
svc2.fit(X_standard,y)
plot_decision_boundary(svc2,axis=[-3,3,-3,3]) # x,y轴都在-3到3之间
# 绘制原始数据
plt.scatter(X_standard[y==0,0],X_standard[y==0,1],color='red')
plt.scatter(X_standard[y==1,0],X_standard[y==1,1],color='blue')
plt.show()
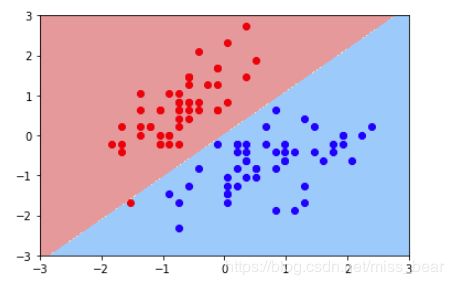
可以看出第二个决策边界有一个错误的红点,C越小的话容错空间就越大
三、使用多项式与核函数
1、加载月亮数据集
加载过后查看X与y的维度
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
#月亮数据集
X, y = datasets.make_moons() #使用生成的数据
print(X.shape) # (100,2)
print(y.shape) # (100,)
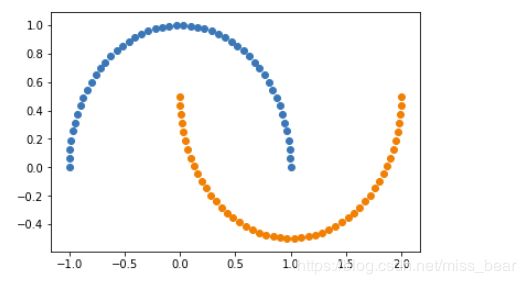
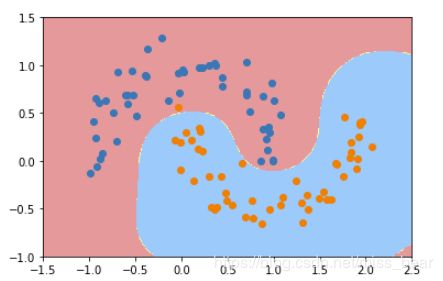
2、绘制散点图
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
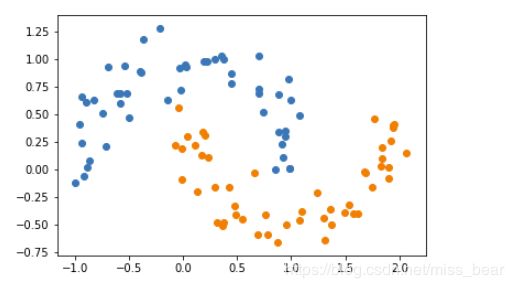
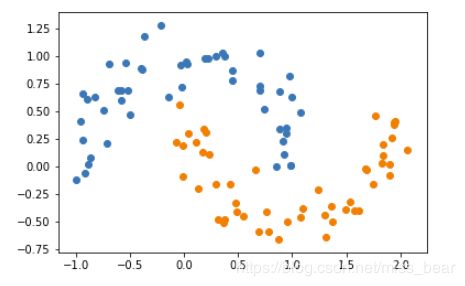
3、加入噪声点
X, y = datasets.make_moons(noise=0.15,random_state=777) #随机生成噪声点,random_state是随机种子,noise是方差
#分类
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
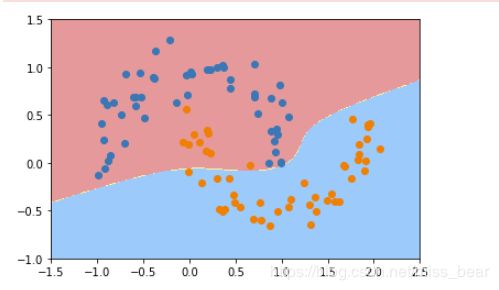
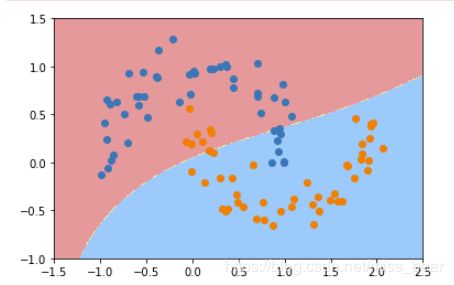
4、通过多项式特征的SVM进行分类
from sklearn.preprocessing import PolynomialFeatures,StandardScaler
from sklearn.svm import LinearSVC
from sklearn.pipeline import Pipeline
def PolynomialSVC(degree,C=1.0):
return Pipeline([
("poly",PolynomialFeatures(degree=degree)),#生成多项式
("std_scaler",StandardScaler()),#标准化
("linearSVC",LinearSVC(C=C))#最后生成svm
])
poly_svc = PolynomialSVC(degree=3)
poly_svc.fit(X,y)
plot_decision_boundary(poly_svc,axis=[-1.5,2.5,-1.0,1.5])
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
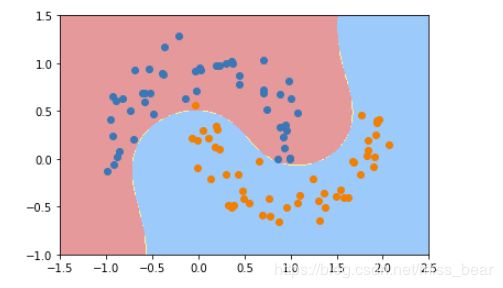
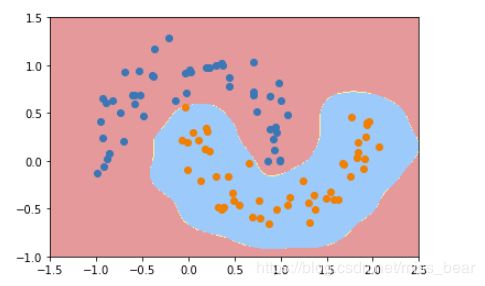
5、使用核技巧来对数据进行处理
通过使用核技巧来对数据进行处理,使其维度提升,使原本线性不可分的数据,在高维空间变成线性可分的。再用线性SVM来进行处理。
from sklearn.svm import SVC
def PolynomialKernelSVC(degree,C=1.0):
return Pipeline([
("std_scaler",StandardScaler()),
("kernelSVC",SVC(kernel="poly")) # poly代表多项式特征
])
poly_kernel_svc = PolynomialKernelSVC(degree=3)
poly_kernel_svc.fit(X,y)
plot_decision_boundary(poly_kernel_svc,axis=[-1.5,2.5,-1.0,1.5])
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
四、核函数
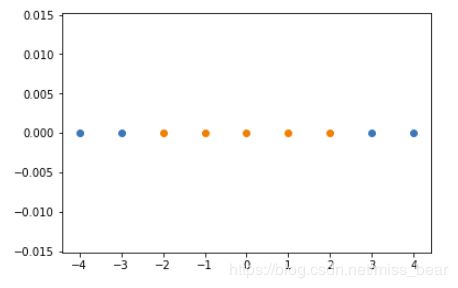
1、产生测试点以及绘制散点图
import numpy as np
import matplotlib.pyplot as plt
x = np.arange(-4,5,1)#生成测试数据
y = np.array((x >= -2 ) & (x <= 2),dtype='int')
plt.scatter(x[y==0],[0]*len(x[y==0]))# x取y=0的点, y取0,有多少个x,就有多少个y
plt.scatter(x[y==1],[0]*len(x[y==1]))
plt.show()
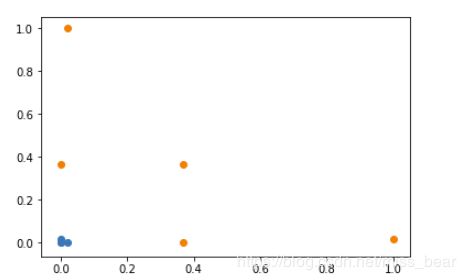
2、将数据升为二维
使用高斯核函数,将一维的数据映射到二维空间上
# 高斯核函数
def gaussian(x,l):
gamma = 1.0
return np.exp(-gamma * (x -l)**2)
l1,l2 = -1,1
X_new = np.empty((len(x),2)) #len(x) ,2
for i,data in enumerate(x):
X_new[i,0] = gaussian(data,l1)
X_new[i,1] = gaussian(data,l2)
plt.scatter(X_new[y==0,0],X_new[y==0,1])
plt.scatter(X_new[y==1,0],X_new[y==1,1])
plt.show()
五、超参数γ
1、加载月亮数据集
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
#月亮数据集
X,y = datasets.make_moons(noise=0.15,random_state=777)
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
2、定义一个RBF核的SVM
from sklearn.preprocessing import StandardScaler
from sklearn.svm import SVC
from sklearn.pipeline import Pipeline
def RBFKernelSVC(gamma=1.0):
return Pipeline([
('std_scaler',StandardScaler()),
('svc',SVC(kernel='rbf',gamma=gamma))
])
svc = RBFKernelSVC()
svc.fit(X,y)
plot_decision_boundary(svc,axis=[-1.5,2.5,-1.0,1.5])
plt.scatter(X[y==0,0],X[y==0,1])
plt.scatter(X[y==1,0],X[y==1,1])
plt.show()
3、修改γ值
①、修改γ为100
只需要修改调用函数中的参数即可,也就修改下面一行代码即可
svc = RBFKernelSVC(100)

γ取值越大,就是高斯分布的钟形图越窄,这里相当于每个样本点都形成了钟形图。很明显这样是过拟合的。
②、修改γ为10
svc = RBFKernelSVC(10)
③、修改为0.1
svc = RBFKernelSVC(0.1)
六、回归问题
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
boston = datasets.load_boston()
X = boston.data
y = boston.target
from sklearn.model_selection import train_test_split
X_train,X_test,y_train,y_test = train_test_split(X,y,random_state=777) # 把数据集拆分成训练数据和测试数据
from sklearn.svm import LinearSVR
from sklearn.svm import SVR
from sklearn.preprocessing import StandardScaler
def StandardLinearSVR(epsilon=0.1):
return Pipeline([
('std_scaler',StandardScaler()),
('linearSVR',LinearSVR(epsilon=epsilon))
])
svr = StandardLinearSVR()
svr.fit(X_train,y_train)
svr.score(X_test,y_test) #0.6989278257702748
![]()
文章个人博客地址
参考链接