基于细胞自动机Cellular Automata(CA)的区域生长
本来没想研究这个,但Nvidia NPP的《NVIDIA 2D Image And Signal Performance Primitives》这个模块的NPP Image Processing部分的Filtering Functions中的computer vision部分有连通域标记以及分水岭分割现成的函数。但是当我google这个函数的内容时,却发现根本没普通人使用,只弹出nvidia官网对这个函数的介绍:
 如果有很多人使用过NPP的这个函数,怎么可能就2条结果。我内心有点忐忑是不是这部分内容并不好用。
如果有很多人使用过NPP的这个函数,怎么可能就2条结果。我内心有点忐忑是不是这部分内容并不好用。
其中官网介绍这个分水岭分割是基于论文《Effiffifficient 2D and 3D Watershed on Graphics Processing Unit: Block-Asynchronous Approches Based on Cellular automata》然后我就查关于CA的内容,然后看到了有CA的区域生长,对于粘连目标,选每个目标的中心为种子点,分别区域生长是不是就把这个粘连目标分割开了。想这样做。
代码:https://github.com/KiriteeGak/region-growing-by-cellular-automata 其参考的论文是Vezhnevets, Vladimir, and Vadim Konouchine. "GrowCut: Interactive multi-label ND image segmentation by cellular automata." proc. of Graphicon. Vol. 1. 2005. 我的理解如下:
class RegionGrowing(object):
def region_growing(self, image_array, file_path, cutoff_threshold, iterations):
size = np.shape(image_array)
#图像矩阵的数组
image_array_map = {(r, c): pixel for r, each_row in enumerate(image_array) for c, pixel in
enumerate(each_row)}
#种子点状态初始化为1,其他设置为0??
seeds_map = {(r, c): 1 for (r, c) in self._seed_points(file_path)}
#默认要经过50次迭代
for it in range(0, iterations):
#先复制上一次的种子点的状态
_update_seeds_map = seeds_map.copy()
for pixel_coord, strength in seeds_map.iteritems():
#更新每个点的邻域的值
_temp_weights_neighbors_pixel = self._neighborhood_weighting(pixel_coord, image_array_map,
seeds_map, cutoff_threshold, size)
#更新种子点的坐标和值
_update_seeds_map = self._update_weights(_temp_weights_neighbors_pixel, _update_seeds_map)
#将更新后的种子点作为下一次迭代开始的种子点
seeds_map = _update_seeds_map
self._save_image(self._make_binary_image(np.shape(image_array), seeds_map))
@staticmethod
def _seed_points(file_path):
fid = open(file_path, 'rb')
return [list(map(lambda x: int(x.replace(' ', '')), line.strip().split(','))) for line in fid]
@staticmethod
#更新每个点的邻域的值后,更新种子点的统计(包括种子点坐标和值)
def _update_weights(_temp_weights_neighbors_pixel, _update_seeds_map):
for pixel_address, strength in _temp_weights_neighbors_pixel.iteritems():
#如果原来的种子点统计内没有这个点,那要把这个点新加进去。
if pixel_address not in _update_seeds_map:
_update_seeds_map[pixel_address] = strength
#如果原来的种子点内有这个点,已经是种子点,但原来这个种子点的值小于现在的值,那么这个将种子点统计内这个点的值进行更新
elif pixel_address in _update_seeds_map and _update_seeds_map[pixel_address] < strength:
_update_seeds_map[pixel_address] = strength
else:
pass
return _update_seeds_map
#更新每个像素点的邻域点的值
def _neighborhood_weighting(self, coord, image_map, seeds_map, threshold, canvas_size):
#所有像素点还是种子点???应该是所有像素点的具体坐标[r,c],图像尺寸大小[max_r,max_c]
[[r, c], [max_r, max_c]] = [coord, canvas_size]
_temp_weights = {}
#计算所有像素点3X3邻域
for i in range(-1, 2, 1):
for j in range(-1, 2, 1):
pixel_key = (r, c)
#中心点不用计算,判断越界
if [i, j] != [0, 0] and 0 <= r + i < max_r and 0 <= c + j < max_c:
#如果这个中心点是种子点
if pixel_key in seeds_map:
#计算这个中心点与邻域的strength
trans_strength = self._calculate_strength(image_map[pixel_key],
image_map[(r + i, c + j)],
seeds_map[pixel_key], threshold)
else:
##计算这个中心点与邻域的strength
trans_strength = self._calculate_strength(image_map[pixel_key],
image_map[(r + i, c + j)], 0,
threshold)
if trans_strength != 0:
#对这个点的邻域进行更新
_temp_weights[(r + i, c + j)] = trans_strength
return _temp_weights
@staticmethod
#中心点,待计算的邻域点,种子点,阈值默认是0.5
def _calculate_strength(dat1, dat2, strength, threshold):
if dat1 != 0 or dat2 != 0:
strength_trans = strength * (1 - (abs(dat1 - dat2) / max(dat1, dat2)))
if strength_trans >= threshold:
return strength_trans
return 0然后准备按照这个理解实现C++版本,我的注释部分即每个像素点都要计算3X3邻域然后更新邻域的值,那岂不是一个邻域会被相邻的几个中心点更新很多次?这样不是很浪费时间吗??另外一点,我按照这个注释运行完C++结果,这是以[300,300]为种子点迭代50、100、200、300、400、450、480次的结果:
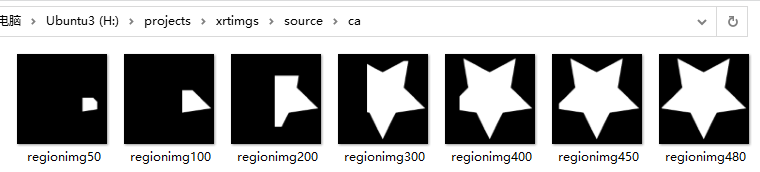 可以看到需要迭代很多次轮廓才是完全的。是不是因此网上给出的python代码才选了不止一个种子点,而是5个然后迭代几十次就够了!!!因为单一种子点耗时太长是吗。
可以看到需要迭代很多次轮廓才是完全的。是不是因此网上给出的python代码才选了不止一个种子点,而是5个然后迭代几十次就够了!!!因为单一种子点耗时太长是吗。
论文中提到对于多个目标的分割也是可以应用的,但我看论文中多个目标时选的种子点更多,我觉得这个算法应用有点狭窄,最起码现在我有点不想使用了。
但是我还是要试试对于粘连轮廓分割怎样,是否可以达到分水岭的效果
这是我的待分割原图,其实分水岭的效果已经分割得可以。然后以下分别是迭代10、15、20次的结果
看来还是要去理解关于CA的分水岭算法那篇论文。这篇论文实在太难了.....
我刚刚找到Nvidia NPP提供的watershedSegment的API,它是基于CA分水岭实现的https://docs.nvidia.com/cuda/npp/group__image__filter__watershed__segmentation.html,https://github.com/NVIDIA/CUDALibrarySamples/tree/master/NPP/watershedSegmentation Nvidia官方上个月才更新。每次查这些资料的时候,总会对我们自己失望,在技术方面与国际接轨还是太慢了,知识的更新落后了很多。问一堆CUDA或图像相关的群里,或查国内一些网站,都有这个感觉。
NPP中关于分水岭与连通域标记都是需要CUDA11.0支持的,而我的电脑不是,所以我真的很想看明白那篇论文自己实现,再看看吧。
另外感觉这个博客平台越来越功利了,现在只为了赚钱而存在,不想在这里记录了,想转简书或博客园去。



