“疫”来风雨声,全球知多少,且看全球疫情分析
学习使人进步,我刚入门数据分析,有什么不对的地方还请多多指教。
本文代码Fork于:https://www.kesci.com/home/project/5e7a0ae298d4a8002d2cd0fc。
数据来源:https://data.humdata.org/dataset/novel-coronavirus-2019-ncov-cases(1月22日-3月28日全球疫情数据)
目录
- 一、结果展示分析
- (一)全球疫情情况
- (二)疫情死亡率
- (三)疫情治愈率
- (四)每日被感染人数
- (五)每日新增确诊人数
- (六)每日新增死亡人数
- (七)每日新增治愈人数
- (八)每日累计确诊人数
- (九)每日累计死亡人数
- (十)每日累计治愈人数
- 二、代码展示
- 导入库
- 数据读取
- 数据清洗
- 地图的相关数据准备
- 时间轴
- 地图数据
- 特征处理
- 可视化
- 全球疫情热力地图
- 各国每日死亡率
- 各国每日治愈率
- 医院每日确诊感染人数
- 各国每日新增确诊案例
- 各国每日新增死亡案例
- 各国每日新增治愈案例
- 累计确诊案例
- 累计死亡案例
- 累计治愈案例
一、结果展示分析
(一)全球疫情情况
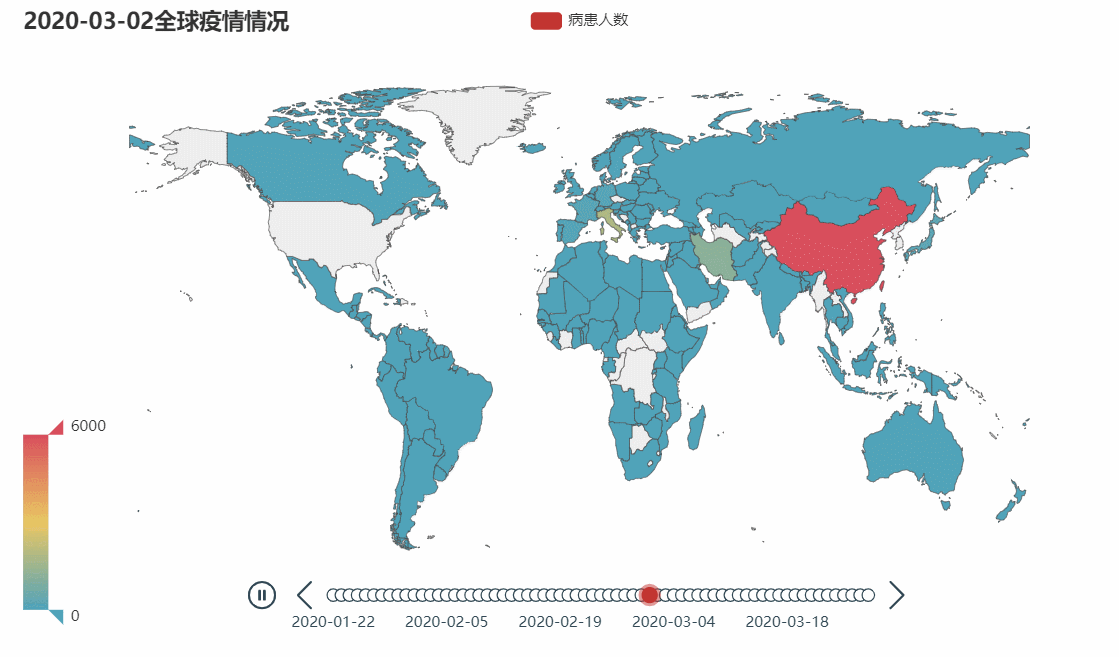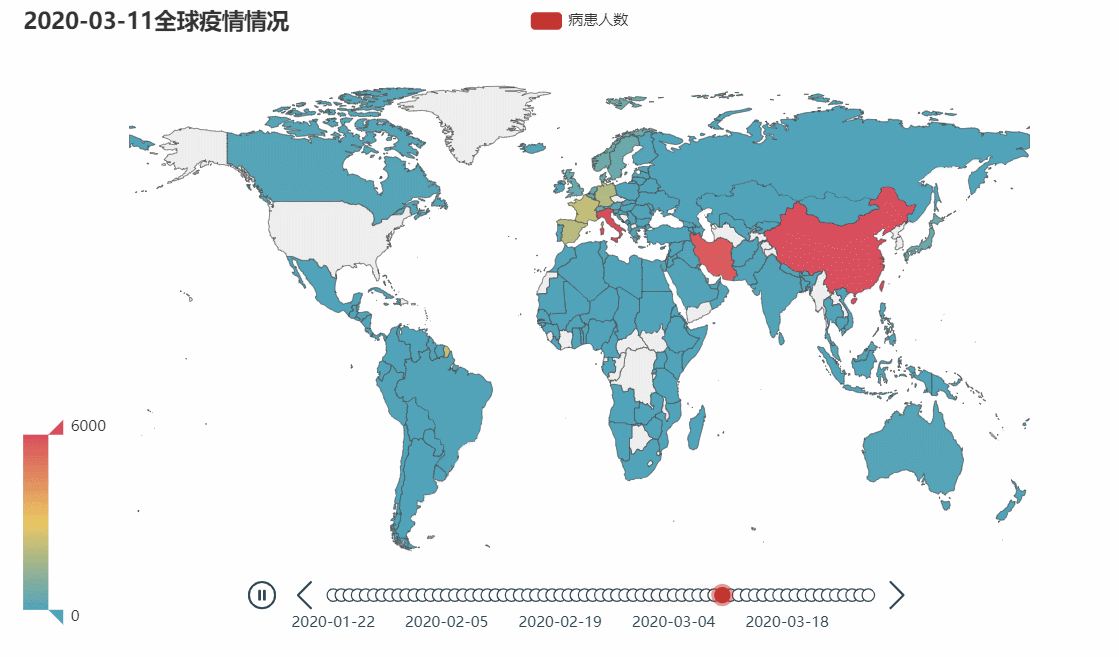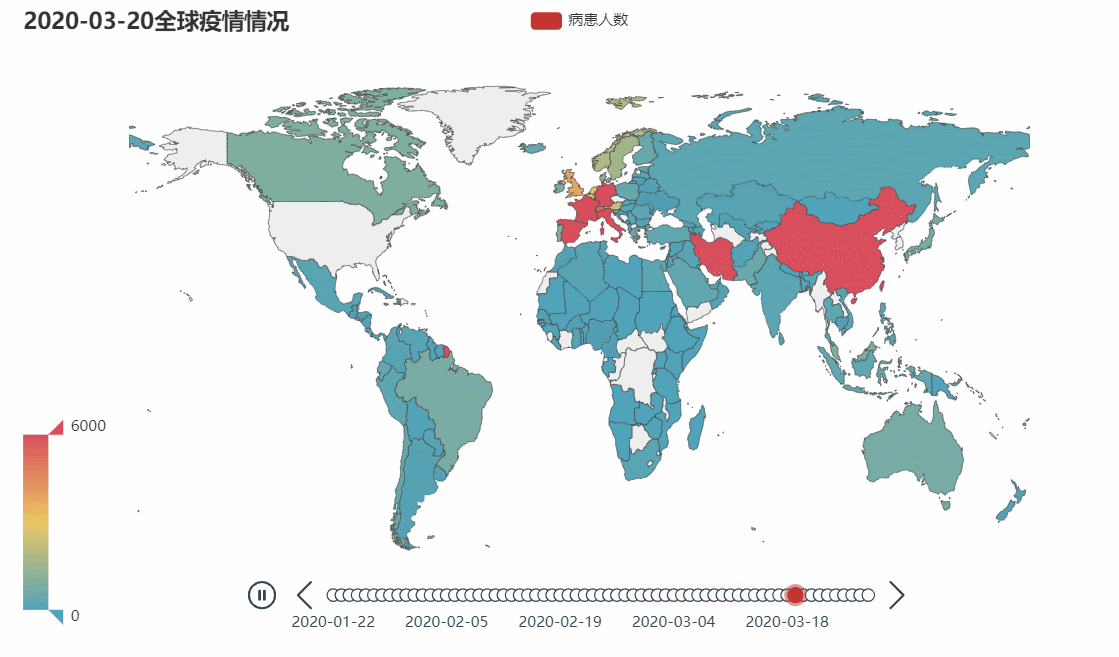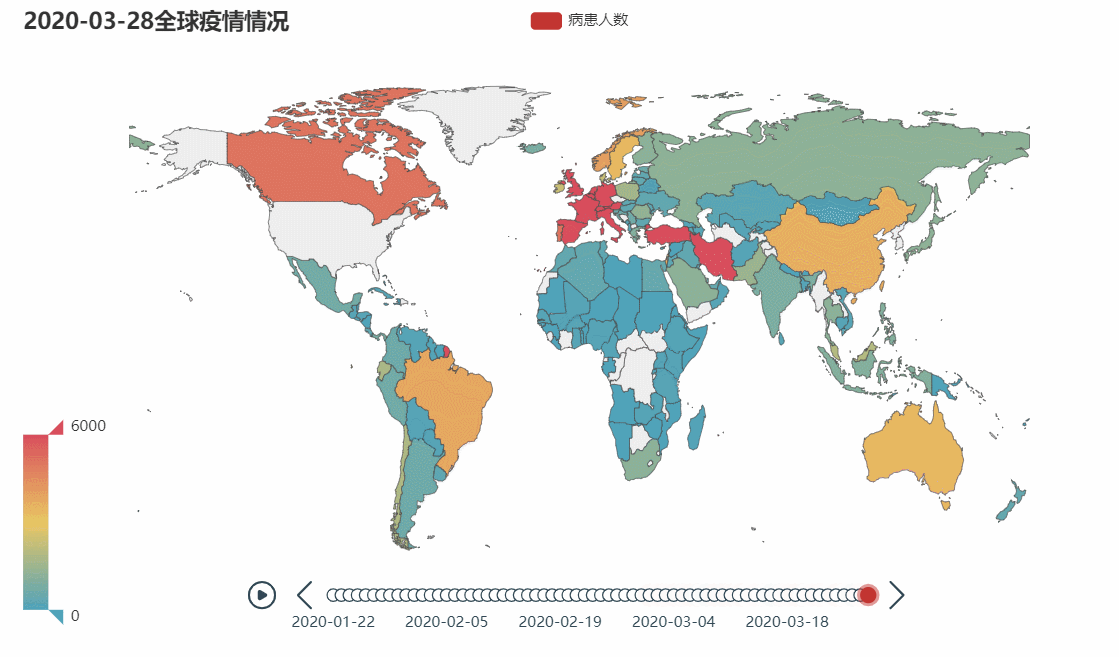
该图展示的是各个国家的感染新型冠状病毒的病患人数。三月份之前也是有不少国家已经出现了新型冠状病毒患者,但是基于我国这巨大数据,在地图上实在看不出其他国家有什么变化,所以我这里展示的是3月份以后的数据。
Iran(伊朗)和Italy(意大利)地区,是全球范围内除中国外出现严重状况的首批国家(首先出现红色),随后与Italy(意大利)接壤的几个国家相继出现严重状况,纵观全球都有不同程度的出现疫情状况,还在往更严重的方向发展的趋势,再反观中国在三月已经出现好转的趋势,并在月末左右地图标记变成了非红色(相比其他国家已经不是很严重了)。
这次的疫情是全人类共同面临的难关,我国已经快恢复正常了,疫情期间还不断的在与其他国家分享疫情的研究数据,为祖国感到骄傲,大家都要加油呀。
这里开个玩笑,是看B站来的,感叹中国文化博大精深。
中国说:我们快完了
外国说:我们快完了
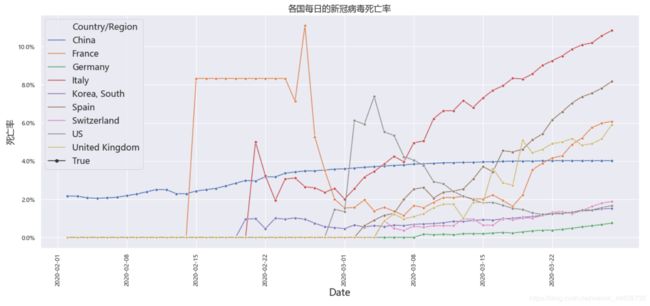
(二)疫情死亡率
【注】为了不显得整个图太过混乱,以下的图(包括该图)都只截取了各别国家的数据
[‘Italy’, ‘Korea, South’, ‘Germany’, ‘US’, ‘Spain’, ‘France’, ‘Switzerland’, ‘China’, ‘Iran’, ‘United Kingdom’]
昨天也和朋友说到国际疫情,就说到这个Italy(意大利)是目前最严重的地方,今天细看这个图,实在是有点儿吓人,这都是人命呀,这里特别想diss那些乱吃野味和疑似患病还逃跑的人。
中国疫情发生的比较久了,看中国的死亡率发现新冠病毒的致死性还是有的,不能百分百的治疗好,还有待医疗部门继续研究,另外大家出门还是要戴口罩,不要去人群密集的场所,国内还没完全安全。
这个图里没有Iran(伊朗),但我还是要提一下,不知道是数据的问题还是怎么,Iran(伊朗)的死亡率 特别高,高到把它画出来 ,其他线都挤成一根线了,是医疗条件不发达吗,有了解的朋友可以聊一聊。
(三)疫情治愈率
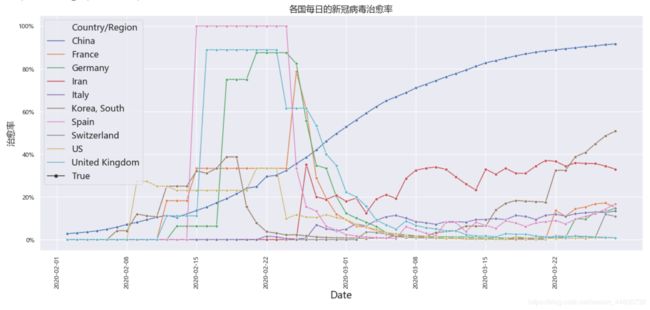
在二月下旬有不少西方国家的治愈率陡然下跌,我猜想是患病人数的急剧上升,而西方的医疗资源紧缺这两方面导致的,但在后续的三月份中,各国的治愈率整体是有上升趋势的,说明大家都有在往好的方面发展,再看中国现在的治愈率是很高的,虽说没有达到百分百治愈,但在全球各国比较下,我国是在领先位置上的,也反证了中国疫情状况良好起来了。
(四)每日被感染人数
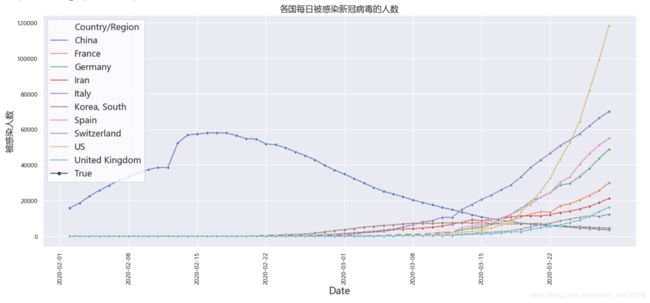
每日被感染的人数,我国在二月十五日开始有明显的减少,而其他国家在三月份开始呈现递增的走势,人传人,越来越多,特别是美国都有种类似于爆炸式增长的错觉,这些日子特朗普是被骂的比较的惨呀。
(五)每日新增确诊人数
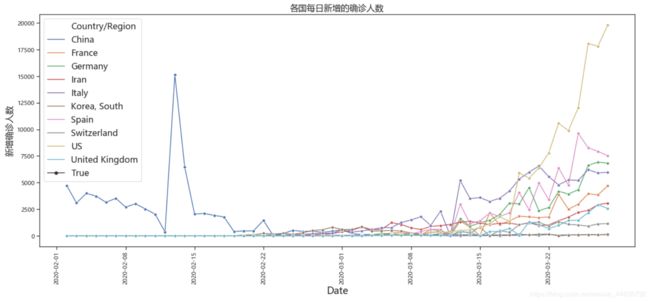
中国在二月十五日前的一小段数据有点儿突然,突然上升又突然下降,我严重怀疑是数据的问题,国内的整体状况是逐渐变好的,每日新增确诊人数都在减少,而其他国家每日新增确诊人数都在提高,还没控制下来。
(六)每日新增死亡人数
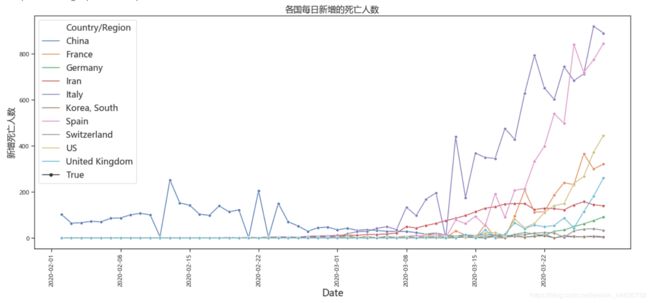
我国每日新增的致死人数是在比较小的范围里的,且在三月已经控制到更低的范围了,而其他国家的数据均处于上升趋势中,而这里Iran(伊朗)的死亡人数显示的不是很高,再联想到之前超高的死亡率,是伊朗政府还未开展行动?我不敢妄下定论。
(七)每日新增治愈人数
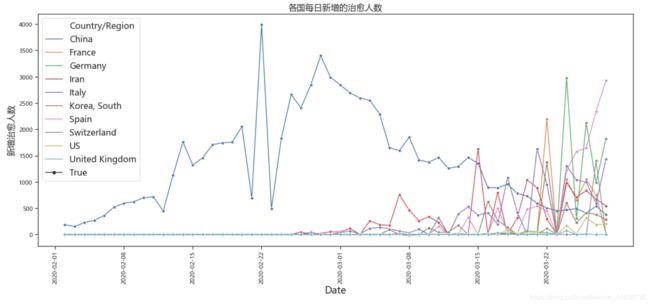
其他国家的数据量还是比较少的,又处在刚爆发疫情不久,医疗研究相比还不是很好,还有待增加数据分析。看中国的线条大体类似于一个山坡,后面减少不是说我国治愈好的人变少了,情况变坏了,这是因为我国的情况变好了,没有那么多人需要治愈了,所以相比之前就是在减少的趋势。
(八)每日累计确诊人数
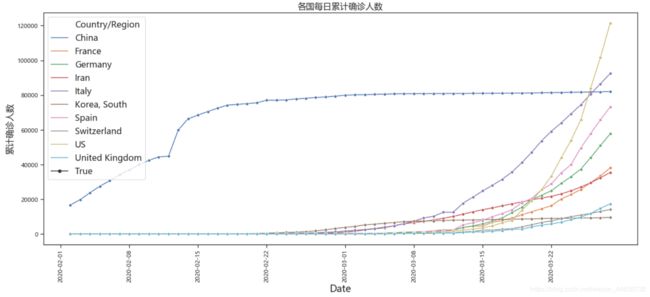
拿我国的数据和国外各国的数据相比较,我国现在是一个比较稳定的数据,而其他国家是呈上升趋势的,这一比较容易发现我国已经控制住了疫情,而其他国家正处在爆发阶段。
(九)每日累计死亡人数
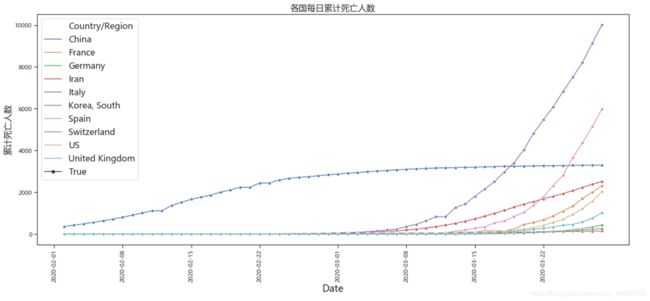
和上面所说的日累计确诊人数的分析类似,我国控制住了疫情,其他各国处于疫情爆发的阶段,这里可以看出 Italy(意大利)的死亡人数是最多的,意大利人口本就不多,又面临这样的问题,这次疫情过后,其国内可能会受到很大的影响。
(十)每日累计治愈人数
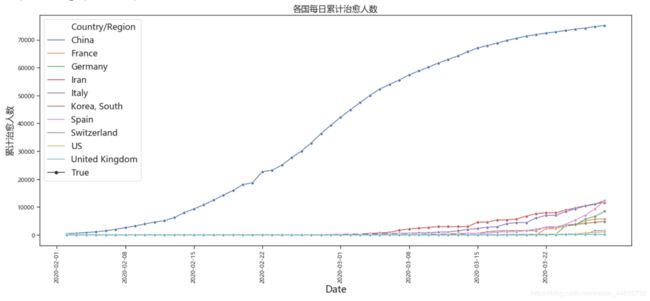
日累计治愈人数都在增加,各国都有往好的方面发展,而且我国也在积极帮助其他国家,想必这次全球问题会较快的结束。
二、代码展示
有需要源码和数据的可以联系我
数据来源: https://data.humdata.org/dataset/novel-coronavirus-2019-ncov-cases
导入库
import pandas as pd
import numpy as np
import matplotlib as mpl
import matplotlib.pyplot as plt
import seaborn as sns
import matplotlib.ticker as mtick
import datetime
import copy
from pyecharts import options as opts
from pyecharts.charts import Map
from pyecharts.charts import Timeline
from pyecharts.globals import ChartType
%matplotlib inline
数据读取
confirmed = pd.read_csv('data/time_series_covid19_confirmed_global_narrow.csv', skiprows=[1])
deaths = pd.read_csv('data/time_series_covid19_deaths_global_narrow.csv', skiprows=[1])
recovered = pd.read_csv('data/time_series_covid19_recovered_global_narrow.csv', skiprows=[1])
数据清洗
confirmed['Key'] = 'confirmed'
deaths['Key'] = 'deaths'
recovered['Key'] = 'recovered'
data = pd.concat([confirmed, deaths, recovered])
data = data[['Country/Region', 'Date', 'Value', 'Key']]
data['Date'] = pd.to_datetime(data['Date'])
data.groupby(['Country/Region', 'Date', 'Key']).sum().reset_index()
| Country/Region | Date | Key | Value | |
|---|---|---|---|---|
| 0 | Afghanistan | 2020-01-22 | confirmed | 0 |
| 1 | Afghanistan | 2020-01-22 | deaths | 0 |
| 2 | Afghanistan | 2020-01-22 | recovered | 0 |
| 3 | Afghanistan | 2020-01-23 | confirmed | 0 |
| 4 | Afghanistan | 2020-01-23 | deaths | 0 |
| ... | ... | ... | ... | ... |
| 35572 | Zimbabwe | 2020-03-27 | deaths | 1 |
| 35573 | Zimbabwe | 2020-03-27 | recovered | 0 |
| 35574 | Zimbabwe | 2020-03-28 | confirmed | 7 |
| 35575 | Zimbabwe | 2020-03-28 | deaths | 1 |
| 35576 | Zimbabwe | 2020-03-28 | recovered | 0 |
35577 rows × 4 columns
data = pd.pivot_table(data, values='Value', index=['Country/Region', 'Date'], columns=['Key'], aggfunc=np.sum).reset_index()
map_data = data.copy()
country_list = ['Italy', 'Korea, South', 'Germany', 'US', 'Spain', 'France', 'Switzerland', 'China', 'Iran', 'United Kingdom']
data = data[data['Country/Region'].isin(country_list)]
map_data['Date'] = pd.to_datetime(map_data['Date'])
map_data.set_index('Date', inplace=True)
地图的相关数据准备
时间轴
timeLine_index = list(map_data.index.unique().map(lambda x:x.strftime('%Y-%m-%d')))
地图数据
time_data = []
for i in map_data.resample('D'):
time_data.append(pd.DataFrame(i[1]))
patient_data = []
for d in time_data:
patient_data.append([tuple(z) for z in zip(d['Country/Region'].to_list(), (d['confirmed']-d['recovered']-d['deaths']).to_list())])
特征处理
def get_yesterday_data(flag_str, date, country):
# 获取昨日增长数据
DD = datetime.timedelta(days=1)
date = date - DD
date = date.strftime("%Y-%m-%d")
try:
res = data.loc[(data['Country/Region'] == country) & (data['Date']==date)][flag_str].reset_index()[flag_str][0]
except:
res = 0
return res
# 获取昨日数据,关键是借此获取每日新增数据
data['yesterday_confirmed'] = data.apply(lambda x: get_yesterday_data('confirmed', x['Date'], x['Country/Region']), axis=1)
data['yesterday_deaths'] = data.apply(lambda x: get_yesterday_data('deaths', x['Date'], x['Country/Region']), axis=1)
data['yesterday_recovered'] = data.apply(lambda x: get_yesterday_data('recovered', x['Date'], x['Country/Region']), axis=1)
# 每日新增数据
data['new_confirmed'] = data['confirmed'] - data['yesterday_confirmed']
data['new_deaths'] = data['deaths'] - data['yesterday_deaths']
data['new_recovered'] = data['recovered'] - data['yesterday_recovered']
data.head()
| Key | Country/Region | Date | confirmed | deaths | recovered | yesterday_confirmed | yesterday_deaths | yesterday_recovered | new_confirmed | new_deaths | new_recovered |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 2278 | China | 2020-01-22 | 548 | 17 | 28 | 0 | 0 | 0 | 548 | 17 | 28 |
| 2279 | China | 2020-01-23 | 643 | 18 | 30 | 548 | 17 | 28 | 95 | 1 | 2 |
| 2280 | China | 2020-01-24 | 920 | 26 | 36 | 643 | 18 | 30 | 277 | 8 | 6 |
| 2281 | China | 2020-01-25 | 1406 | 42 | 39 | 920 | 26 | 36 | 486 | 16 | 3 |
| 2282 | China | 2020-01-26 | 2075 | 56 | 49 | 1406 | 42 | 39 | 669 | 14 | 10 |
# 死亡率
data['deaths_by_confirmed'] = round(data['deaths']/data['confirmed']*100, 2)
# 治愈率
data['recovered_by_confirmed'] = round(data['recovered']/data['confirmed']*100, 2)
# 住院治疗人数
data['confirmed_minus_recovered&deaths'] = data['confirmed'] - data['recovered'] - data['deaths']
可视化
sns.set()
marker_pool = ('h')
全球疫情热力地图
tl = Timeline()
tl.add_schema(
play_interval = 800,
is_auto_play = True,
is_loop_play = False,
)
for t,d in zip(timeLine_index, patient_data):
m = (
Map()
.add(
series_name="病患人数",
data_pair=d,
maptype="world",
is_map_symbol_show=False,
is_roam=False
)
.set_series_opts(label_opts=opts.LabelOpts(is_show=False))
.set_global_opts(
title_opts=opts.TitleOpts(title='{}全球疫情情况'.format(t)),
visualmap_opts=opts.VisualMapOpts(max_=6000),
)
)
tl.add(m, t)
tl.render("全球疫情分析.html")
各国每日死亡率
df = data.loc[data['Date']>'2020-02-01']
df = df.loc[df['Country/Region'] != 'Iran']
plt.figure(figsize=(20, 8))
plt.xticks(rotation=90)
mpl.rcParams['font.family']='Microsoft YaHei'
plt.title('各国每日的新冠病毒死亡率', fontsize=16)
plot = sns.lineplot(x='Date', y='deaths_by_confirmed', hue='Country/Region', style=True, markers=marker_pool, data=df)
plt.xlabel('Date', fontsize=18)
plt.ylabel('死亡率', fontsize=16)
plot.legend(loc=2, prop={'size': 16})
plot.yaxis.set_major_formatter(mtick.PercentFormatter())
display(plot)
各国每日治愈率
df = data.loc[data['Date']>'2020-02-01']
plt.figure(figsize=(20, 8))
plt.xticks(rotation=90)
mpl.rcParams['font.family']='Microsoft YaHei'
plt.title('各国每日的新冠病毒治愈率', fontsize=16)
plot = sns.lineplot(x='Date', y='recovered_by_confirmed', hue='Country/Region', style=True, markers=marker_pool, data=df)
plt.xlabel('Date', fontsize=18)
plt.ylabel('治愈率', fontsize=16)
plot.legend(loc=2, prop={'size': 16})
plot.yaxis.set_major_formatter(mtick.PercentFormatter())
display(plot)
医院每日确诊感染人数
df = data.loc[data['Date']>'2020-02-01']
plt.figure(figsize=(20, 8))
plt.xticks(rotation=90)
sns.set_style("ticks", {"xtick.major.size": 12, "ytick.major.size": 8})
mpl.rcParams['font.family']='Microsoft YaHei'
plt.title('各国每日被感染新冠病毒的人数', fontsize=16)
plot = sns.lineplot(x='Date', y='confirmed_minus_recovered&deaths', hue='Country/Region', style=True, markers=marker_pool, dashes=False, data=df)
plt.xlabel('Date', fontsize=18)
plt.ylabel('被感染人数', fontsize=16)
plot.legend(loc=2, prop={'size': 16})
display(plot)
各国每日新增确诊案例
df = data.loc[data['Date']>'2020-02-01']
plt.figure(figsize=(20, 8))
plt.xticks(rotation=90)
sns.set_style("ticks", {"xtick.major.size": 12, "ytick.major.size": 8})
mpl.rcParams['font.family']='Microsoft YaHei'
plt.title('各国每日新增的确诊人数', fontsize=16)
plot = sns.lineplot(x='Date', y='new_confirmed', hue='Country/Region', style=True, markers=marker_pool, dashes=False, data=df)
plt.xlabel('Date', fontsize=18)
plt.ylabel('新增确诊人数', fontsize=16)
plot.legend(loc=2, prop={'size': 16})
display(plot)
各国每日新增死亡案例
df = data.loc[data['Date']>'2020-02-01']
plt.figure(figsize=(20, 8))
plt.xticks(rotation=90)
sns.set_style("ticks", {"xtick.major.size": 12, "ytick.major.size": 8})
mpl.rcParams['font.family']='Microsoft YaHei'
plt.title('各国每日新增的死亡人数', fontsize=16)
plot = sns.lineplot(x='Date', y='new_deaths', hue='Country/Region', style=True, markers=marker_pool, dashes=False, data=df)
plt.xlabel('Date', fontsize=18)
plt.ylabel('新增死亡人数', fontsize=16)
plot.legend(loc=2, prop={'size': 16})
display(plot)
各国每日新增治愈案例
df = data.loc[data['Date']>'2020-02-01']
plt.figure(figsize=(20, 8))
plt.xticks(rotation=90)
sns.set_style("ticks", {"xtick.major.size": 12, "ytick.major.size": 8})
mpl.rcParams['font.family']='Microsoft YaHei'
plt.title('各国每日新增的治愈人数', fontsize=16)
plot = sns.lineplot(x='Date', y='new_recovered', hue='Country/Region', style=True, markers=marker_pool, dashes=False, data=df)
plt.xlabel('Date', fontsize=18)
plt.ylabel('新增治愈人数', fontsize=16)
plot.legend(loc=2, prop={'size': 16})
display(plot)
累计确诊案例
df = data.loc[data['Date']>'2020-02-01']
df = df.dropna()
plt.figure(figsize=(20, 8))
plt.xticks(rotation=90)
sns.set_style("ticks", {"xtick.major.size": 12, "ytick.major.size": 8})
mpl.rcParams['font.family']='Microsoft YaHei'
plt.title('各国每日累计确诊人数', fontsize=16)
plot = sns.lineplot(x='Date', y='confirmed', hue='Country/Region', style=True, markers=marker_pool, data=df)
plt.xlabel('Date', fontsize=18)
plt.ylabel('累计确诊人数', fontsize=16)
plot.legend(loc=2, prop={'size': 16})
display(plot)
累计死亡案例
df = data.loc[data['Date']>'2020-02-01']
df = df.dropna()
plt.figure(figsize=(20, 8))
plt.xticks(rotation=90)
sns.set_style("ticks", {"xtick.major.size": 12, "ytick.major.size": 8})
mpl.rcParams['font.family']='Microsoft YaHei'
plt.title('各国每日累计死亡人数', fontsize=16)
plot = sns.lineplot(x='Date', y='deaths', hue='Country/Region', style=True, markers=marker_pool, data=df)
plt.xlabel('Date', fontsize=18)
plt.ylabel('累计死亡人数', fontsize=16)
plot.legend(loc=2, prop={'size': 16})
display(plot)
累计治愈案例
df = data.loc[data['Date']>'2020-02-01']
df = df.dropna()
plt.figure(figsize=(20, 8))
plt.xticks(rotation=90)
sns.set_style("ticks", {"xtick.major.size": 12, "ytick.major.size": 8})
mpl.rcParams['font.family']='Microsoft YaHei'
plt.title('各国每日累计治愈人数', fontsize=16)
plot = sns.lineplot(x='Date', y='recovered', hue='Country/Region', style=True, markers=marker_pool,dashes=False, data=df)
plt.xlabel('Date', fontsize=18)
plt.ylabel('累计治愈人数', fontsize=16)
plot.legend(loc=2, prop={'size': 16})
display(plot)