来源网站:http://dbtemp.blogspot.com/2011/08/at-last-starting-openmx.html
OpenMx是一个R的库,其官方网站连接为 http://openmx.ssri.psu.edu/installing-openmx?q=installing-openmx
在R中安装OpenMx非常简单,只需要输入
source('http://openmx.psyc.virginia.edu/getOpenMx.R')
1. 在R中使用OpenMx(OpenMx model specification in R )
当我们需要在R中调用OpenMx包中的函数时,需要在脚本中最前面包含以下:
require(OpenMx)
2.OpenMx模型
在OpenMx中,我们需要先构建模型然后在运行模型。模型构建时需要设定好模型的输入、所需要进行的运算以及其他一些限制条件。
下面是一个简单的例子,这个例子完成了将两个矩阵相加的运算。这里仅使用这个例子让读者了解OpenMx的使用规范,因为仅用R语言内置的函数便可以很好完成加法运算。
#---------------------------------------------------------------------------------------------------------
# Add_matrices
# DVM Bishop, 7th March 2010
# Toy program to illustrate features of OpenMx
#---------------------------------------------------------------------------------------------------------
# first of all specify the model; it does not run at this stage
# 构建模型变量myeasymodel,模型名字叫add up;
# The name 'add up' is used in output from the model
myeasymodel=mxModel("add up",
#模型中涉及到两个矩阵,A和B,都是3x3的定值矩阵,可以通过myeasymodel@matrices$A$values语句查看
mxMatrix(type="Full",nrow=3,ncol=3, values=c(3,2,1,0,2,4,3,1,2),name="A"),
mxMatrix(type="Full",nrow=3,ncol=3, values=c(0,0,1,1,2,0,3,0,0),name="B"),
#模型中使用到的运算表达式为A+B,myeasymodel@algebras语句查看
mxAlgebra(expression=A+B,name="mysum") ,
) # Final close bracket denotes end of model specification
# 运行设置好的模型,把结果保存在myrun变量中
# results of the model are saved to 'myrun'
myrun=mxRun(myeasymodel)
#输出运行结果
print(myrun@output$algebras) # shows matrix mysum
#---------------------------------------------------------------------------------------------------------
最后输出部分,只输出了部分结果(数值部分)。在控制台输入myrun可以输出全部的模型结果。大部分的输出结果都显示为NULL。这些输出都是模型相关的一些属性。
下面的一个复杂一些的例子完成了对三个变量之间相关的估计并给出置信区间。
note:这里需要使用到一个R包:bayesm,可以通过
install.packages(bayesm)进行安装,在脚本中,需要引用这个R包,即需要包括以下语句:require(bayesm)
Find3corr_OpenMx示例代码
#-----------------------------------------------------------------------------------
# Find3corr_OpenMx
# by DVM Bishop, 7th March 2010
# based on script on p 13 of OpenMx Users Guide
#---------------------------------------------------------------------------------
require(OpenMx) # loads OpenMx package 加载OpenMx
require(bayesm) #l You'll need to have installed this package, see text 加载bayesm
data(attitude) # read in one of R's prepackaged datasets 加载R自带数据包
mydata=attitude[,1:3]
#select the first 3 columns (ratings, complaints, privileges) 选取数据集前三列(评分、抱怨和特权三个变量)
#---------------------------------------------------------------------------------
# set up a model which specifies the observed data, and
# matrices for computed expected values, and optimization method
# 新建模型变量mytriCovModel,模型名为triCov
mytriCovModel <- mxModel("triCov",
mxMatrix( type="Full", nrow=1, ncol=3, free=TRUE, values=60, name="expMean" ),
# 以上语句定义矩阵名为expMean(均值期望)(1x3矩阵,对应三个变量的均值期望)
# 'values=60'为估计量设定初始值:选择一个接近真实均值的数
# 如果只设定一个初始值,则所有变量的初始值均为该值,这里即三个变量均值期望初始均为60
# 也可以为每个变量设定不同初值, e.g. c(60,55,50)
mxMatrix( type="Lower", nrow=3, ncol=3, free=TRUE, values=100, name="Chol" ),
# see text for explanation of 'Chol'
mxAlgebra( expression=Chol %*% t(Chol), name="expCov", ),
# 协方差矩阵(实对称正定矩阵性质)的期望可以写成Cholesky分解与转置相乘形式
# Chol is multiplied by its transpose to give a square matrix: see text
mxData( observed=mydata, type="raw" ),
# 'type' can be raw or cov (if covariance matrix as input)
# 当输入的是raw数据时,OpenMx将会对协方差矩阵进行计算
# if raw data input, then covariance matrix is computed by OpenMx
mxFIMLObjective( covariance="expCov", means="expMean", dimnames=colnames(mydata) )
# 选用完全信息最大似然法估计均值和协方差的期望值
# dimnames 用来指定协方差矩阵对应的变量的名字
# This command specifies we will use the FIML method to find best fit between observed
# and expected covariances and means for the variables in mydata
# dimnames is required to allow mapping of observed data to expected covariance matrix
)
#-------------------------------------------------------------------------------------------------------------------------
# end of model specification;
# Opening bracket after mxModel is matched by a close bracket
#------------------------------------------------------------------------------------------------------------------------
triCovFit <- mxRun(mytriCovModel)
# 运行模型
# perform optimisation with model as set up above
my_emean=triCovFit[['expMean']]@values
# 查看拟合得到的xepMean的值,同triCovFit$expMean@values
# see text for explanation of '@values'
print("observed means")
print(c(mean(mydata$rating),mean(mydata$complaints),mean(mydata$privileges)))
# 输出样本的三个变量的均值
print("expected means under optimal fit")
print(my_emean)
# 输出拟合得到均值期望
my_ecov=triCovFit[['expCov']]@result
print("observed covariances")
print(cov(mydata))
print("expected covariances under optimal fit")
print(my_ecov)
# 同理比较协方差的拟合结果
LL <- mxEval(objective,triCovFit);
print("-log likelihood")
print(LL)
# 估计似然值
print("observed correlations")
print(cor(mydata))
print("expected correlations under optimal fit")
ecor=matrix(nmat(my_ecov),ncol=3) #nmat function from bayesm
print(ecor)
# 比较拟合得到的均值和真实均值
# nmat是bayesm中的函数,可以将协方差矩阵变换成相关矩阵
#-------------------------------------------------------------------------------------------------------------------------
上述脚本计算似然度(基于观测变量的协方差和协方差的期望值之间的相似程度)。并且在模型中设置了协方差的初始值。然后调整这些矩阵的值使得似然值更大(或者使得-2LL值更小),从而使得这些值收敛于最优拟合值。出了FIML方法外,还有许多逼得方法也可以进行这个寻优的过程。
在模型设计中,要注意,任何命名中都不能包含"+-!~?:*/^%<>=&|$"字符。例子中命名为mytriCovModel。OpenMx中的模型包含很多子属性,很类似编程中常见的数据结构——结构体。
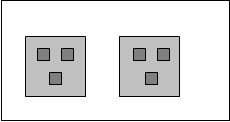
模型的形式是灵活多变的,甚至可以在模型中再定义模型。另外一种形象的理解方法可以将OpenMx的模型看做俄罗斯套娃。每个模型中可以含有其他模型。如下图中,最外层的白色框表示主模型,其中包括两个浅灰色的子模型,每个子模型中有包含三个深灰色的子模型。每个模型并不必须为完整的模型,也可以是一些部分:matrices, algebra statements, data specifications and optimization statements
经过mxRun运行过的模型 会给每个自由参数赋予最终的值。
需要注意的一些地方包括:
- 观测数的矩阵据通过mxData来指定。
- 为了计算协方差所需的矩阵(expMean和Chol)需要通过mxMatrix来制定。优化过程首先对用户给定的初始值的-LL进行估计。mxMatrix定义的矩阵中,Free=TRUE的矩阵的值会在优化过程中不断被修订。而Free=FALSE的矩阵则会固定为用户设置的值。
- 代数表达式指定对矩阵的运算操作,通过mxAlgebra指定。
- 优化方法在例子中选定为FIML,用mxFIMLObjective指定。
Cholesky decomposition
在上例子脚本中的mxAlgebra部分指定了优化中常用的Cholesky分解方法。当我们设定协方差方阵的值可以是不同的时候,最后得到的结果是不好的,即得到的结果不是一个正定矩阵(然而协方差矩阵天然应当是正定的)。因此,我们不直接定义协方差矩阵本身,而是通过定义一个下三角矩阵Chol来构建一个实对称正定协方差矩阵。这就是Cholesky分解。这个过程可以对我们要估计的协方差矩阵的形式进行限制。
- 此外 还有一点应当注意的是,在
mxMatrix( type="Lower", nrow=3, ncol=3, free=TRUE, values=100, name="Chol" ),语句中,我们设定了type为Lower,其意思是该矩阵为下三角阵,且下三角中每个值都是100.
比如,我们可以建立一个新模型,模型中存在两个有限制条件的矩阵。
mytoy=mxModel("mytoy",
mxMatrix(type="Full",nrow=1,ncol=3,free=TRUE,values=0,name="mymat1"),
mxMatrix(type="Lower",nrow=3,ncol=3,free=TRUE,values=.5,name="mymat2")
)
在控制台输入mytoy可以看到模型所有的属性。
输入mytoy@matrices可以看到两个矩阵的细节信息。
输入mytoy@matrices[2]可以看到第二个矩阵的信息,或者可以通过矩阵名字进行选定:mytoy[['mymat2']]并且可以使用mytoy[['mymat2']]@values查看初始值。