PCA与KPCA
PCA是利用特征的协方差矩阵判断变量间的方差一致性,寻找出变量之间的最佳的线性组合,来代替特征,从而达到降维的目的,但从其定义和计算方式中就可以看出,这是一种线性降维的方法,如果特征之间的关系是非线性的,用线性关系去刻画他们就会显得低效,KPCA正是应此而生,KPCA利用核化的思想,将样本的空间映射到更高维度的空间,再利用这个更高的维度空间进行线性降维。
如果样本的维度是k,样本个数是n(n>k),那么首先需要将样本投射到n维空间,这个n维空间是这样计算的:首先计算n个样本间的距离矩阵D(n*n),核函数F,则F(D)就是他的高维空间投射。
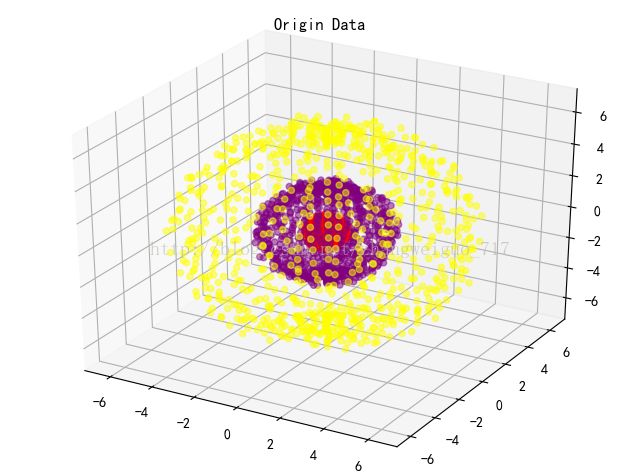
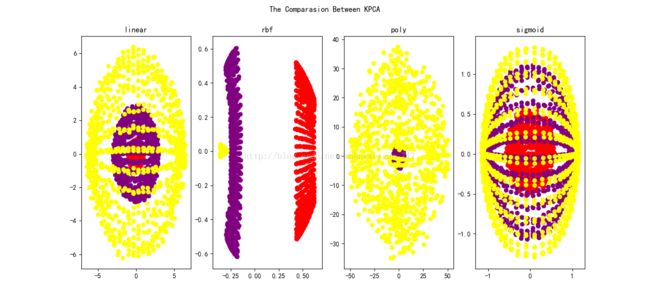
我们用几个例子来看KPCA与PCA的不同:我们用三维空间中三个同心球面来作为三类原始数据,用不同的核函数来将其降维到二维平面,当核函数是linear(线性)时,就是PCA,其他的核函数,如RBF,SIGMOID,多项式等,都是非常常用的核化函数。
原始数据分布点如下,共2700个样本,每一类样本900个:
用KPCA各类核函数将其降维后,达到效果如下:
很明显,RBF核能将不同类别的数据分开,而PCA只是将其做了一个投影,在这里,由于三个球是相互嵌套的,很难找到合适的投影方向,将三类数据很好的分开,由此造成了非常差的表现,KPCA的优点由此可见。
# -*-encoding:utf-8 -*-
from time import time
import numpy as np
import matplotlib.pyplot as plt
from matplotlib import colors
from mpl_toolkits.mplot3d import Axes3D as ax3
from sklearn import metrics
from sklearn.cluster import KMeans
from sklearn.datasets import load_digits,fetch_olivetti_faces,load_digits
from sklearn.decomposition import PCA
from sklearn.preprocessing import scale,StandardScaler
from sklearn import decomposition
from pylab import mpl
mpl.rcParams['font.sans-serif']=['SimHei']
mpl.rcParams['axes.unicode_minus']=False
color=['red','purple','yellow']
for i in colors.cnames:
if i in color:
pass
else:
color.append(i)
def generate_circle_data3():
xx=np.zeros((2700,3))
x1=np.ones((900,))+0.5*np.random.rand(900)-0.5
r1=np.linspace(0,2*np.pi,30)
r2=np.linspace(0,np.pi,30)
r1,r2=np.meshgrid(r1,r2)
r1=r1.ravel()
r2=r2.ravel()
xx[0:900,0]=x1*np.sin(r1)*np.sin(r2)
xx[0:900,1]=x1*np.cos(r1)*np.sin(r2)
xx[0:900,2]=x1*np.cos(r2)
x1=3*np.ones((900,))+0.6*np.random.rand(900)-0.6
xx[900:1800,0]=x1*np.sin(r1)*np.sin(r2)
xx[900:1800,1]=x1*np.cos(r1)*np.sin(r2)
xx[900:1800,2]=x1*np.cos(r2)
x1=6*np.ones((900,))+1.1*np.random.rand(900)-0.6
xx[1800:2700,0]=x1*np.sin(r1)*np.sin(r2)
xx[1800:2700,1]=x1*np.cos(r1)*np.sin(r2)
xx[1800:2700,2]=x1*np.cos(r2)
target=np.zeros((2700,))
target[0:900]=0
target[900:1800]=1
target[1800:2700]=2
target=target.astype('int')
return xx,target
def compare_KPCA():
data,target=generate_circle_data3()
pca=decomposition.PCA(n_components=2)
data1=pca.fit_transform(data)
try:
figure1=plt.figure(1)
ax=ax3(figure1)
ax.scatter3D(data[:,0],data[:,1],data[:,2],c=[color[i] for i in target],alpha=0.5)
plt.title('Origin Data')
except:
pass
figure2=plt.figure(2)
k=1
for kernel in ['linear','rbf','poly','sigmoid']:
plt.subplot(1,4,k)
k+=1
kpca=decomposition.KernelPCA(n_components=2,kernel=kernel)
data2=kpca.fit_transform(data)
plt.scatter(data2[:,0],data2[:,1],c=[color[i] for i in target])
plt.title(kernel)
plt.suptitle('The Comparasion Between KPCA')
plt.show()
def bench_k_means(estimator, name, data,labels):
t0 = time()
estimator.fit(data)
print('%-9s\t%.2fs\t%i\t%.3f\t%.3f\t%.3f\t%.3f\t%.3f\t%.3f'
% (name, (time() - t0), estimator.inertia_,
metrics.homogeneity_score(labels, estimator.labels_),
metrics.completeness_score(labels, estimator.labels_),
metrics.v_measure_score(labels, estimator.labels_),
metrics.adjusted_rand_score(labels, estimator.labels_),
metrics.adjusted_mutual_info_score(labels, estimator.labels_),
metrics.silhouette_score(data, estimator.labels_,
metric='euclidean')))
compare_KPCA()