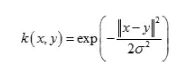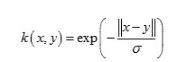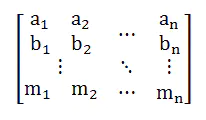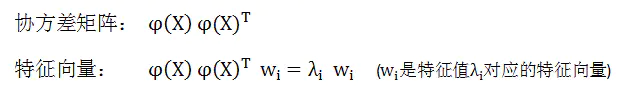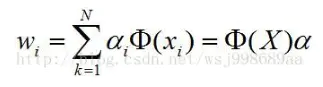KPCA降维——python
KPCA,中文名称”核主成分分析“,是对PCA算法的非线性扩展。PCA是线性的,其对于非线性数据往往显得无能为力(虽然这二者的主要目的是降维,而不是分类,但也可以用于分类),其中很大一部分原因是,KPCA能够挖掘到数据集中蕴含的非线性信息。
一、KPCA较PCA存在的创新点:
1. 为了更好地处理非线性数据,引入非线性映射函数,将原空间中的数据映射到高维空间,注意,这个是隐性的,我们不知道,也不需要知道它的具体形式是啥。
2. 引入了一个定理:空间中的任一向量(哪怕是基向量),都可以由该空间中的所有样本线性表示,这点对KPCA很重要,我想大概当时那个大牛想出KPCA的时候,这点就是它最大的灵感吧。话说这和”稀疏“的思想比较像。
二、核函数定义
核函数K(kernel function)可以直接得到低维数据映射到高维后的内积,而忽略映射函数具体是什么,即K(x, y) = <φ(x), φ(y)>,其中x和y是低维的输入向量,φ是从低维到高维的映射,
常见核函数:
三、KPCA的简单推导:
假设原始数据是如下矩阵X:(数据每一列为一个样本,每个样本有m个属性,一共有n个样本)
将每个样本通过函数φ映射到高维空间,得到高维空间的数据矩阵φ(X)。
用同样的方法计算高维空间中数据的协方差矩阵,进一步计算特征值与特征向量。
定理:空间中的任一向量(哪怕是基向量),都可以由该空间中的所有样本线性表示,这点对KPCA很重要。
所以根据这个定理,我们就可以用所有样本来表示特征向量:
将这个线性组合带回到特征向量公式,替换特征向量,得到:
进一步,等式两边同时左乘一个 φ(X)的转置:(目的是构造2个φ(X)的转置 乘 φ(X))
等式两边同时除以K,得到:
得到了与PCA相似度极高的求解公式。
总结一下KPCA算法的计算过程
- 去除平均值,进行中心化。
- 利用核函数计算核矩阵K。
- 计算核矩阵的特征值和特征向量。
- 将特征相量按对应特征值大小从上到下按行排列成矩阵,取前k行组成矩阵P。
- P即为降维后的数据。
四、代码示例:
# -*- coding: utf-8 -*-
"""kNN和降维KernelPCA """
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets,decomposition
def load_data():
'''加载用于降维的数据。return: 一个元组,依次为训练样本集和样本集的标记'''
iris=datasets.load_iris()# 使用 scikit-learn 自带的 iris 数据集
return iris.data,iris.target
def test_KPCA(*data):
'''测试 KernelPCA 的用法
:param data: 可变参数。它是一个元组,这里要求其元素依次为:训练样本集、训练样本的标记
:return: None'''
X,y=data
kernels=['linear','poly','rbf','sigmoid']
for kernel in kernels:
kpca=decomposition.KernelPCA(n_components=None,kernel=kernel) # 依次测试四种核函数
kpca.fit(X)
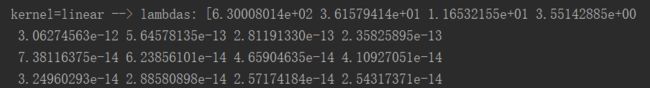
print('kernel=%s --> lambdas: %s'% (kernel,kpca.lambdas_))
if __name__=='__main__':
X,y=load_data() # 产生用于降维的数据集
test_KPCA(X,y) # 调用 test_KPCA绘制降维后样本的分布图的函数:
def plot_KPCA(*data):
''' 绘制经过 KernelPCA 降维到二维之后的样本点'''
X,y=data
kernels=['linear','poly','rbf','sigmoid']
fig=plt.figure()
colors=((1,0,0),(0,1,0),(0,0,1),(0.5,0.5,0),(0,0.5,0.5),(0.5,0,0.5),
(0.4,0.6,0),(0.6,0.4,0),(0,0.6,0.4),(0.5,0.3,0.2),)# 颜色集合,不同标记的样本染不同的颜色
for i,kernel in enumerate(kernels):
kpca=decomposition.KernelPCA(n_components=2,kernel=kernel)
kpca.fit(X)
X_r=kpca.transform(X)# 原始数据集转换到二维
ax=fig.add_subplot(2,2,i+1) ## 两行两列,每个单元显示一种核函数的 KernelPCA 的效果图
for label ,color in zip( np.unique(y),colors):
position=y==label
ax.scatter(X_r[position,0],X_r[position,1],label="target= %d"%label,
color=color)
ax.set_xlabel("X[0]")
ax.set_ylabel("X[1]")
ax.legend(loc="best")
ax.set_title("kernel=%s"%kernel)
plt.suptitle("KPCA")
plt.show()def plot_KPCA_poly(*data):
''' 绘制经过 使用 poly 核的KernelPCA 降维到二维之后的样本点 '''
X,y=data
fig=plt.figure()
colors=((1,0,0),(0,1,0),(0,0,1),(0.5,0.5,0),(0,0.5,0.5),(0.5,0,0.5),
(0.4,0.6,0),(0.6,0.4,0),(0,0.6,0.4),(0.5,0.3,0.2),)# 颜色集合,不同标记的样本染不同的颜色
Params=[(3,1,1),(3,10,1),(3,1,10),(3,10,10),(10,1,1),(10,10,1),(10,1,10),(10,10,10)] # poly 核的参数组成的列表。
# 每个元素是个元组,代表一组参数(依次为:p 值, gamma 值, r 值)
# p 取值为:3,10
# gamma 取值为 :1,10
# r 取值为:1,10
# 排列组合一共 8 种组合
for i,(p,gamma,r) in enumerate(Params):
kpca=decomposition.KernelPCA(n_components=2,kernel='poly'
,gamma=gamma,degree=p,coef0=r) # poly 核,目标为2维
kpca.fit(X)
X_r=kpca.transform(X)# 原始数据集转换到二维
ax=fig.add_subplot(2,4,i+1)## 两行四列,每个单元显示核函数为 poly 的 KernelPCA 一组参数的效果图
for label ,color in zip( np.unique(y),colors):
position=y==label
ax.scatter(X_r[position,0],X_r[position,1],label="target= %d"%label,
color=color)
ax.set_xlabel("X[0]")
ax.set_xticks([]) # 隐藏 x 轴刻度
ax.set_yticks([]) # 隐藏 y 轴刻度
ax.set_ylabel("X[1]")
ax.legend(loc="best")
ax.set_title(r"$ (%s (x \cdot z+1)+%s)^{%s}$"%(gamma,r,p))
plt.suptitle("KPCA-Poly")
plt.show()
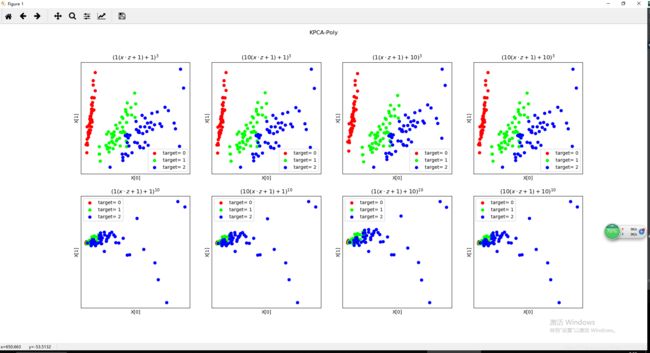 可以看到采用不同的多项式核函数,如果参数不同,降维后数据分布不同。
可以看到采用不同的多项式核函数,如果参数不同,降维后数据分布不同。
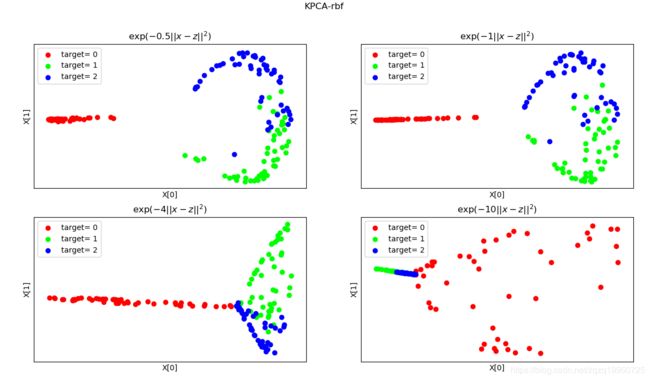
def plot_KPCA_rbf(*data):
''' 绘制经过 使用 rbf 核的KernelPCA 降维到二维之后的样本点 '''
X,y=data
fig=plt.figure()
colors=((1,0,0),(0,1,0),(0,0,1),(0.5,0.5,0),(0,0.5,0.5),(0.5,0,0.5),
(0.4,0.6,0),(0.6,0.4,0),(0,0.6,0.4),(0.5,0.3,0.2),)# 颜色集合,不同标记的样本染不同的颜色
Gammas=[0.5,1,4,10]# rbf 核的参数组成的列表。每个参数就是 gamma值
for i,gamma in enumerate(Gammas):
kpca=decomposition.KernelPCA(n_components=2,kernel='rbf',gamma=gamma)
kpca.fit(X)
X_r=kpca.transform(X)# 原始数据集转换到二维
ax=fig.add_subplot(2,2,i+1)## 两行两列,每个单元显示核函数为 rbf 的 KernelPCA 一组参数的效果图
for label ,color in zip( np.unique(y),colors):
position=y==label
ax.scatter(X_r[position,0],X_r[position,1],label="target= %d"%label,
color=color)
ax.set_xlabel("X[0]")
ax.set_xticks([]) # 隐藏 x 轴刻度
ax.set_yticks([]) # 隐藏 y 轴刻度
ax.set_ylabel("X[1]")
ax.legend(loc="best")
ax.set_title(r"$\exp(-%s||x-z||^2)$"%gamma)
plt.suptitle("KPCA-rbf")
plt.show()def plot_KPCA_sigmoid(*data):
''' 绘制经过 使用 sigmoid 核的KernelPCA 降维到二维之后的样本点 '''
X,y=data
fig=plt.figure()
colors=((1,0,0),(0,1,0),(0,0,1),(0.5,0.5,0),(0,0.5,0.5),(0.5,0,0.5),
(0.4,0.6,0),(0.6,0.4,0),(0,0.6,0.4),(0.5,0.3,0.2),)# 颜色集合,不同标记的样本染不同的颜色
Params=[(0.01,0.1),(0.01,0.2),(0.1,0.1),(0.1,0.2),(0.2,0.1),(0.2,0.2)]# sigmoid 核的参数组成的列表。
# 每个元素就是一种参数组合(依次为 gamma,coef0)
# gamma 取值为: 0.01,0.1,0.2
# coef0 取值为: 0.1,0.2
# 排列组合一共有 6 种组合
for i,(gamma,r) in enumerate(Params):
kpca=decomposition.KernelPCA(n_components=2,kernel='sigmoid',gamma=gamma,coef0=r)
kpca.fit(X)
X_r=kpca.transform(X)# 原始数据集转换到二维
ax=fig.add_subplot(3,2,i+1)## 三行两列,每个单元显示核函数为 sigmoid 的 KernelPCA 一组参数的效果图
for label ,color in zip( np.unique(y),colors):
position=y==label
ax.scatter(X_r[position,0],X_r[position,1],label="target= %d"%label,
color=color)
ax.set_xlabel("X[0]")
ax.set_xticks([]) # 隐藏 x 轴刻度
ax.set_yticks([]) # 隐藏 y 轴刻度
ax.set_ylabel("X[1]")
ax.legend(loc="best")
ax.set_title(r"$\tanh(%s(x\cdot z)+%s)$"%(gamma,r))
plt.suptitle("KPCA-sigmoid")
plt.show()