【机器学习实战】降维方法的sklearn实现----PCA和LDA
利用sklearn库来给PCA和LDA做一个比较。首先先了解一下这两个库,然后通过(iris)鸢尾花数据集来进行实践操作。
【PCA】
主要参数:
n_components
int, float, None or string
这个参数类型有int型,float型,string型,默认为None。
它的作用是指定PCA降维后的特征数(也就是降维后的维度)。
若取默认(None),则n_components==min(n_samples, n_features),即降维后特征数取样本数和原有特征数之间较小的那个;
若n_components}设置为‘mle’并且svd_solver设置为‘full’则使用MLE算法根据特征的方差分布自动去选择一定数量的主成分特征来降维;
若 0< 0 < n_components <1 < 1 并且svd_solver设置为‘full’,则n_components为主成分方差的阈值;
若n_components ≥1 ≥ 1 ,则降维后的特征数为n_components;
若svd_solver设置为‘arpack’,则n_components不能等于n_features。
copy
bool (default True)
在运行算法时,将原始训练数据复制一份。参数为bool型,默认是True,传给fit的原始训练数据X不会被覆盖;若为False,则传给fit后,原始训练数据X会被覆盖。
whiten
bool, optional (default False)
是否对降维后的数据的每个特征进行归一化。参数为bool型,默认是False。
svd_solver
string {‘auto’, ‘full’, ‘arpack’, ‘randomized’}
指定奇异值分解SVD的方法,参数为string类型,可选择‘auto’,‘full’,
‘arpack’,‘randomized’。默认是‘auto’。‘auto’适用于输入数据大于500 × × 500且要提取的成分低于最小维数数据的80%。
注:前面的PCA方法那篇中,对PCA算法的描述是用特征值分解方法, 注 : 前 面 的 P C A 方 法 那 篇 中 , 对 P C A 算 法 的 描 述 是 用 特 征 值 分 解 方 法 ,
由于特征分解是奇异值分解SVD的一个特例,实际应用时可以基于奇异 由 于 特 征 分 解 是 奇 异 值 分 解 S V D 的 一 个 特 例 , 实 际 应 用 时 可 以 基 于 奇 异
值分解来实现。 值 分 解 来 实 现 。
主要属性:
components_
array, shape (n_components, n_features)
降维后各主成分方向,并按照各主成分的方差值大小排序。
explained_variance_
array, shape (n_components,)
降维后各主成分的方差值,方差值越大,越主要。
explained_variance_ratio_
array, shape (n_components,)
降维后的各主成分的方差值占总方差值的比例,比例越大,则越主要。
singular_values_
array, shape (n_components,)
奇异值分解得到的前n_components个最大的奇异值。
主要方法:
fit(X,y=None)
用训练数据X训练模型,由于PCA是无监督降维,因此y=None。
transform(X,y=None)
对X进行降维。
fit_transform(X)
用训练数据X训练模型,并对X进行降维。相当于先用fit(X),再用transform(X)。
inverse_transform(X)
将降维后的数据转换成原始数据。
【LinearDiscriminantAnalysis】
主要参数:
solver
string, optional
求LDA超平面特征矩阵使用的方法,参数类型为string,可选‘svd’、‘lsqr’和‘eigen’,默认选‘svd’。
‘svd’:奇异值分解,不计算协方差矩阵,因此适用于具有大量特征的数据集;
‘lsqr’:最小二乘法,可与正则化一起使用;
‘eigen’:特征值分解,可与正则化一起使用,适用于特征数不多的数据集。
shrinkage
string or float, optional
正则化参数,可以增强LDA分类的泛化能力。若是只用LDA降维,可以不用。参数类型为string或float(0~1之间),默认是None。
None:不进行正则化;
‘auto’:用Ledoit-Wolf引理自动决定是否使用正则化;
float(0~1之间):给定正则化参数
正则化只在solver选择‘lsqr’,‘eigen’时有用。
priors
array, optional, shape (n_classes,)
类别权重,用于分类问题时。若是只用LDA降维,可以不用。参数类型为array,大小为类别个数n_classes。
n_components
int, optional
它的作用是指定LDA降维后的特征数(也就是降维后的维度)。参数类型为int,默认为None,1 ≤ ≤ n_components < < n_classes-1。若是不做降维,则不设置,用默认即可。
主要属性:
coef_
array, shape (n_features,) or (n_classes, n_features)
特征系数
intercept_
array, shape (n_features,)
偏置
covariance_
array-like, shape (n_features, n_features)
协方差矩阵
explained_variance_ratio_
array, shape (n_components,)
降维后的各主成分的方差值占总方差值的比例,比例越大,则越主要。只在solver选择‘svd’和‘eigen’的时候有用。
主要方法:
与PCA的类似,不过多了predict(X)、decision_function(X)等用于分类问题的方法。
【实践】
导入库:
##用于3D可视化
from mpl_toolkits.mplot3d import Axes3D
##用于可视化图表
import matplotlib.pyplot as plt
##用于做科学计算
import numpy as np
##用于做数据分析
import pandas as pd
##用于加载数据或生成数据等
from sklearn import datasets
##导入PCA库
from sklearn.decomposition import PCA
##导入LDA库
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis导入数据集:
这次实践就使用iris(鸢尾花)数据集。
iris = datasets.load_iris()
iris_X = iris.data ##获得数据集中的输入
iris_y = iris.target ##获得数据集中的输出,即标签(也就是类别)
print(iris_X.shape)
print(iris.feature_names)
print(iris.target_names)输出为:
(150, 4)
['sepal length (cm)', 'sepal width (cm)', 'petal length (cm)', 'petal width (cm)']
['setosa' 'versicolor' 'virginica']可以看出,iris数据集共有150个样本,每个样本有四个特征(四维),分别是萼片长度(sepal length),萼片宽度(sepal width),花瓣长度(petal length),花瓣宽度(petal width)。
标签有三种,分别是setosa,versicolor和virginica。
降维:
【PCA】
##加载PCA模型并训练、降维
model_pca = PCA(n_components=3)
X_pca = model_pca.fit(iris_X).transform(iris_X)
print(iris_X.shape)
print(iris_X[0:5])
print(X_pca.shape)
print(X_pca[0:5])输出为:
(150, 4)
[[ 5.1 3.5 1.4 0.2]
[ 4.9 3. 1.4 0.2]
[ 4.7 3.2 1.3 0.2]
[ 4.6 3.1 1.5 0.2]
[ 5. 3.6 1.4 0.2]]
(150, 3)
[[-2.68420713 0.32660731 -0.02151184]
[-2.71539062 -0.16955685 -0.20352143]
[-2.88981954 -0.13734561 0.02470924]
[-2.7464372 -0.31112432 0.03767198]
[-2.72859298 0.33392456 0.0962297 ]]可以发现原本为四维的样本变为了三维的。让我们分别看看四维和三维时的方差分布。
四维时:
model_pca = PCA(n_components=4)
X_pca = model_pca.fit(iris_X).transform(iris_X)
print("各主成分方向:\n",model_pca.components_)
print("各主成分的方差值:",model_pca.explained_variance_)
print("各主成分的方差值与总方差之比:",model_pca.explained_variance_ratio_)
print("奇异值分解后得到的特征值:",model_pca.singular_values_)
print("主成分数:",model_pca.n_components_)输出为:
各主成分方向:
[[ 0.36158968 -0.08226889 0.85657211 0.35884393]
[ 0.65653988 0.72971237 -0.1757674 -0.07470647]
[-0.58099728 0.59641809 0.07252408 0.54906091]
[ 0.31725455 -0.32409435 -0.47971899 0.75112056]]
各主成分的方差值: [ 4.22484077 0.24224357 0.07852391 0.02368303]
各主成分的方差值与总方差之比: [ 0.92461621 0.05301557 0.01718514 0.00518309]
奇异值分解后得到的特征值: [ 25.08986398 6.00785254 3.42053538 1.87850234]
主成分数: 4三维时:
model_pca = PCA(n_components=3)
X_pca = model_pca.fit(iris_X).transform(iris_X)
print("降维后各主成分方向:\n",model_pca.components_)
print("降维后各主成分的方差值:",model_pca.explained_variance_)
print("降维后各主成分的方差值与总方差之比:",model_pca.explained_variance_ratio_)
print("奇异值分解后得到的特征值:",model_pca.singular_values_)
print("降维后主成分数:",model_pca.n_components_)输出为:
降维后各主成分方向:
[[ 0.36158968 -0.08226889 0.85657211 0.35884393]
[ 0.65653988 0.72971237 -0.1757674 -0.07470647]
[-0.58099728 0.59641809 0.07252408 0.54906091]]
降维后各主成分的方差值: [ 4.22484077 0.24224357 0.07852391]
降维后各主成分的方差值与总方差之比: [ 0.92461621 0.05301557 0.01718514]
奇异值分解后得到的特征值: [ 25.08986398 6.00785254 3.42053538]
降维后主成分数: 3我们可以看出从四维降到三维,也就是将四维时,主成分方差值(方差值与总方差之比)最小的那个成分给去掉了。选取的是前三个最大的特征值。
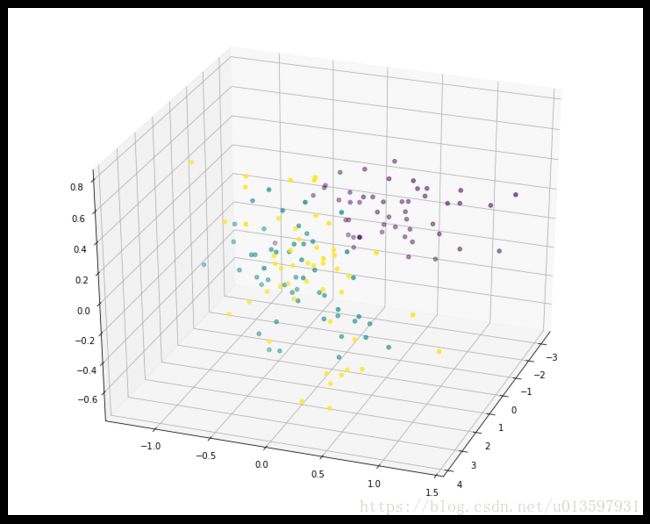
由于降到了三维,我们可以看看用图来看看三维的点的情况。
fig = plt.figure(figsize=(10,8))
ax = Axes3D(fig,rect=[0, 0, 1, 1], elev=30, azim=20)
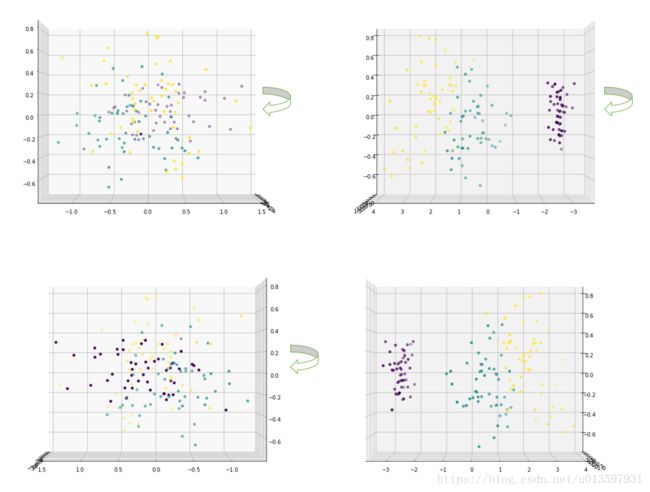
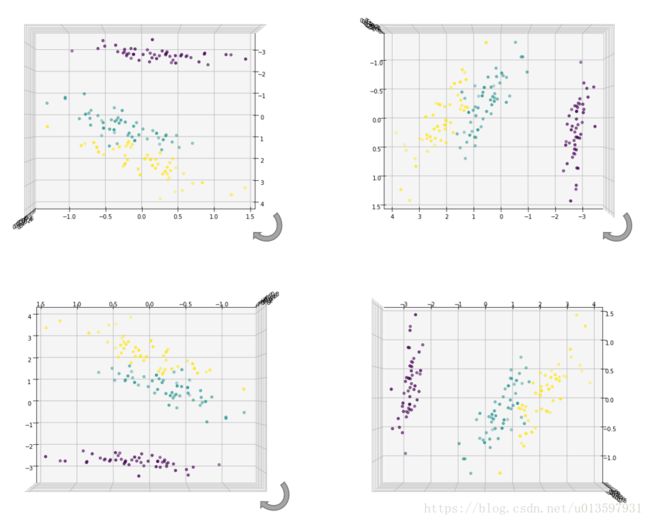
ax.scatter(X_pca[:, 0], X_pca[:, 1], X_pca[:, 2], marker='o',c=iris_y)我们也可以通过固定elev的值,改变azim的值来看看将这些点投影到各个平面的情况。
fig = plt.figure(figsize=(10,8))
##固定elev=0,改变azim为0,90,180,270
ax = Axes3D(fig,rect=[0, 0, 1, 1], elev=0, azim=0)
ax.scatter(X_pca[:, 0], X_pca[:, 1], X_pca[:, 2], marker='o',c=iris_y)
plt.show()fig = plt.figure(figsize=(10,8))
##固定elev=90,改变azim为0,90,180,270
ax = Axes3D(fig,rect=[0, 0, 1, 1], elev=90, azim=0)
ax.scatter(X_pca[:, 0], X_pca[:, 1], X_pca[:, 2], marker='o',c=iris_y)
plt.show()我们还可以在看看降维到二维的情况。
model_pca = PCA(n_components=2)
X_pca = model_pca.fit(iris_X).transform(iris_X)
print("降维后各主成分方向:\n",model_pca.components_)
print("降维后各主成分的方差值:",model_pca.explained_variance_)
print("降维后各主成分的方差值与总方差之比:",model_pca.explained_variance_ratio_)
print("奇异值分解后得到的特征值:",model_pca.singular_values_)
print("降维后主成分数:",model_pca.n_components_)输出为:
降维后各主成分方向:
[[ 0.36158968 -0.08226889 0.85657211 0.35884393]
[ 0.65653988 0.72971237 -0.1757674 -0.07470647]]
降维后各主成分的方差值: [ 4.22484077 0.24224357]
降维后各主成分的方差值与总方差之比: [ 0.92461621 0.05301557]
奇异值分解后得到的特征值: [ 25.08986398 6.00785254]
降维后主成分数: 2我们可以看出从降到二维,其实就是取了方差值(方差值与总方差之比)最大的前两个主成分。继续画图看看。
fig = plt.figure(figsize=(10,8))
plt.scatter(X_pca[:, 0], X_pca[:, 1],marker='o',c=iris_y)
plt.show()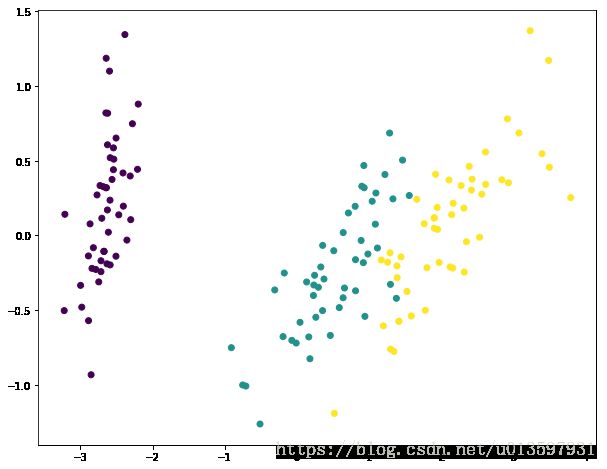
和上面有一张从上往下投影的图对应上了,也就是通过降维将那个轴对应的主成分给去掉了。
【LDA】
##载入LDA模型,设置n_components=3
model_lda = LinearDiscriminantAnalysis(n_components=3)
X_lda = model_lda.fit(iris_X, iris_y).transform(iris_X)
print("降维后各主成分的方差值与总方差之比:",model_lda.explained_variance_ratio_)
print("降维前样本数量和维度:",iris_X.shape)
print("降维后样本数量和维度:",X_lda.shape)输出为:
降维后各主成分的方差值与总方差之比: [ 0.99147248 0.00852752]
降维前样本数量和维度: (150, 4)
降维后样本数量和维度: (150, 2)我们可以发现并不是按照我设置的维度降维的,而是直接降到了2维。这是因为LDA1的n_component需满足1 ≤ ≤ n_components ≤ ≤ n_classes-1的情况,而这里n_classes为3,因此n_component无论取3还是4都是降维到2。
因此下面这段代码的结果和上面是一样的:
model_lda = LinearDiscriminantAnalysis(n_components=2)
X_lda = model_lda.fit(iris_X, iris_y).transform(iris_X)
print("降维后各主成分的方差值与总方差之比:",model_lda.explained_variance_ratio_)
print("降维前样本数量和维度:",iris_X.shape)
print("降维后样本数量和维度:",X_lda.shape)得到的各主成分方差值与总方差之比就是前两个主成分。
看看降维后的图像:

参考:
1.http://www.cnblogs.com/pinard/p/6243025.html
2.http://www.cnblogs.com/pinard/p/6249328.html
3.http://scikit-learn.org/stable/auto_examples/decomposition/plot_pca_vs_lda.html#sphx-glr-auto-examples-decomposition-plot-pca-vs-lda-py
代码存放于:https://github.com/htshinichi/ML_practice/tree/master