NEAT(基于NEAT-Python模块)实现监督学习和强化学习
参考链接:http://nn.cs.utexas.edu/downloads/papers/stanley.ec02.pdf NEAT论文
https://morvanzhou.github.io/tutorials/machine-learning/evolutionary-algorithm/4-01-neuro-evolution/ 莫凡python NEAT
http://neat-python.readthedocs.io/en/latest/neat_overview.html NEAT-Python模块官网
NEAT (NeuroEvolution of Augmenting Topologies) 是一种遗传算法,能够对神经网络的参数和形态进行进化。
NEAT(NeuroEvolution of Augmenting Topologies)是一种创建人工神经网络的进化算法。想要详细了解该算法,可以阅读Stanley’s paper在他的网站(http://www.cs.ucf.edu/~kstanley/#publications)。
如果只想了解算法的要点,只需要读几篇早期的NEAT论文是一个好的建议。大多数比较短并且很好的解释了概念。最初的NEAT论文(http://nn.cs.utexas.edu/downloads/papers/stanley.cec02.pdf)只有6页长度,而且Section 2是一个高水平的overview。
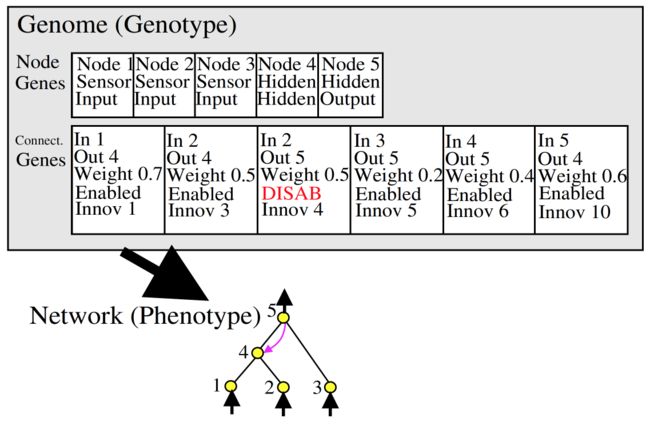
在当前的NEAT-Python实现上,个体基因组种群被保留下来。每一个基因组包括2个基因集合去描述怎样去建立一个人工神经网络。
1.结点基因,唯一地指向单个神经元。
2.连接基因,唯一地指向神经元之间的单个连接。
为了进化解决问题的方法,使用者必须提供一个fitness function计算实数值表明个体基因组的质量:拥有更好能力者有更高的分数。算法通过使用者指定的进化次数进行进化,每一代个体产生于上一代最优个体之间的繁殖以及变异。
繁殖和变异操作可能在基因组上添加结点 and/or 连接,从而该算法所得的基因组可能会变得复杂。当预先设定的迭代次数到达或者至少有一个个体的fitness(fitness criterion function=max)超过了用户指定的fitness threshold,算法终止。
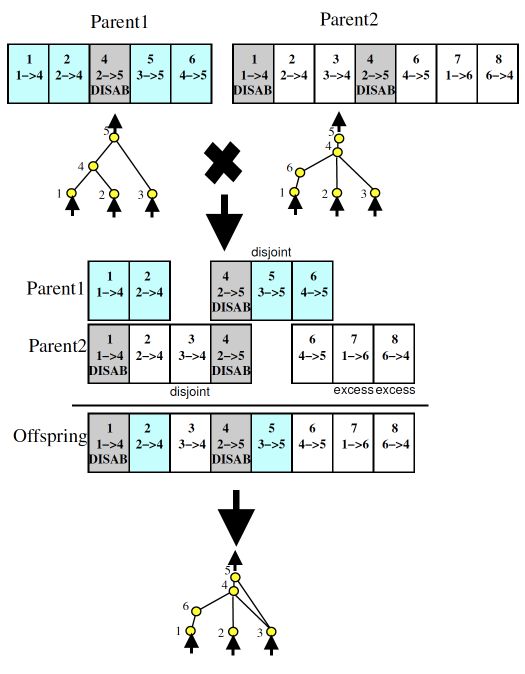
一个难点是crossover的实现-怎样实现两个不同结构网络的crossover?NEAT用identifying number(new and higher序号产生于每个附加的结点)追踪结点的起源,来源于相同祖先(homologous)的结点进行crossover,连接的结点具有相同的祖先的连接进行匹配。
另一个潜在的难点是结构变异,与连接的weights变异相比,添加node或者connection对于未来是有前景的,但是在短期有可能是破坏性的(直到被破坏性小的变异所调整)。NEAT将genomes分为多个物种来处理上述问题,genome之间相似性越高,genomic distance越近,genome相似的群体为一个物种,物种内部(而不是物种间)存在激烈的竞争。genomic distance怎样测量?非同源结点和连接的数量(非同源结点被称为disjoint或excess,取决于id的范围是在另一个父代的范围内还是范围外)
关键步骤:
(1)使用创新ID(innovation ID)对神经网络的结点和连接进行直接编码(direct coding)
(2)根据innovation ID进行交叉配对(crossover)
(3)对神经元(node)和神经链接(link)进行基因突变(mutation)
(4)尽量保留生物多样性(speciation),因为性能较差的网络可能会突变为性能好的网络
(5)初始化为只有input和output相连的神经网络,从最简单的网络开始进化。
神经网络在NEAT的表现形式:
Node genes是结点类型,输入结点、隐藏结点还是输出结点。
Connect genes是链接,存储该链接是哪个点到哪个点、权重weight、是否使用(Enable or Disable)还有创新号Innov ID。
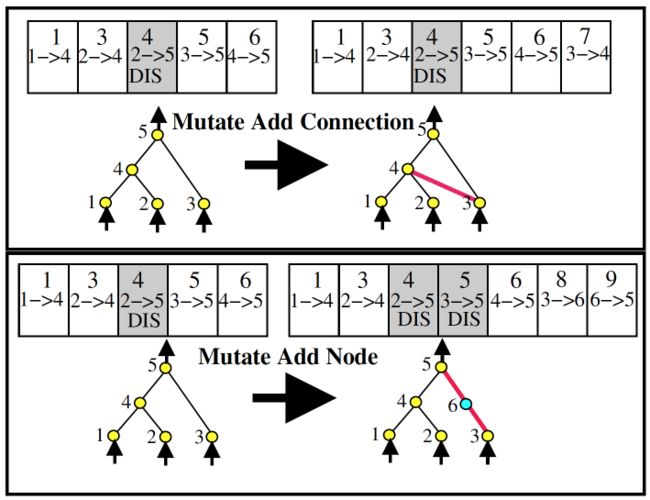
神经网络的变异
如上图所示,2→5链接为disable,因为2→5之间变异出结点4,所以现在是通过2→4→5相连。
分为结点变异和链接变异两种,如下图所示:
神经网络配对
主要根据父代和母代的基因根据Innov id进行两两对齐,双方都有的innovation就随机选择一个,如果有不匹配的基因,那么继承具有更好fitness的parent;如果Parent1和Parent2 fitness相同,则随机选择。
实例:使用NEAT进化具有xor功能的神经网络(监督学习)
进化具有如下xor函数的神经网络:
| Input 1 | Input 2 | Output |
|---|---|---|
| 0 | 0 | 0 |
| 0 | 1 | 1 |
| 1 | 0 | 1 |
| 1 | 1 | 0 |
适应度函数
适应度值通常为float类型。在本例中,基于基因组genome创建了一个feed-forward前向神经网络,对于表中的每一个例子,提供输入并且计算网络的输出。每个基因组genome的误差为![]() ,其中e(i)为期望输出(expected),a(i)为实际输出(actual)。fitness值越高越准确,若fitness=1表示准确输出。
,其中e(i)为期望输出(expected),a(i)为实际输出(actual)。fitness值越高越准确,若fitness=1表示准确输出。
neat-python模块使用eval_genomes计算适应度,函数需要两个参数:genomes列表(现有的种群)和激活的配置文件。
运行NEAT主框架
实现fitness函数之后,需要使用模板文件去实现以下步骤:
1.创建neat.config.Config对象,以配置文件为参数(配置文件参数描述)
2.使用Config对象创建neat.population.Population对象
3.调用Population对象的run方法,传入fitness function和最大迭代次数
完成上述之后,NEAT会运行直到迭代次数或者有一个genome达到fitness的阈值。
获得运行结果
当run方法return之后,可以通过Population对象的statistics成员( neat.statistics.StatisticsReporter 对象)获得在运行过程中的最优genome(s)。在本例中,通过pop.statistics.best_genome()获得最优winner genome。
通过statistics对象可以获得其他信息,比如每代的fitness平均值和标准差,最优的n个genomes等
可视化
使用visualize module可视化每代最佳和平均适应度,物种的变化以及一个genome的网络结构,使用可视化功能需要安装graphviz和python-graphviz,conda命令安装如下(在Windows上):
conda install -c conda-forge graphviz
conda install -c conda-forge python-graphviz在Ubuntu上:
sudo apt-get install graphviz配置文件解析(不能直接使用,因为添加了注释,影响了编码,要用从github上地址下载):
[NEAT]
fitness_criterion = max # genome fitness集合的最大准则
fitness_threshold = 3.9 # fitness阈值
pop_size = 150 # 种群数量
# if True,所有species由于stagnation become extinct时,重新生成一个random种群
# if False,CompleteExtinctionException异常会被抛出
reset_on_extinction = False
[DefaultGenome]
# node activation options
activation_default = sigmoid
activation_mutate_rate = 0.0 # activation函数变异概率,可能变异为activation_options中的函数
activation_options = sigmoid # activation_function列表
# node aggregation options
aggregation_default = sum # 即w0*v0+w1*v1+...+wn*vn,sum就是求和
aggregation_mutate_rate = 0.0
aggregation_options = sum
# node bias options
bias_init_mean = 0.0 # 正态分布的mean
bias_init_stdev = 1.0 # 正态分布的stdev
bias_max_value = 30.0 # bias最大值
bias_min_value = -30.0 # bias最小值
bias_mutate_power = 0.5 # 以0为中心的正态分布的标准差来得到bias的变异值
bias_mutate_rate = 0.7 # bias加上一个random值的变异概率
bias_replace_rate = 0.1 # bias用一个random值替换的变异概率
# genome compatibility options
compatibility_disjoint_coefficient = 1.0 # disjoint和excess基因数量在计算genomic distance的系数
compatibility_weight_coefficient = 0.5 # 基因组平均weight差值在计算genomic distance的系数
# connection add/remove rates
conn_add_prob = 0.5 # 在现存node之间添加connection的变异概率
conn_delete_prob = 0.5 # 删除现存connection之间的变异概率
# connection enable options
enabled_default = True # 新创建的connection的enable是True还是False
enabled_mutate_rate = 0.01 # enabled状态变为disabled概率
feed_forward = True # True表示不存在recurrent连接
initial_connection = full #
# node add/remove rates
node_add_prob = 0.2 # 添加新结点的变异概率
node_delete_prob = 0.2 # 删除新结点的变异概率
# network parameters
num_hidden = 0
num_inputs = 2
num_outputs = 1
# node response options # 同bias options
response_init_mean = 1.0
response_init_stdev = 0.0
response_max_value = 30.0
response_min_value = -30.0
response_mutate_power = 0.0
response_mutate_rate = 0.0
response_replace_rate = 0.0
# connection weight options # 同bias options
weight_init_mean = 0.0
weight_init_stdev = 1.0
weight_max_value = 30
weight_min_value = -30
weight_mutate_power = 0.5
weight_mutate_rate = 0.8
weight_replace_rate = 0.1
[DefaultSpeciesSet]
compatibility_threshold = 3.0 # genomic distance小于此距离被认为是同一物种
[DefaultStagnation]
species_fitness_func = max # 计算种群适应度的函数为种群中某个fitness最大的个体
max_stagnation = 20 # 超过此次数,该种群被视为stagnant并且移除
species_elitism = 2 # 保护该数量的fitness最大的种群不受max_stagnation的影响
[DefaultReproduction]
elitism = 2 # 每个种群的elitism个最优个体被保留到下一代
survival_threshold = 0.2 # 每一代每个species允许繁殖的概率import os
import neat
import visualize
# 2-input XOR inputs and expected outputs.
# 神经网络的输出值一般为float
xor_inputs = [(0.0, 0.0), (0.0, 1.0), (1.0, 0.0), (1.0, 1.0)]
xor_outputs = [ (0.0,), (1.0,), (1.0,), (0.0,)]
# 结果无需返回
def eval_genomes(genomes, config):
for genome_id, genome in genomes:
genome.fitness = 4.0
# 创建genome对应的net
net = neat.nn.FeedForwardNetwork.create(genome, config)
for xi, xo in zip(xor_inputs, xor_outputs):
output = net.activate(xi)
genome.fitness -= (output[0] - xo[0]) ** 2
def run(config_file):
# Load configuration.
config = neat.Config(neat.DefaultGenome, neat.DefaultReproduction,
neat.DefaultSpeciesSet, neat.DefaultStagnation,
config_file)
# 根据配置文件创建种群
p = neat.Population(config)
# Add a stdout reporter to show progress in the terminal.
p.add_reporter(neat.StdOutReporter(True))
stats = neat.StatisticsReporter()
p.add_reporter(stats)
# 每5次迭代生成一个checkpoint
p.add_reporter(neat.Checkpointer(5))
# Run for up to 300 generations.
winner = p.run(eval_genomes, 300)
# Display the winning genome.
print('\nBest genome:\n{!s}'.format(winner))
# 展示最优net的输出结果对比训练数据
print('\nOutput:')
winner_net = neat.nn.FeedForwardNetwork.create(winner, config)
for xi, xo in zip(xor_inputs, xor_outputs):
output = winner_net.activate(xi)
# 使用惊叹号!后接a 、r、 s,声明 是使用何种模式, acsii模式、引用__repr__ 或 __str__
print("input {!r}, expected output {!r}, got {!r}".format(xi, xo, output))
# 用于展示net时输入节点和输出节点的编号处理
# input node从-1,-2,-3...编号,output node从0,1,2...编号
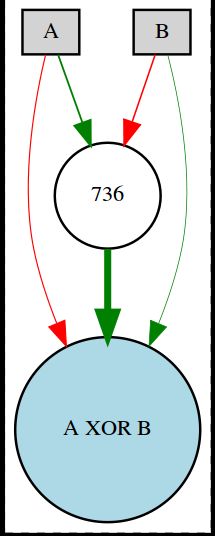
node_names = {-1: 'A', -2: 'B', 0: 'A XOR B'}
# 绘制net
visualize.draw_net(config, winner, view=True, node_names=node_names)
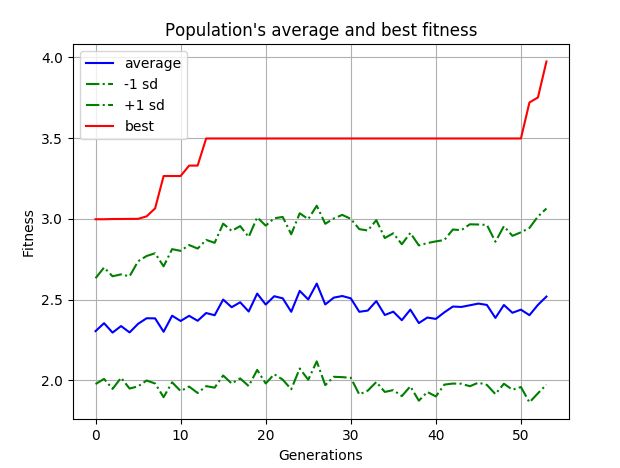
# 绘制最优和平均适应度,ylog表示y轴使用symlog(symmetric log)刻度
visualize.plot_stats(stats, ylog=False, view=True)
# 可视化种群变化
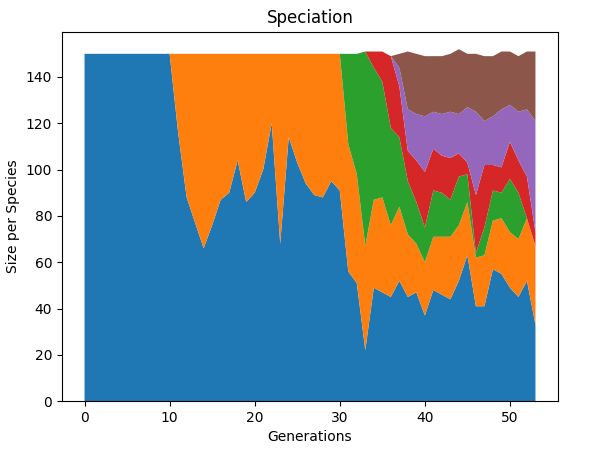
visualize.plot_species(stats, view=True)
# 使用restore_checkpoint方法使得种群p恢复到的checkpoint-4时的状态,返回population
p = neat.Checkpointer.restore_checkpoint('neat-checkpoint-4')
p.run(eval_genomes, 10)
if __name__ == '__main__':
local_dir = os.path.dirname(__file__)
config_path = os.path.join(local_dir, 'config-feedforward')
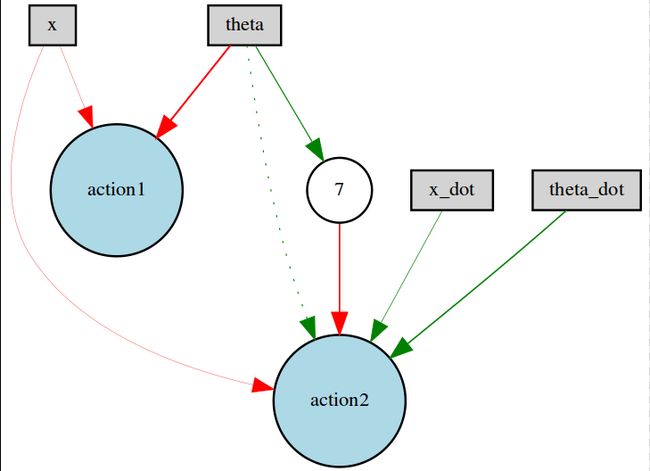
run(config_path)在net中,如果是实线,表示为Enable,若为虚线,则为Disable;红线表示权重weight<=0,绿色表示weight>0,线的粗细和大小有关。
实例:利用NEAT实现深度强化学习网络
借用CartPole实例。
程序当中,is_training= True时,为进化神经网络;当为False时,可以调出最后的checkpoint复现进化的最终状态。
import os
import neat
import visualize
import numpy as np
import gym
env = gym.make('CartPole-v0').unwrapped
max_episode = 10
winner_max_episode = 10
episode_step = 50 # 控制每轮episode的最多步数
is_training = False # True为进行进化,False为输出最优网络
checkpoint = 9 # 最终状态
# 只要杆满足条件不落下,reward=1.0
# 评估fitness就看每轮episode的总reward
# 根据木桶效应,选择最小reward的episode为fitness值
def eval_genomes(genomes, config):
for genome_id, genome in genomes:
net = neat.nn.FeedForwardNetwork.create(genome, config)
episode_reward = []
for episode in range(max_episode):
accumulative_reward = 0
observation = env.reset()
for step in range(episode_step):
action = np.argmax(net.activate(observation))
observation_, reward, done, _ = env.step(action)
accumulative_reward += reward
observation = observation_
if done:
break
episode_reward.append(accumulative_reward)
genome.fitness = np.min(episode_reward)/episode_step
def run(config_file):
config = neat.Config(neat.DefaultGenome, neat.DefaultReproduction,
neat.DefaultSpeciesSet, neat.DefaultStagnation,
config_file)
p = neat.Population(config)
p.add_reporter(neat.StdOutReporter(True))
stats = neat.StatisticsReporter()
p.add_reporter(stats)
p.add_reporter(neat.Checkpointer(5))
p.run(eval_genomes, 10)
visualize.plot_stats(stats, ylog=False, view=True)
visualize.plot_species(stats, view=True)
def evoluation():
p = neat.Checkpointer.restore_checkpoint('neat-checkpoint-%d' % checkpoint)
winner = p.run(eval_genomes, 1)
net = neat.nn.FeedForwardNetwork.create(winner, p.config)
for episode in range(winner_max_episode):
s = env.reset()
for step in range(100):
env.render()
a = np.argmax(net.activate(s))
s, r, done, _ = env.step(a)
if done:
break
node_names = {-1: 'x', -2: 'x_dot', -3: 'theta', -4: 'theta_dot',
0: 'action1', 1: 'action2'}
visualize.draw_net(p.config, winner, view=True, node_names=node_names)
if __name__ == '__main__':
local_dir = os.path.dirname(__file__)
config_path = os.path.join(local_dir, 'config-feedforward')
if is_training is True:
run(config_path)
else:
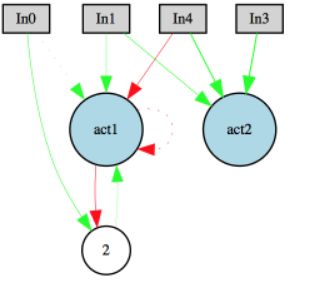
evoluation()改进为recurrent link与node
方法:将config配置文件中的的feed_forward=True改为False,所有原来的 net = neat.nn.FeedForwardNetwork 改成 neat.nn.RecurrentNetwork。可以使得神经网络结构产生循环链接和结点,使得网络具有记忆功能,神经网络的形式结构就能变化的多种多样.
比如上述带有循环链接和结点的神经网络,和RNN不同的是RNN会通过hidden state来传递记忆;而NEAT RecurrentNetwork是通过延迟刷新的形式,在某一次更新时,出了输入结点In(0,1,2,3)采用新feed的值,其他所有结点都采用上一次更新时的结点值进行计算,比如act1计算的是上次更新结点2的值而不是本次更新结点2的值,这样可以避免前向feed造成的无限循环问题。