MER: 基于ITS区域marker扩增真菌群落的准确性
Title:基于ITS区域marker扩增真菌群落的准确性
Journal: Molecular Ecology Resources
IF: 7.049
Link: https://onlinelibrary.wiley.com/doi/abs/10.1111/1755-0998.13097
Published: 09 October 2019
第一作者:厉舒祯
通讯作者:邓晔,曲媛媛
![]()
摘要
随着测序技术的发展与数据库的不断更新,基于扩增的DNA marker检测真菌多样性及组成已经成为可能。然而引物的选择和实验流程仍需要细致的验证。
本文用多种方法详细的评价了三对真菌引物(ITS1F/ITS2,gITS7/ITS4,5.8S-Fun/ITS4-Fun)。In silico结果表明引物的简并度及所在区域不同使得覆盖度存在着较大的差异。
通过扩增均匀和梯度模拟群落,发现了不同引物得到的嵌合体和错配序列比例显著不同,得到的物种组成也不同。表明了引物选择对结果存在很大的影响。选择具有校正功能和高保真的聚合酶(KAPA)显著减少了嵌合体和错配序列的比例,进而减少了OTU膨胀。
此外,使用三对引物考察了植物根内生与土壤两种环境中不同处理条件下真菌群落的结构,发现引物,而不是处理,对群落组成及多样性的影响更大。未来的研究应该对引物的效应引起重视。
![]()
背景
基于扩增子的高通量测序已经应用于真菌群落的检测。对于真菌来说,很多区域都能作为marker,如rRNA基因的小亚基 (SSU) 和大亚基(LSU) 和两个转录间隔区 (ITS1 and ITS2)。
ITS区域得到的物种多样性及分辨率更高,但是由于整个ITS太长,通常只扩增ITS1或ITS2。目前只针对一个区域的引物也有很多,如ITS1F/ITS2(ITS1区)出现时间较早,设计引物的数据库很小,但是由于其能有效避免扩增植物序列目前仍被广泛使用。近几年出现的引物也没有被系统的评价过(如gITS7/ITS4和5.8S-Fun/ITS4-Fun,ITS2区)。
ITS1和ITS2究竟哪个区域更好仍没有定论。两区域得到的物种相似或者互补的矛盾结论在之前很多研究中被报道。
引物的选择要遵循三个重要的指标:
首先是覆盖度要高;
其次是特异性要保证;
第三是扩增长度要有一定限制。
目前没有任何一对引物可以完美的满足这三个要求。因此在选择的时候需要进行权衡。
ITS1通常比ITS2短,对物种分辨率低一些,但是扩增和测序错误也低一些。ITS2分辨率高,但是各种错误会增加OTU的数量。
有研究表明引物与模板单个碱基的错配使得群落相对丰度减少了10倍。
目前模拟群落的方法已经被用于引物的评价。但是还很少有研究涉及到不同实验流程下的真菌群落。
本文通过In siliso,两类模拟群落及两类环境样本考察了三对引物扩增得到群落的特性。
方法
![]()
引物
ITS1区
ITS1F(5’-CTTGGTCATTTAGAGGAAGTAA-3’)
ITS2(5’-GCTGCGTTCTTCATCGATGC-3’)
ITS2区
gITS7(5’-GTGARTCATCGARTCTTTG-3’)
ITS4(5’-TCCTCCGCTTATTGATATGC-3’);
5.8S-Fun(5’-AACTTTYRRCAAYGGATCWCT-3’)
ITS4-Fun(5’-AGCCTCCGCTTATTGATATGCTTAART-3’)
![]()
![]()
In silico
由于引物分布在不同的区域,需要下载UNITE(用于ITS2, 5.8S-Fun, gITS7)和Silva的SSU(用于ITS1F)及LSU(用于ITS4, ITS4-FunR)数据库。引物与数据库进行BLASTn比对,计算了完美匹配和允许一个碱基错配的比对的比例。
![]()
![]()
Mock community
8种真菌构建了2类模拟群落
均匀群落(even):8种真菌16S rRNA基因数相同
梯度群落(staggered):8种真菌16S rRNA基因数存在4个数量级差异
分别用Taq和KAPA酶进行PCR扩增
![]()
![]()
环境样本
植物根内生:空白对照与添加塑化剂DBP作为处理
森林土壤:空白对照与长期模拟增温与切根实验作为处理
![]()
分析
在我们的平台进行分析(http://mem.rcees.ac.cn:8080)
![]()
结果
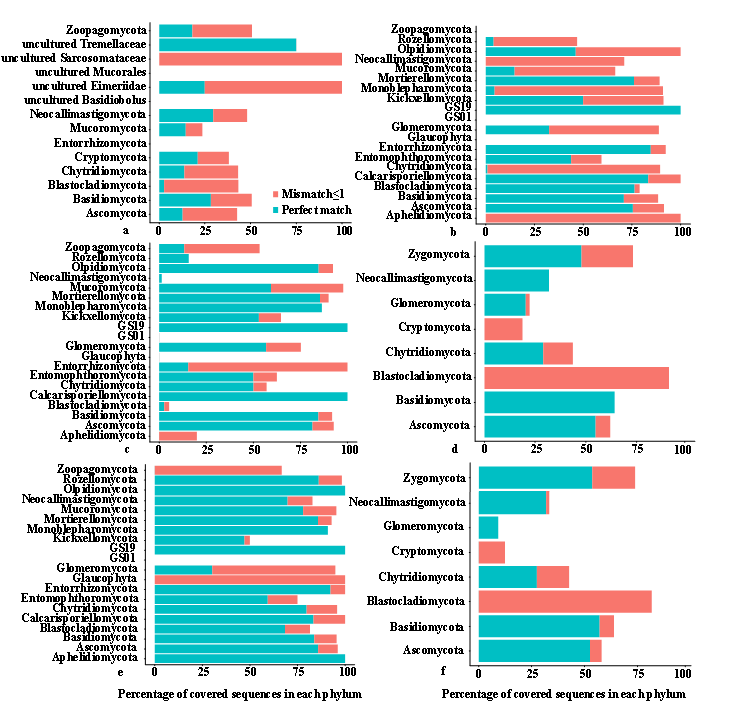
In silico结果。绿色为完美匹配的比例,红色为允许一个错配比对上的比例。不同引物在门水平上的覆盖度各有不同。由于不同数据库本身在门水平就存在差别,因此需要模拟群落及环境样本进一步验证。
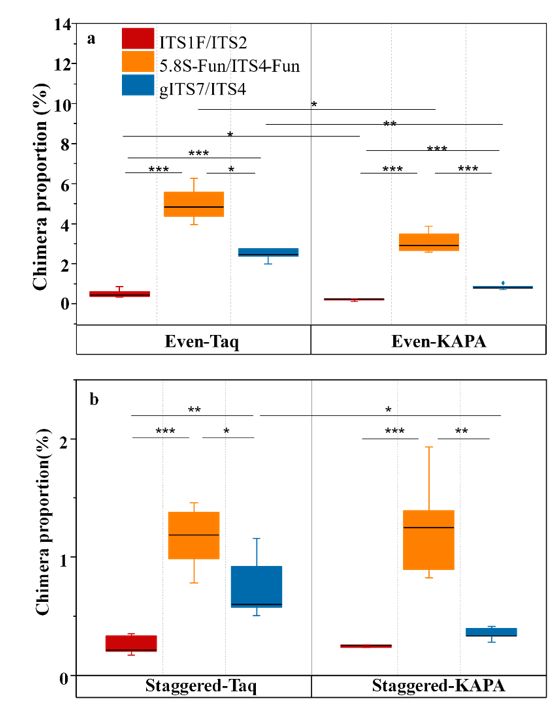
模拟群落中,引物扩增的长度越长,嵌合体(Chimeras)比例越高。ITS1F/ITS2产物长度最短,嵌合体最少。5.8S-Fun/ITS4-Fun产物最长,嵌合体比例最高。且不同引物之间存在显著的差异。KAPA酶有效的减少了嵌合体比例。
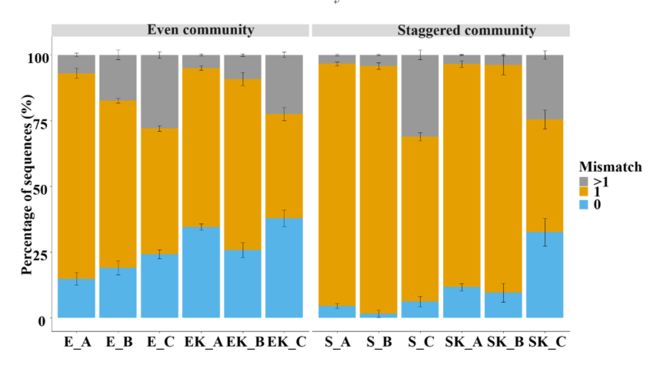
错配(mismatch)序列比例。梯度群落得到了更多的错配。KAPA酶有效减少了mismatch的比例。不同引物之间同样存在显著差异。
模拟群落得到的实际丰度与理论值的差异。虚线为理论相对丰度。
意料之外,我们发现每对引物都会丢掉8种真菌中的一种。但是之前的In silico结果却表明了丢失的真菌理论上是可以被覆盖的。这表明了仅仅利用In silico评价会有偏差。
因此将ITS1F/ITS2 或gITS7/ITS4 分别与 5.8S-fun/ITS4-Fun结合,发现组合引物的结果与理论值的相关性要高于单独一对引物。
在以上基础上,考察了两类环境样本中真菌群落在不同处理中的群落变化。由于之前已经证明了KAPA效果显著优于Taq,因此环境样本只用KAPA做。
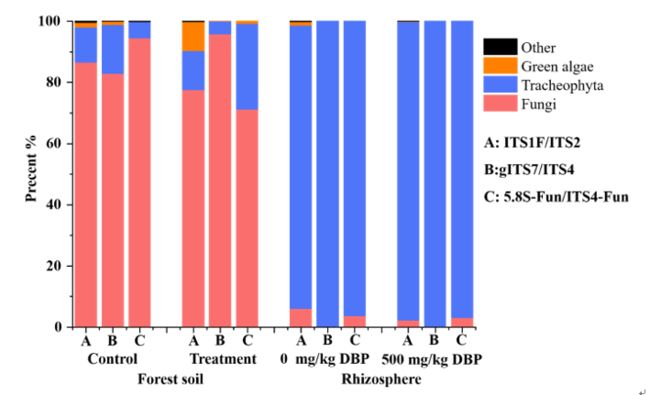
ITSx鉴定得到的真菌序列比例。对于内生真菌群落,得到的大部分仍是植物序列。对于森林土壤群落,大部分是真菌序列。
对于土壤真菌群落,不同引物得到的群落α多样性之间存在显著差异
对于土壤真菌群落,不同引物得到的群落β多样性之间存在显著差异。且处理之后群落在第一轴朝着相似的方向变化。
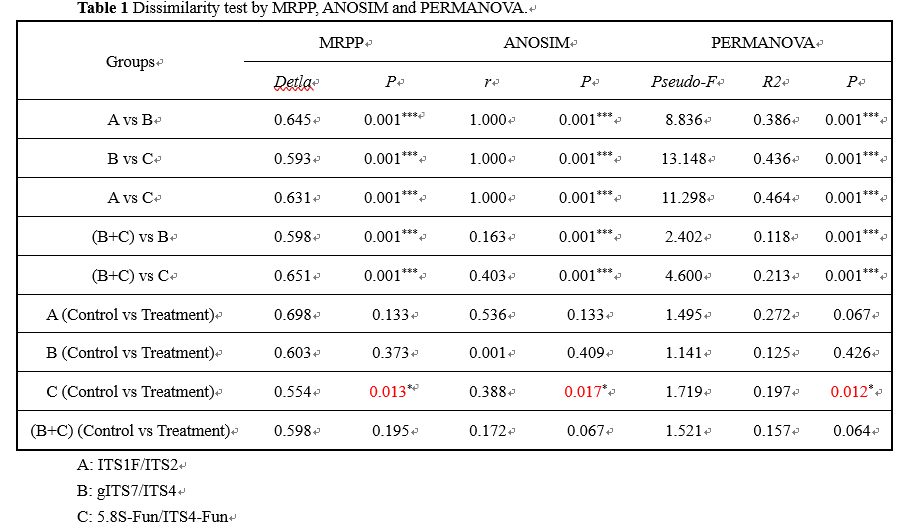
对于土壤真菌群落,Dissimilarity分析表明,引物之间存在显著的差异。此外,对照与处理之间仅5.8S-Fun/ITS4-Fun这一对引物表现出显著差异。这揭示了一个重要的结果,即我们看到的群落在处理条件下的变化很有可能是选择的引物造成的。对于另外两对引物,处理的效应就不显著。基于此我们对不同引物在门水平及属水平的物种分类进行了分析。
两类样本群落门水平的物种组成
a.不同引物共有及特有的属的个数;b.不同引物之间变化显著的属。聚类结果表明这些属按照引物聚类,而不是按照对照或处理聚类。
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
一文读懂:宏基因组 寄生虫益处 进化树
必备技能:提问 搜索 Endnote
文献阅读 热心肠 SemanticScholar Geenmedical
扩增子分析:图表解读 分析流程 统计绘图
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
在线工具:16S预测培养基 生信绘图
科研经验:云笔记 云协作 公众号
编程模板: Shell R Perl
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
![]()
学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]() 点击阅读原文,跳转最新文章目录阅读
点击阅读原文,跳转最新文章目录阅读