机器学习篇:LibSVM训练模型简单测试iris数据集
在下载好libsvm后,它自带了一个heart_scale可以用来测试,今天我用UCI的iris数据集简单测试了SVM。
iris数据集共150个样本,我采用了100个作为训练样本,50个作为测试样本,以 iris_train.scale作为训练数据和以iris_test.scale作为测试数据,同时修改好将 python的路径,并将 grid.py 文件中关于 gnuplot 路径的默认值修改为实际解压缩后的路径。 我这里将iris_train.scale、iris_test.scale放置在tools文件夹下,在cmd下进行操作:
1)运行 cmd,定位到目录 D:\libsvm-3.23\tools 如下所示:
2)运行命令:
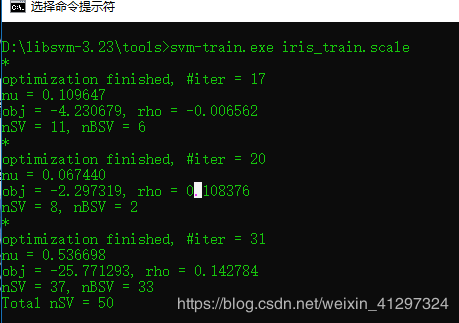
svm-train.exe iris_train.scale得到一下结果:
详细信息:
*
optimization finished, #iter = 17
nu = 0.109647
obj = -4.230679, rho = -0.006562
nSV = 11, nBSV = 6
*
optimization finished, #iter = 20
nu = 0.067440
obj = -2.297319, rho = 0.108376
nSV = 8, nBSV = 2
*
optimization finished, #iter = 31
nu = 0.536698
obj = -25.771293, rho = 0.142784
nSV = 37, nBSV = 33
Total nSV = 50
3)此时,已经得到 heart_scale.model,下面进行预测:
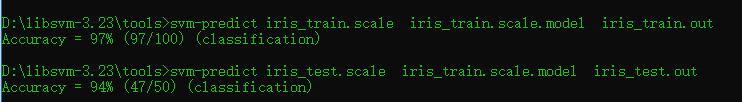
#将训练集作为预测集预测精度
svm-predict iris_train.scale iris_train.scale.model iris_train.out #将测试集作为预测集预测精度
svm-predict iris_test.scale iris_train.scale.model iris_test.out
4)进行参数最优估计,运行命令 :
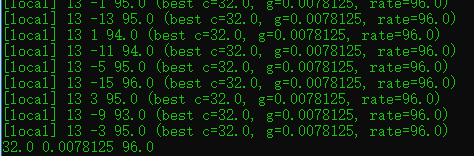
python grid.py iris_train.scaledos 窗口中会乱串的 [local] 数据,以及一个 gnuplot 的动态绘图窗口。 大约过 几秒钟停止。 Dos 窗口中的 [local] 数据时局部最优值可以不用管,我们关心最后一行。结果:
最后一行的参数就是最优参数: 最优参数 c=32,g=0.0078125
其意义表示: C = 32;γ=0.0078125(具体的说明参见SVM参数详解);
交叉验证精度 CV Rate = 96% ,这就是最优结果。
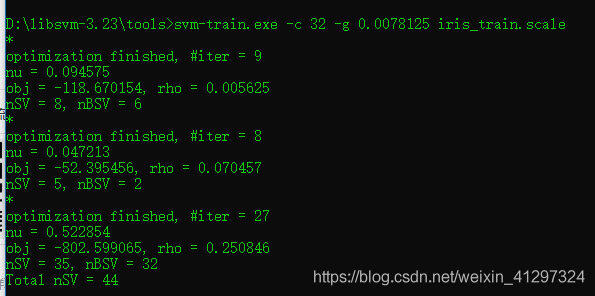
5)然后在调用命令得到 model
结果:
详细信息:
*
optimization finished, #iter = 9
nu = 0.094575
obj = -118.670154, rho = 0.005625
nSV = 8, nBSV = 6
*
optimization finished, #iter = 8
nu = 0.047213
obj = -52.395456, rho = 0.070457
nSV = 5, nBSV = 2
*
optimization finished, #iter = 27
nu = 0.522854
obj = -802.599065, rho = 0.250846
nSV = 35, nBSV = 32
Total nSV = 447)调用命令预测精度:
svm-predict.exe iris_test.scale iris_train.scale.model iris_test_cg.out结果:
得到的正确率为 Accuracy = 94%.