隐马尔科夫模型(HMM)模型训练:Baum-Welch算法
在上一篇博客中隐马尔科夫模型(HMM)原理详解,对隐马尔科夫模型的原理做了详细的介绍。今天,我们要对其中的模型训练算法Baum-Welch做一个实现,Baum-Welch算法可以在不知道状态序列的情况下,对模型的参数进行训练拟合。
这其实是非常实用的,例如在分词任务,对文本进行词性的标注成本是很高,所以我们经常面临着没有词性标注的训练文本,这个时候Baum-Welch算法就派上用场了。
首先,我们创建一个隐马尔科夫模型的类
class HMM:
"""
状态取值有N个,观测序列取值有M个
"""
def __init__(self, A: np.ndarray, B: np.ndarray, pi):
self.A = A # 状态转移概率矩阵 [N, N]
self.B = B # 输出观测概率矩阵 [N, M]
self.pi = pi # 初设状态概率向量 [N]
self.N = len(pi)
self.M = self.A.shape[1]
self.map_i = {} # 存储状态取值对应的编码, key为编码,value为状态值
self.map_o = {} # 存储观测值对应的编码, key为观测值,value为编码
前向概率计算
根据公式,我们通过递归很容易实现:
def forward(self, t, i, O):
"""
前向算法
:param t: 时刻t,以0为起点
:param i: 状态值,[0, 1, ...., N-1]
:param O: 观测序列
:return:
"""
if t == 0:
return self.pi[i] * self.B[i][O[t]]
return self.B[i][O[t]] * sum([self.forward(t - 1, j, O) * self.A[j][i] for j in range(self.N)])
我们通过书里的例子来验证一下我们的程序,
if __name__ == '__main__':
A = np.array([[0.5, 0.2, 0.3],
[0.3, 0.5, 0.2],
[0.2, 0.3, 0.5]])
B = np.array([[0.5, 0.5],
[0.4, 0.6],
[0.7, 0.3]])
pi = [0.2, 0.4, 0.4]
O = [0, 1, 0]
hmm = HMM(A, B, pi)
print(hmm.forward(t=2, i=0, O=O))
因为我们的t和i是从0开始的,所以这里其实相当于书里的t=3和i=1,程序运行的结果是0.4187。经过我的测试,其他的也是跟书里一致,小伙伴们可以自行验证。
后向概率计算
根据公式,我们通过递归很容易实现:
def backward(self, t, i, O):
"""
后向算法
:param t: 时刻t,以0为起点
:param i: 状态值,[0, 1, ...., N-1]
:param O: 观测序列
:return:
"""
if t == len(O) - 1:
return 1
return sum([self.A[i][j] * self.B[j][O[t+1]] * self.backward(t+1, j, O) for j in range(self.N)])
参数更新
def gamma(self, t, i, O):
"""
根据已知隐马尔科夫模型的参数和观测序列O,时刻t处于状态i的概率
:param t: 时刻t,以0为起点
:param i: 状态值,[0, 1, ...., N-1]
:param O: 观测序列
:return:
"""
molecule = self.forward(t, i, O) * self.backward(t, i, O)
denominator = sum([self.forward(t, j, O) * self.backward(t, j, O)] for j in range(self.N))
return molecule / denominator
def xi(self, t, i, j, O):
"""
根据已知隐马尔科夫模型的参数和观测序列O,时刻t处于状态i且时刻t+1处于状态j的概率
:param t: 时刻t,以0为起点
:param i: 状态值,[0, 1, ...., N-1]
:param O: 观测序列
:return:
"""
molecule = self.forward(t, i, O) * self.A[i][j] * self.B[j][O[t+1]] * self.backward(t+1, j, O)
denominator = 0
for n in range(self.N):
for m in range(self.N):
denominator += self.forward(t, n, O) * self.A[n][m] * self.B[m][O[t+1]] * self.backward(t+1, m, O)
return molecule / denominator
这里需要注意的是:
- 我们对一条训练样本数据即一个观测序列就会进行一次参数的更新。
- 然后,我们初始化的时候,即在训练之前,需要首先给模型一个参数的初始值。
def train(self, O):
"""
根据隐马尔科夫模型参数的估计值,极大化模型参数
:param O:
:return:
"""
pi = self.pi
A = self.A
B = self.B
for i in range(self.N):
pi[i] = self.gamma(0, i, O)
# 根据公式计算a(i,j)
for j in range(self.N):
A[i][j] = sum([self.xi(t, i, j, O) for t in range(len(O)-1)]
) / sum([self.gamma(t, i, O) for t in range(len(O)-1)])
# 根据公式计算b_i(k)
B_denominator = sum([self.gamma(t, i, O) for t in range(len(O))]) # 公式中的分母
# 求得观测序列的所有不同的取值,和对应的所有时刻。
# 比如观测序列(1, 2, 1),第一个和最后一个时刻的取值为1,第二个时刻的取值为2
Map = {}
for t in range(len(O)):
if O[t] in Map.keys():
Map[O[t]].append(t)
Map[O[t]] = [t]
# 这里k对应观测序列中的一个不同的值
for k, tl in Map.items():
B[i][k] = sum([self.gamma(t, i, O) for t in tl]) / B_denominator
# 更新模型参数
self.pi = pi
self.A = A
self.B = B
def run(self, train_nums, O):
"""
对训练样本进行训练
:param train_nums: 训练轮数
:param O: 所有训练样本,二维数组
:return:
"""
for i in range(train_nums):
for o in O:
# 将观测文本值映射为对应编码
o = [self.map_o[m] for m in o]
self.train(o) # 对一个观测序列训练,进行参数更新
维特比算法
def viterbi(self, O):
"""
维特比编码求最优路径
:param O:
:return:
"""
pro = np.ones([len(O), self.N]) # 记录累计概率
backpointers = np.ones([len(O), self.N], dtype=np.int32) # 记录最优路径
# 计算时刻t=1,各个状态下观测序列为o_1的概率
pro[0] = [self.pi[i] * self.B[i][O[0]] for i in range(self.N)]
# 时刻t=2,3,....,
for t in range(1, len(O)):
b = [self.B[i][O[t]] for i in range(self.N)] # 计算各个状态下观测序列为o_t的概率
v = np.expand_dims(pro[t - 1], 1) * self.A # 计算前一个时刻状态为j,当前时刻状态为i的概率矩阵
pro[t] = np.max(v, 0) * b # 当前状态为i的最大概率 * 对应状态下观测序列为o_t的概率
backpointers[t] = np.argmax(v, 0) # 记录,当前状态为i的概率最大时,上个时刻的状态
# 开始回溯
viterbi = [np.argmax(pro[-1])] # 最终累计概率最大时,最后一个时刻的状态
# 根据当前状态从最优路径中挑选上个时刻的状态
for bp in reversed(backpointers[1:]):
viterbi.append(bp[viterbi[-1]])
viterbi.reverse()
# 将状态编码转化为对应的状态值
viterbi = [self.map_i[v] for v in viterbi]
viterbi_score = np.max(pro[-1]) # 最高的累计概率
return viterbi, viterbi_score
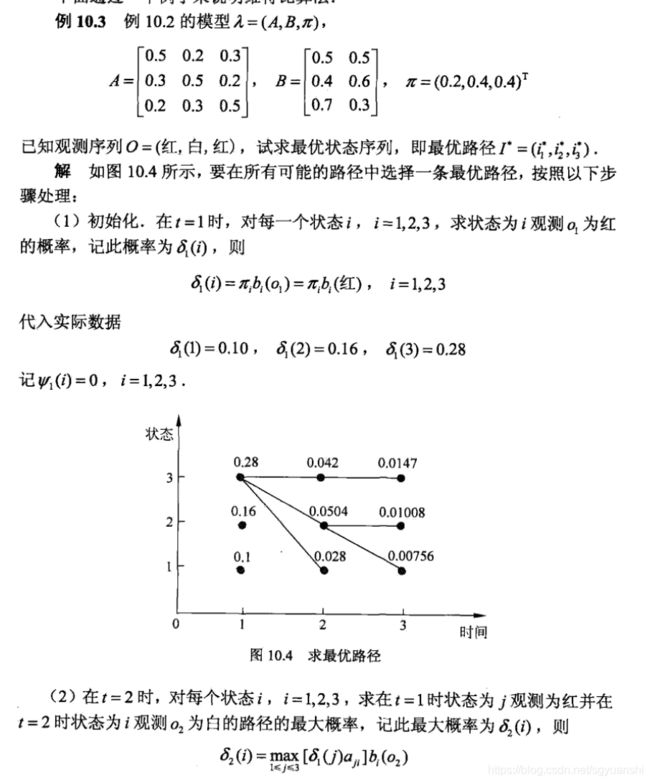
还是书里的例子,经过程序的计算,得到累计概率矩阵、最优路径、最高概率分数分别如下:
前面也说到了,我们的状态值是从0开始的,所以状态2就相当于书里的状态3。
可以看到,跟书里计算的结果完全一致。