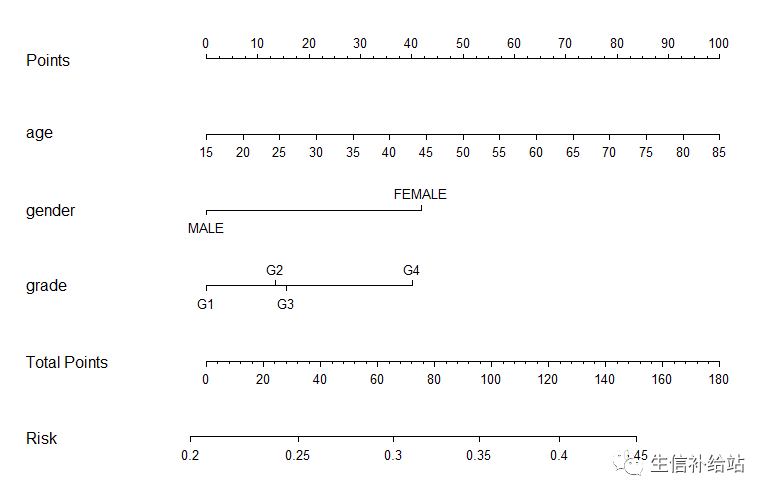Nomogram(诺莫图) | Logistic、Cox生存分析结果可视化
本文首发于“生信补给站”公众号,https://mp.weixin.qq.com/s/BWpy3F-nEKXCdVXmY3GYZg
当然还有更多R语言,生物信息学相关知识。。。?
Nomogram,也常称为诺莫图或者列线图,在医学领域的期刊出现频率越来愈多,常用于评估肿瘤学和医学的预后情况,可将Logistic回归或Cox回归的结果进行可视化呈现。
数据准备
使用TCGA-LIHC队列的临床数据,简单处理后进行示例分析:
## 载入R包
library(rms)
library(survival)
## 读取LIHC数据
LIHC <- read.csv("TCGA-LIHC-nomogram.csv",header=TRUE)
head(LIHC)
绘制nomogram图
1)打包数据
## 关键步骤:按照nomogram要求“打包”数据,可以输入??datadist查看详细说明
dd=datadist(LIHC)
options(datadist="dd")
2)构建logist模型,绘制诺莫图
## 构建logist模型,绘制诺莫图
f <- lrm(status ~ age + gender + grade , data = LIHC)
nom <- nomogram(f, fun=plogis, lp=F, funlabel="Risk")
plot(nom)
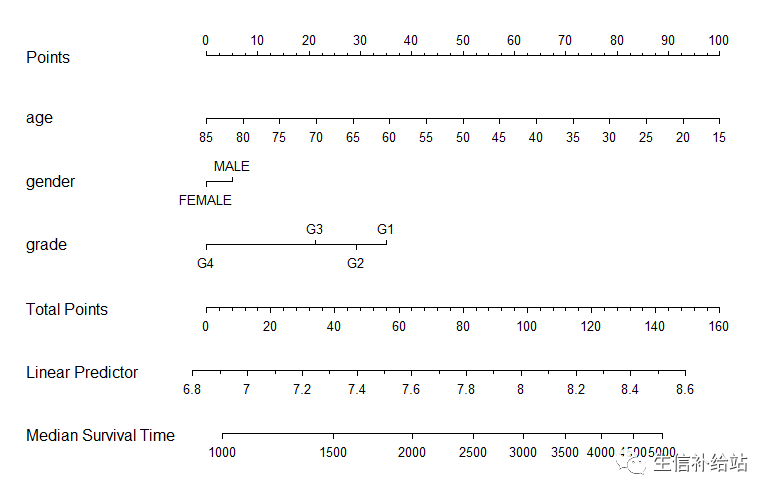
3) 构建COX模型,绘制诺莫图
A :COX回归中位生存时间的Nomogram
## 构建COX比例风险模型
f2 <- psm(Surv(time,status) ~ age+gender+grade,data = LIHC, dist='lognormal')
med <- Quantile(f2) # 计算中位生存时间
surv <- Survival(f2) # 构建生存概率函数
## 绘制COX回归中位生存时间的Nomogram图
nom <- nomogram(f2, fun=function(x) med(lp=x),funlabel="Median Survival Time")
plot(nom)
简单介绍下使用,比如某患者年龄为50岁,那就在列线图年龄为50岁的地方向上画一条垂直线,即可得到其对应的得分(Points);男性,则在男性的地方画一条垂直线,以此类推,找出每个变量状态下对应的得分,相加得到总得分。
最后将患者的总得分(Total Points)再向下画一条垂直线,就可以知道该患者对应的中位生存时间。
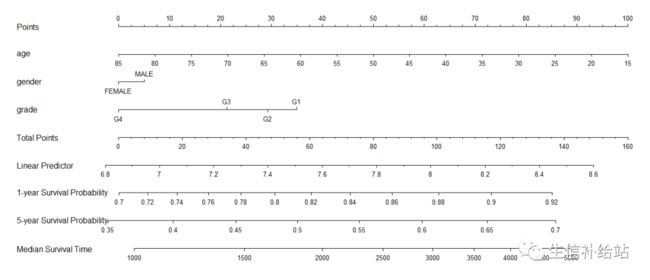
下面例子同,可以知道未来1年、5年的生存率,当然也可以是其他时间节点。
**B:绘制COX回归生存概率的Nomogram图 **
## LIHC数据的time是以”天“为单位,此处绘制1年,5年的生存概率
nom <- nomogram(f2, fun=list(function(x) surv(365, x),
function(x) surv(1825, x),
function(x) med(lp=x)),
funlabel=c("1-year Survival Probability", "5-year Survival Probability","Median Survival Time"))
plot(nom, xfrac=.2)
当然也可以计算其他时间节点的生存率,同样加到list中即可。
使用相对风险的指标(OR、HR)等统计之余,可以结合列线图展示,能够起到预测生存概率的作用,也会使预测模型的结果更直观、易懂。
【期待您的扫码关注,觉得不错点个“在看”,转发就是最好的支持和赞赏,谢谢!】
![]()